遺伝子工学実験支援ソフト SnapGene
製品概要 | クローニングシミュレーション |ユーザー紹介 | 機能詳細 | システム条件 | 製品カタログ
SnapGene(スナップジーン)は、DNAクローニングやPCRの計画、視覚化、記録を手早く簡単に行うことができる遺伝子工学実験支援ソフトウェアです。ヒューマンコンピューターインタラクション(HCI)に基づく操作性の良いインターフェイスに加え、表示言語は英語のほか日本語にも対応しているので、日本人ユーザーにとっても操作しやすいように設計されています。アノテーションやプライマーのデザインの実行もスムーズに行え、分子生物学、遺伝子工学、構造生物学分野に携わる研究者の日々のリサーチを支援します。
NEWS2024/3/19 SnapGene7.2 がリリースされました。SnapGene 7.2 では、プライマーのホモ二量体構造を可視化する機能が追加されました。また、タブをドラッグ&ドロップによって別ウィンドウとして表示することが可能になり、プロジェクト内のファイルをインタラクティブに操作できるようになっています。
追加・強化された機能はこちらから:SnapGene7.2 リリースノート
SnapGeneが選ばれる理由
遺伝子クローニングの手順を最適化
SnapGene は、制限酵素認識配列の確認やプライマー設計だけでなく、遺伝子の向きやフレームチェックなどを通して正しいコンストラクトを提供します。コンストラクトの元となった配列と現在の配列の比較、関連部分の強調などクローニング作業の過程を視覚的に再現します。
グラフィカルで理解しやすいインターフェイスは、クローニング作業計画の作成にかかる時間とコストを削減します。
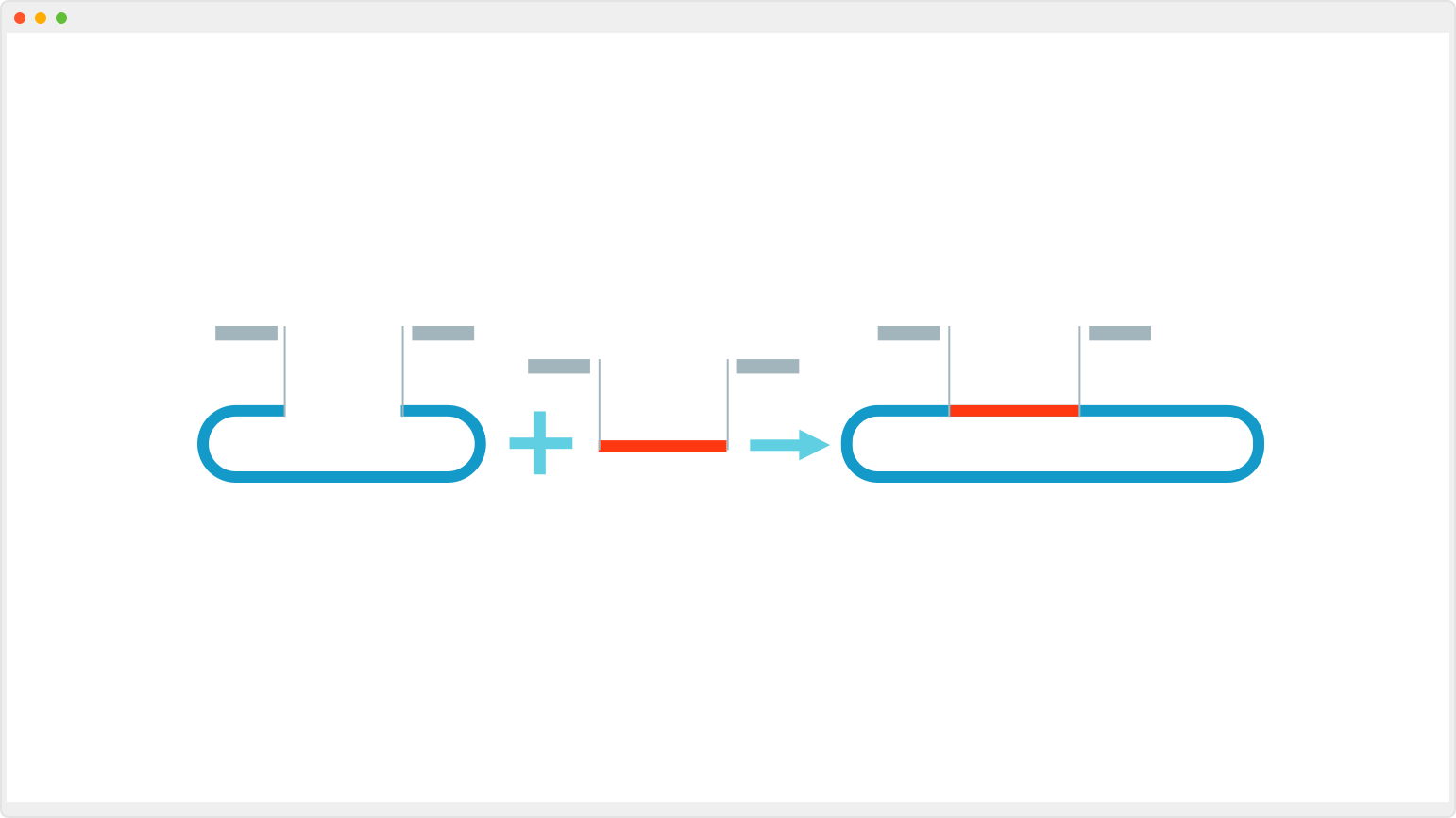
クローニング作業の視覚化
SnapGene は核酸配列やアミノ酸配列の視覚化、コンストラクトに対するシークエンス結果を可視化し、スムーズな管理作業をサポートします。
また、遺伝子工学の作業において、自分が何をしているのかを可視化することができれば問題が生じた時に原因を特定しやすく、またクローニング作業の心理的なハードルも大きく下がります。ヒューマンコンピューターインタラクション(HCI)研究 SnapGene は、作業内容を直感的な把握を可能にしスムーズな管理作業を提供します。詳細
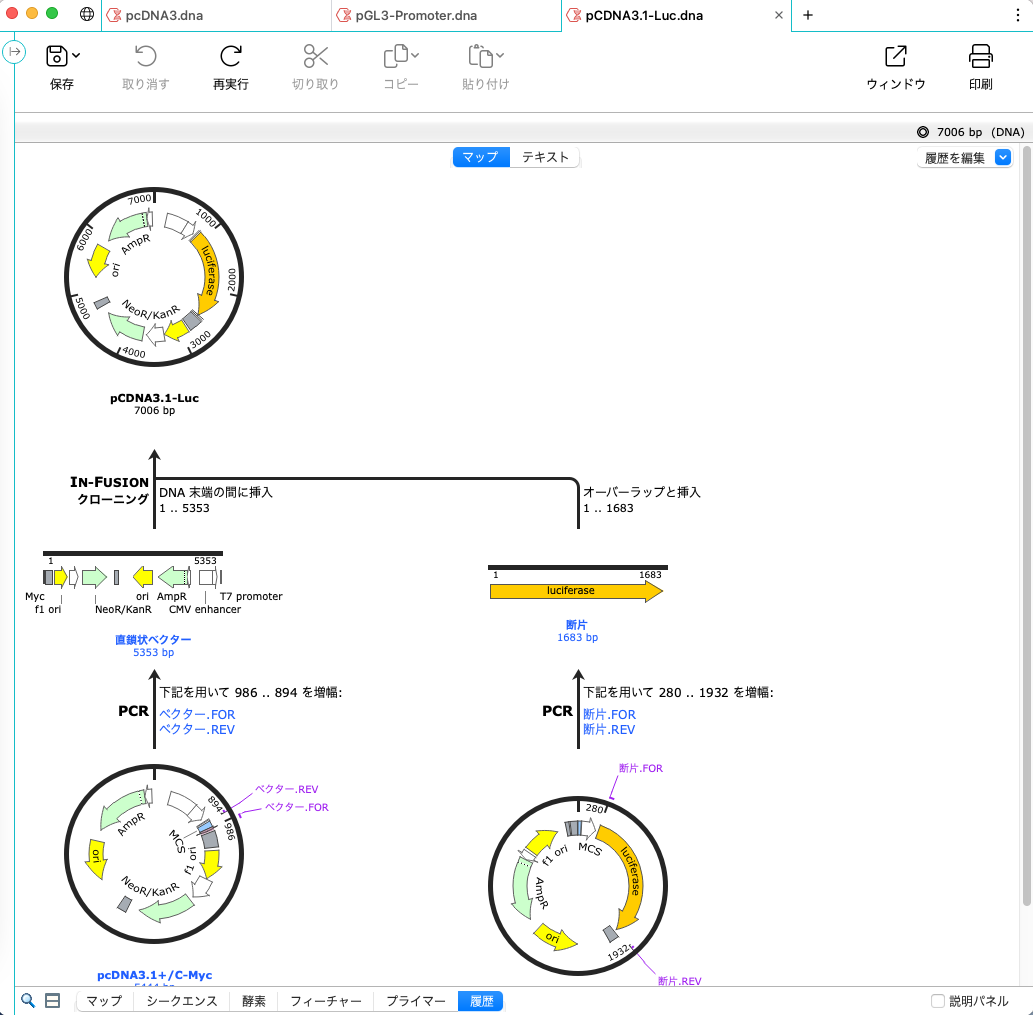
作業記録の自動化
SnapGene はドキュメント作成を自動で行うので、作業記録を別途作成する必要はありません。配列編集やクローニング手順の履歴がグラフィカルに自動生成されます。
プラスミドのコンストラクト作成に関わるシークエンス編集やクローニング手順の確認・共有を簡単に行うことができ、プロジェクトでの円滑な共同作業を支援します。詳細
分子クローニングの基礎から応用へ
遺伝子クローニングの手順を最適化
スピーディーで正確、ユーザーフレンドリーなインターフェイス
- コンストラクトを可視化することでクローニング手順の設計上の問題点を直感的に特定および修正することが可能
- 既存のプライマーを使用して標準的なPCRをシミュレー
- SnapGeneを利用し新たなプライマーを設計
- 専用のクローニングツールにより、主要な分子クローニング技術に対応したコンストラクト設計を正確かつ迅速に実施
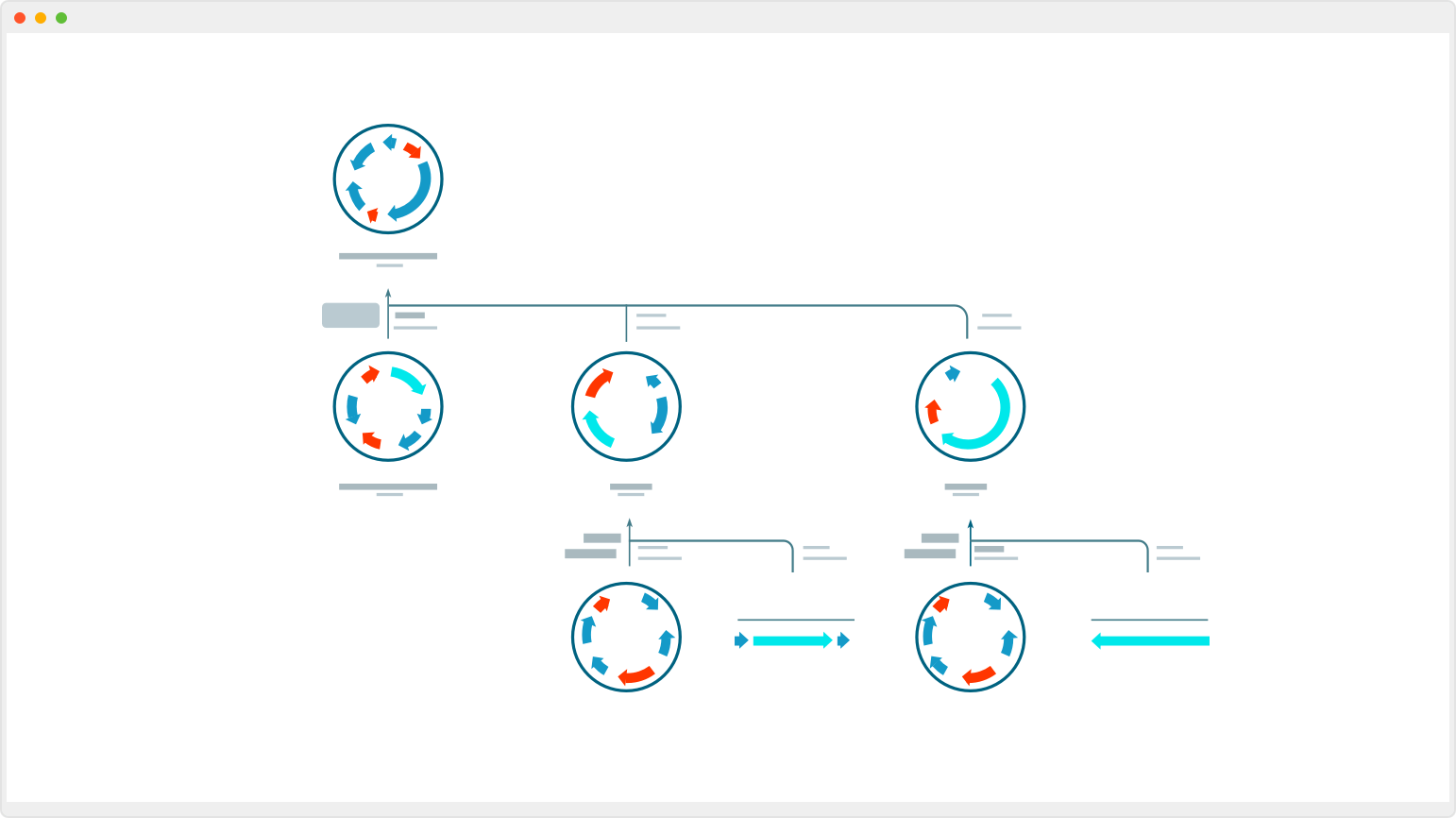
クローニング シミュレーション
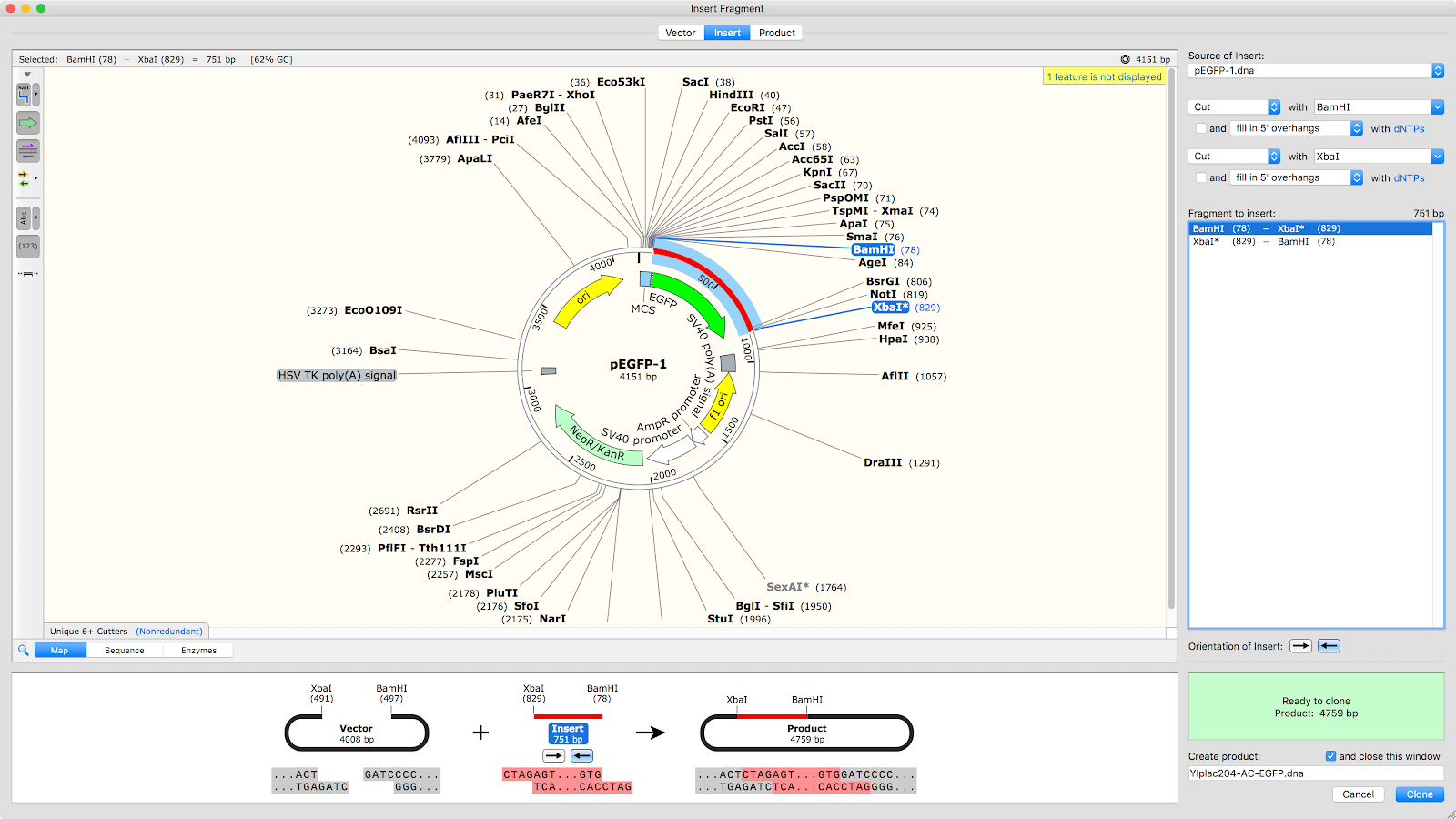
制限酵素クローニング
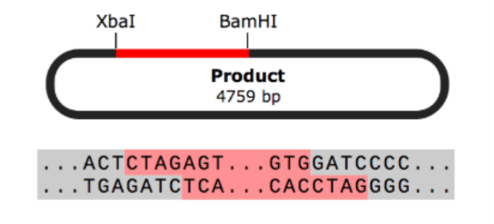
SnapGeneでは、使用する制限酵素の選択からクローニング後のイメージまで、制限酵素とライゲーションによるクローニングのシミュレーションを簡単に行うことができます。またプロトコルに設計上の欠陥がある場合、そのエラーを発見して修正する機能も備えています。
チュートリアルもご参照ください:
制限酵素処理によるクローニング
Gateway®クローニング
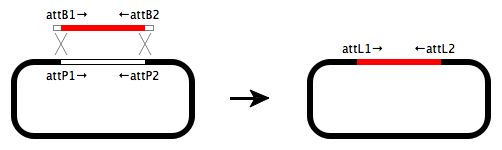
SnapGeneでは、Gateway®クローニング法の様々なバリエーションをシミュレートすることが可能です。
Gibson Assembly®
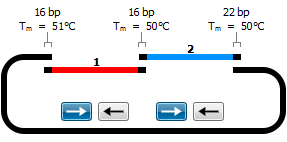
SnapGene は、Gibson Assembly®の方法を直感的にシミュレーションすることが可能です。Gibson Assembly®で必要なプライマーも自動で設計され、ユーザーが行う操作は、融合するDNA断片を選択するだけです。
チュートリアルもご参照ください:
Gibson Assembly®
Golden Gate® アッセンブリー
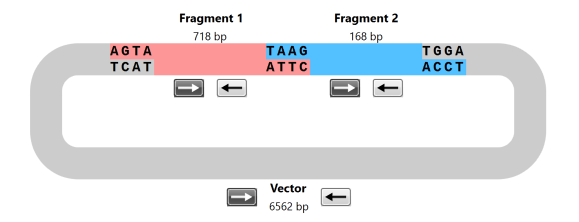
SnapGeneはプライマー設計を自動化し、Golden Gate®アッセンブリーを簡素化します。ユーザーが構築したいDNA断片を選択するだけで、SnapGene がプライマーとオーバーハングを設計し、反応の忠実度を最適化し成功の可能性を最大限に引き出します。
クローニング法の詳細はこちらから:
NEB Golden Gate® Assembly
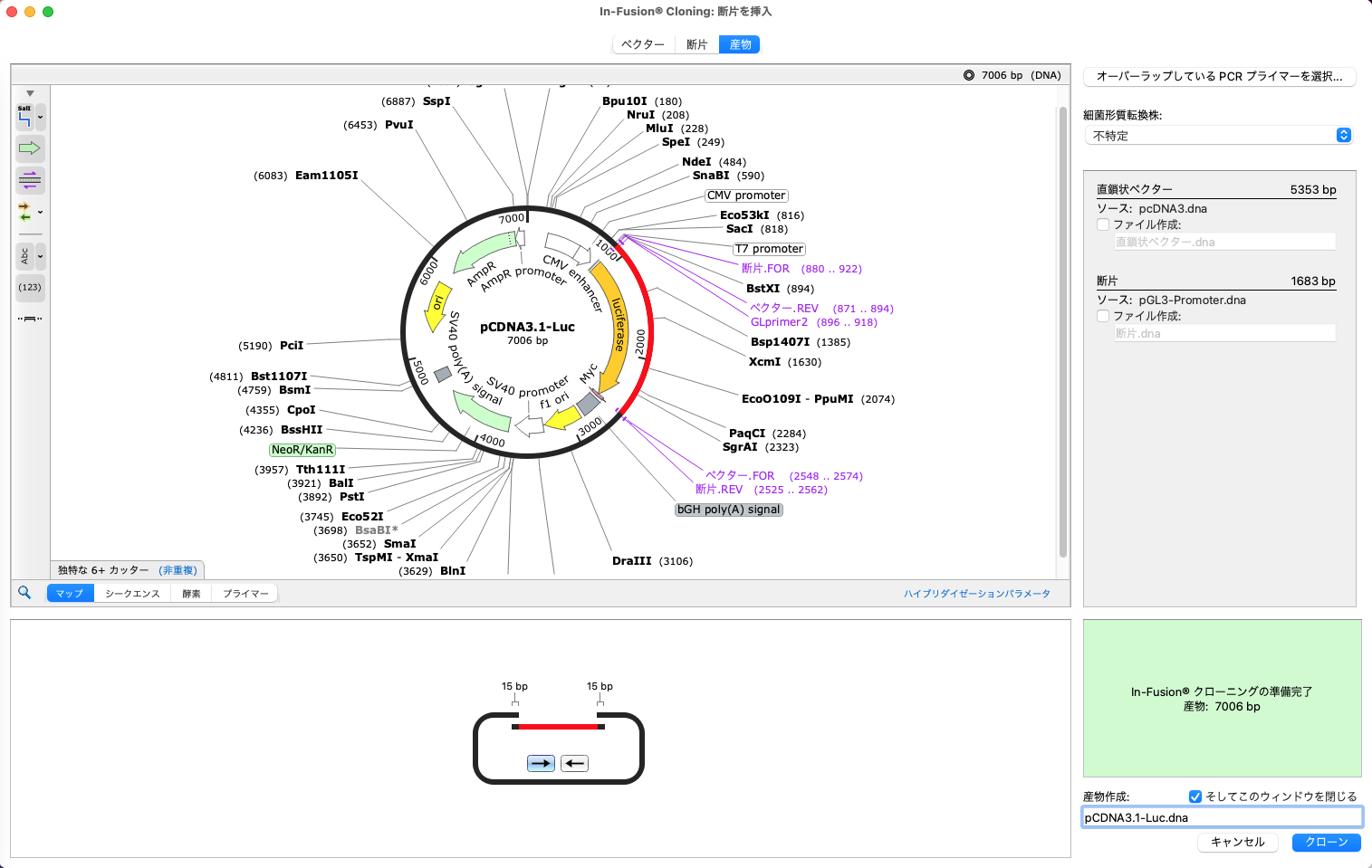
In-Fusion®クローニング
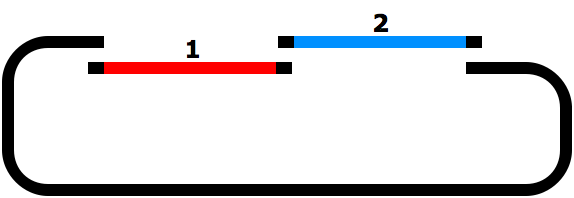
SnapGene は、In-Fusion® クローニングで必要なプライマーを自動で設計します。ユーザーが行う操作は、融合するDNA断片を選択するだけです。
チュートリアルもご参照ください:
In-Fusion®クローニング
NEBuilder HiFi DNA® アッセンブリー
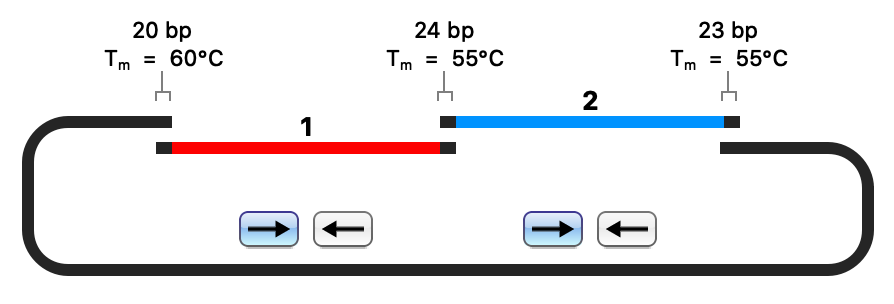
SnapGeneはプライマー設計を自動化し、NEBuilder HiFi DNA® アッセンブリーを簡素化します。ユーザーが構築したいDNA断片を選択するだけで、SnapGene が適切なプライマーを選択します。また、プライマー自動設計では、アセンブリー時に制限部位全体を除去することも可能です。
クローニングの詳細はこちらから:
NEBuilder HiFi DNA®Assembly
TAまたはGCクローニング
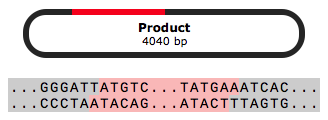
SnapGeneには、市販されている様々な直鎖状 TA および GC クローニングベクターのデータが既に組み込まれています。クローニングのシミュレーションはもちろん、データから目的にあった最適なベクターを選ぶ作業も可視化します。
Directional TOPO®クローニング
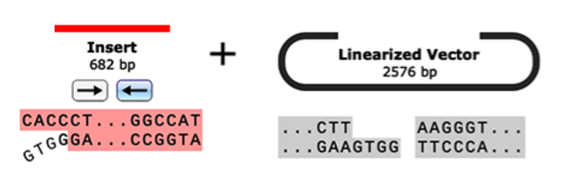
SnapGeneでは、TOPO TA cloning、Blunt TOPO cloning、Directional TOPO cloningの3つの異なるバリエーションのシミュレーションを実行できます。シミュレーションでは、TOPO® cloning で必要なプライマーを自動で設計します。ユーザー側で行う操作は、増幅するDNAフラグメントを選択し、組み込まれているリストからTOPO®クローニングベクターを選ぶだけです。
クローニング計画を検証し、技術的なエラーを回避
クローニング作業の視覚化
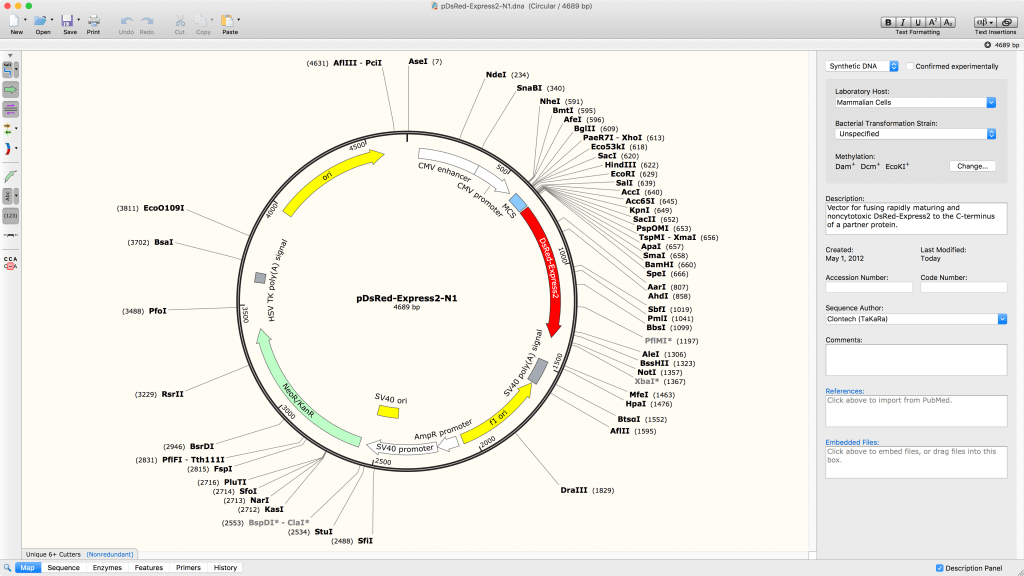
クローニング計画を検証し、技術的なエラーを回避
- SnapGeneのフィーチャーデータベースや独自のカスタムフィーチャーを使用し、プラスミドのフィーチャーへのアノテーション追加が可能
- 制限酵素サイト、フィーチャー、プライマー、ORF、翻訳などをプラスミドマップやシークエンスビューで詳細に表示
- 柔軟なアノテーションコントロールと視覚化コントロールでプラスミドマップを自分好みにカスタマイズ
参照 SnapGne チュートリアル
制限酵素処理によるクローニングIn-Fusion®クローニング
Gibson Assembly®
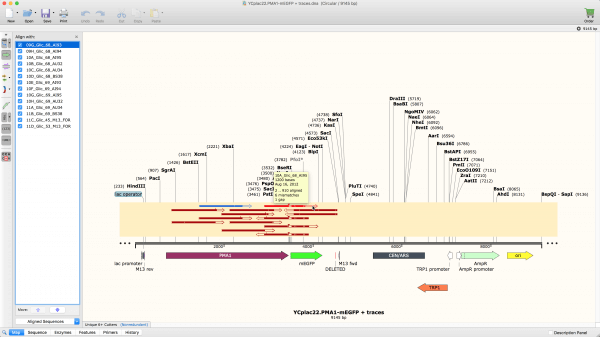
アライメントツールによる配列の確認
- 強力なAlign to Reference(リファレンス配列に対するアライメント)ツールを使用し、シークエンスされたコンストラクトとシミュレーションされたコンストラクトの一致を検証
- Clustal Omega、MAFFT、MUSCLE、T-Coffee などの信頼できるアルゴリズムを使用したペアワイズアライメントおよびマルチプルアラインメント
- CAP3を使用した、サンガーシークエンスリードの完全なコンティグを構築
参照 SnapGne チュートリアル
DNA/RNA配列のペアワイズアライメント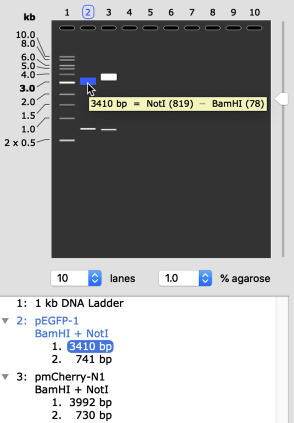
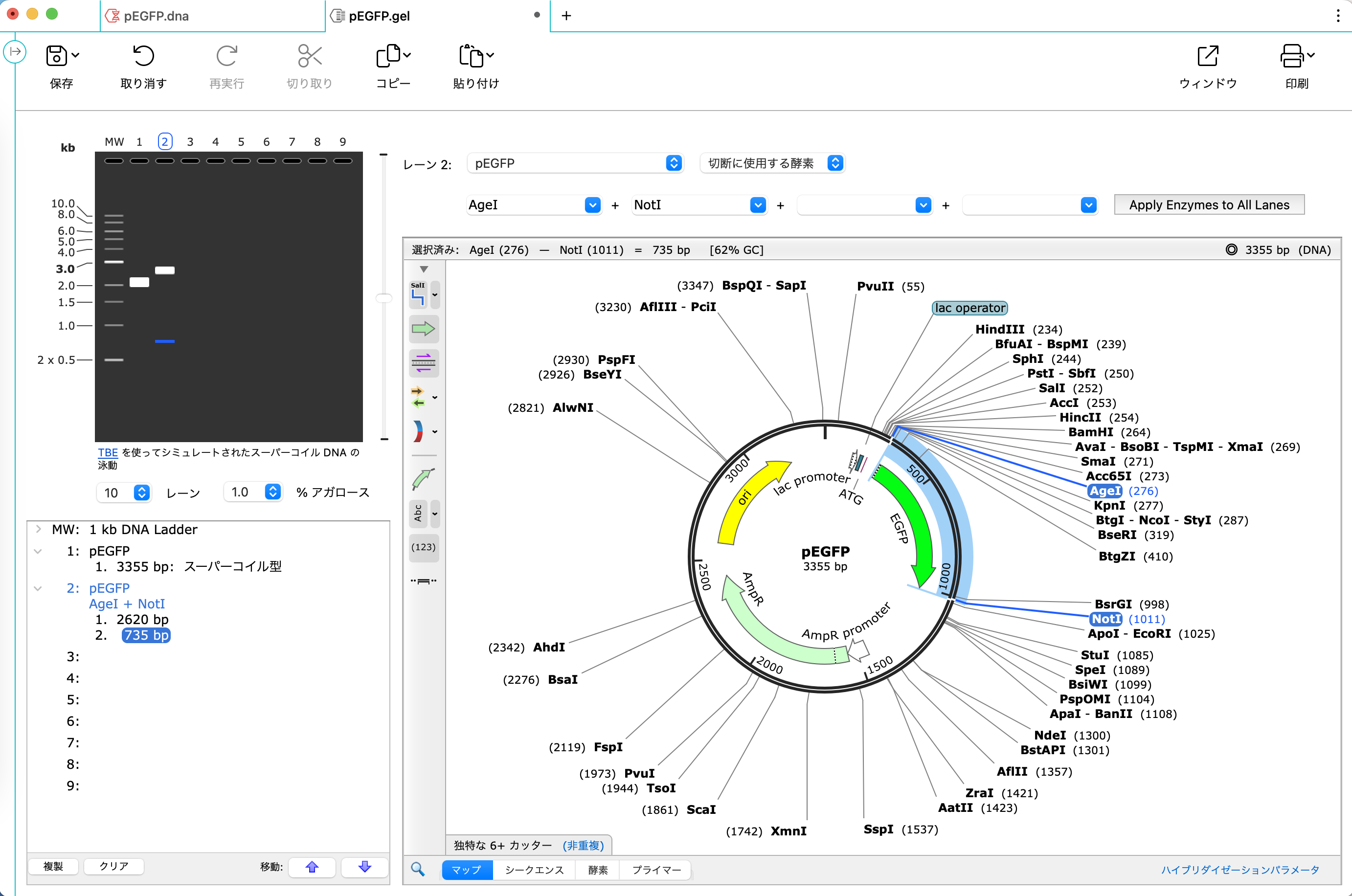
アガロースゲル電気泳動の泳動パターンをシミュレーション
- SnapGene のゲルシミュレーションアルゴリズムを使用したアガロースゲル電気泳動の泳動パターンの再現
- レーン数、アガロースの割合、実行時間、MWマーカーのフルセットなどの要素を柔軟にカスタマイズ可能
- 各レーンの詳細なフラグメント情報により、目的のバンドを記録し識別
参照 SnapGne チュートリアル
アガロースゲル電気泳動像自動文書化とスムーズなデータ交換のための設定
作業記録の自動化
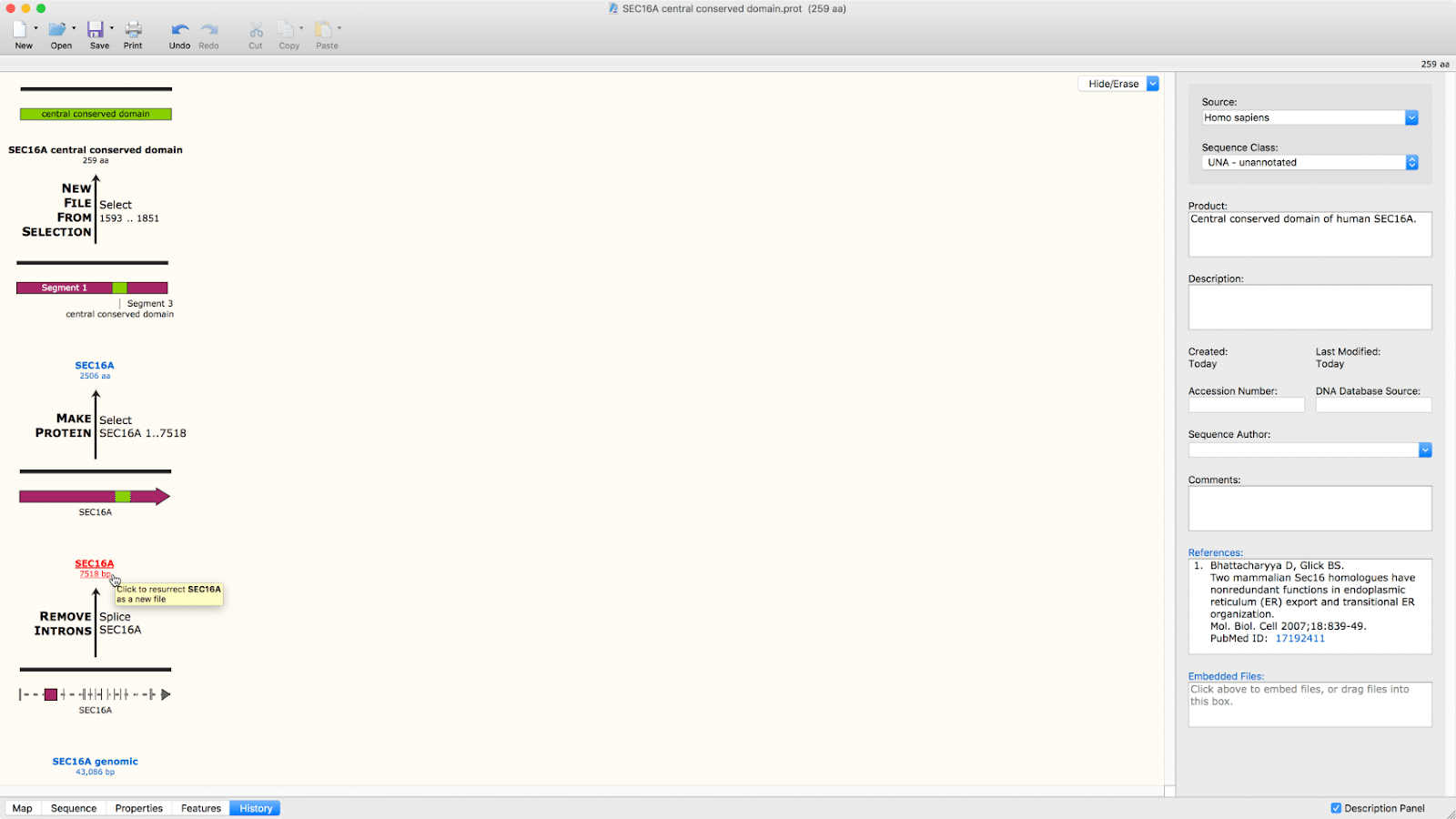
グラフィカルな履歴を自動で作成
- 操作手順やドキュメントの変更を視覚的に記録
- 履歴、遺伝子マップ、シークエンスビューに履歴の色を表示
- 完全に復元可能なシークエンス履歴のドキュメントへの埋め込み
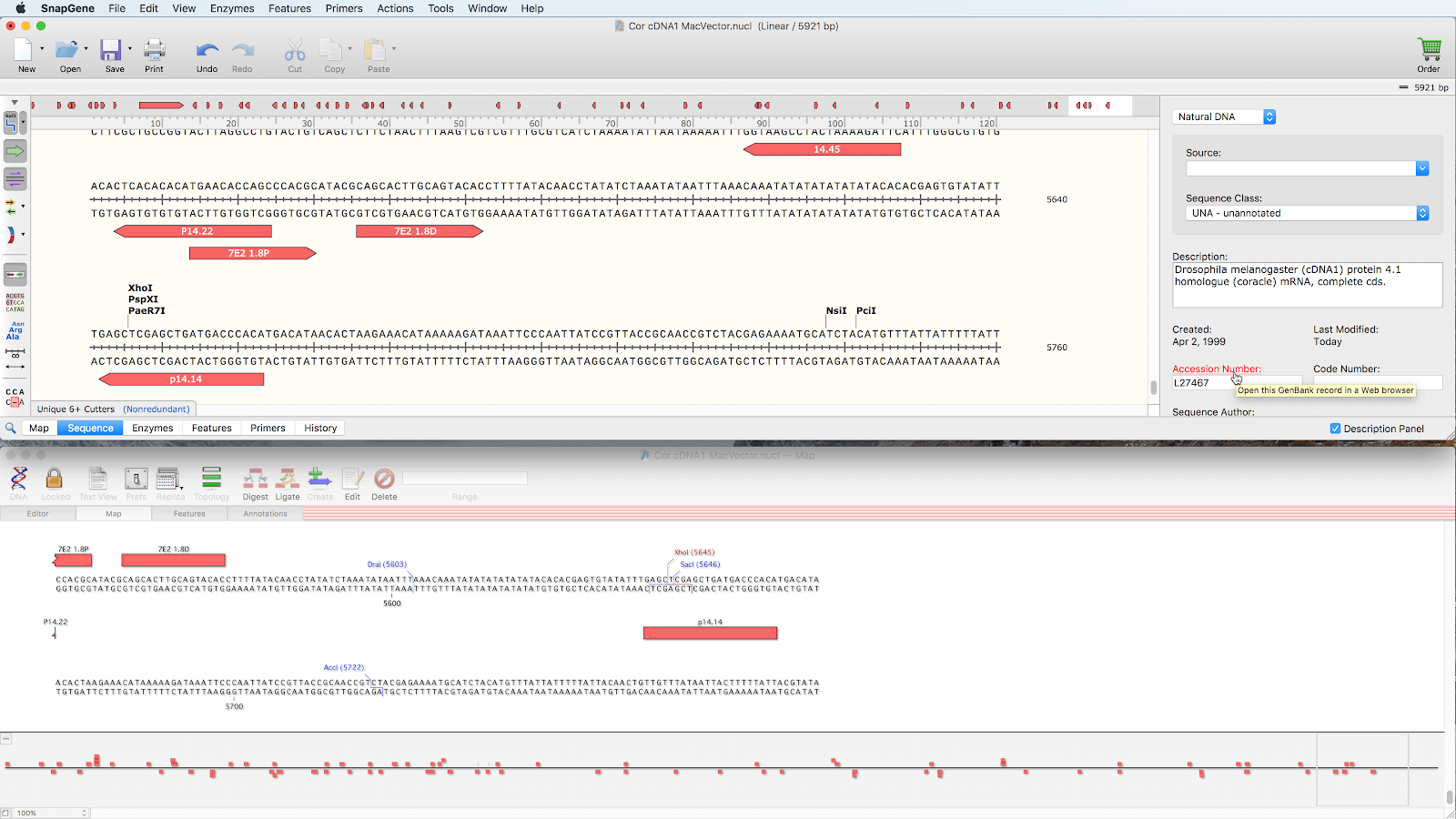
データの変換と共有
- シークエンス、ファイル、各種マップの保存、検索、共有、整理
- 共有可能なドライブ、サーバー、クラウドサービスに保存されたシークエンスを使用しての共同作業が可能
- 多くの一般的なファイルフォーマットから、シークエンス、アノテーション、その他のメタデータを読み取り、それらをエクスポートすることも可能
多くのユーザーに支持されています
SnapGeneは分子生物学研究を支援する強力なツールとして多くの研究者に利用されています。またSnapGeneを使用した論文は、医学・ライフサイエンス分野のジャーナルに広く掲載されています。
世界中の研究者に評価されています
SnapGeneの使い勝手の良さに、すぐに私の研究室に導入を決意しました。まず最初にSnapGeneでクローニングを行い、実験計画における潜在的課題を見つけてから作業を行っています。おかげで、時間とコストを大幅に減らすことができるようになりました。
"SnapGene is so easy to use that my lab adopted it instantly. It saves a lot of time and money as we now do all our cloning in SnapGene first and catch any snags in the strategy before they hold us up."
A. Matouschek
Northwestern University
SnapGeneはとても素晴らしいソフトです。すぐに使い始めることができるほど簡単なのに、優れた機能が豊富で、私がクローニングに求めている点をすべてカバーしています。クローニングを行う人にぜひお勧めしたいソフトウェアです。
"SnapGene is awesome. It is straightforward enough to start using right away, yet rich with sophisticated features that cover all of my cloning needs. I recommend this software to anyone who clones DNA."
D. Strongin
Fred Hutchinson Cancer Center
難しいクローニングに自信を持って、そして最小限の労力で取り組むことができるようになり、私の研究室でのリサーチが劇的に変わりました。私にとってSnapGeneはなくてはならない存在です。
"I just want to say that I LOVE SnapGene. It has dramatically altered the research that my lab does, giving us the confidence to take on difficult cloning with minimal effort! "
D. Bryant
University of Glasgow
SnapGene が使用された論文・ジャーナル
SnapGene は、遺伝子工学・分子生物学・細胞生物学・薬理学など生命科学分野における研究に利用され、解析結果やグラフが数多くの論文で引用されています。
- Reduction of UreB and CagA expression level by siRNA construct in Helicobacter pylori strain SS1
-
"The pGPU6/GFP/Neo plasmid constructs model were designed using SnapGene software (version 5.2.3) . Each constructs model was in silico analyzed for specific restriction enzyme pattern using SnapGene software. "
Motamedi H, Abiri R, Salari F, Jalili C, Alvandi A. Reduction of UreB and CagA expression level by siRNA construct in Helicobacter pylori strain SS1. BMC Microbiol. 2023 Dec 19;23(1):401. doi: 10.1186/s12866-023-03143-x. PMID: 38114907; PMCID: PMC10729366.
- The amyloid plaque proteome in early onset Alzheimer’s disease and Down syndrome
-
"Nucleotide sequences and ORFs were analyzed using SnapGene (GSL Biotech, San Diego, CA, USA, version 6.0.5) and BLAST on the NCBI website [21]. Sequence analysis, alignments, and phylogenetic analysis were performed using MEGA X (version 10.2.4) and SnapGene. "
Lutz, T., Langer, G. & Heinze, C. Complete genome sequence of a novel alternavirus infecting the fungus Ilyonectria crassa. Arch Virol 168, 34 (2023). https://doi.org/10.1007/s00705-022-05652-y
- One-step CRISPR-Cas9-mediated knockout of native TCRαβ genes in human T cells using RNA electroporation
- "A pST1 plasmid vector containing rationally designed eSpCas9(1.1)3 linked to an enhanced green fluorescent protein (EGFP) reporter was designed for in vitro production of Cas9 mRNA. The plasmid vector representation was created with SnapGene."
Flumens D, Campillo-Davo D, Janssens I, Roex G, De Waele J, Anguille S, Lion E. One-step CRISPR-Cas9-mediated knockout of native TCRαβ genes in human T cells using RNA electroporation. STAR Protoc. 2023 Feb 10;4(1):102112. doi: 10.1016/j.xpro.2023.102112. Epub ahead of print. PMID: 36853667; PMCID: PMC9943861.
- Alternative splicing and genetic variation of mhc-e: implications for rhesus cytomegalovirus-based vaccines
-
"In cases where the band size exceeded Sanger Sequencing limitations, forward and reverse primers were designed in the canonical Mamu-E exon 4 to pair with the PCR primers and produce two overlapping sequences for the band. The resulting sequence trace files were imported into SnapGene and exported to produce fastq files. "
Brochu, H., Wang, R., Tollison, T. et al. Alternative splicing and genetic variation of mhc-e: implications for rhesus cytomegalovirus-based vaccines. Commun Biol 5, 1387 (2022). https://doi.org/10.1038/s42003-022-04344-2
- The amyloid plaque proteome in early onset Alzheimer’s disease and Down syndrome
-
"Unpurified PCR products were submitted to Genewiz for Sanger sequencing, and the sequences were analyzed using SnapGene 5.3.1 software."
Drummond, E., Kavanagh, T., Pires, G. et al. The amyloid plaque proteome in early onset Alzheimer’s disease and Down syndrome. acta neuropathol commun 10, 53 (2022). https://doi.org/10.1186/s40478-022-01356-1
- Single molecule, long-read Apoer2 sequencing identifies conserved and species-specific splicing patterns
-
"Sequences were analyzed and compared to their corresponding reference genome in NCBI and exons annotated manually using SnapGene software. "
Christina M. Gallo, Adam T. Labadorf, Angela Ho, Uwe Beffert,Single molecule, long-read Apoer2 sequencing identifies conserved and species-specific splicing patterns,Genomics,Volume 114, Issue 2, 2022, 110318, ISSN 0888-7543, https://doi.org/10.1016/j.ygeno.2022.110318.
- Development of RNA G-quadruplex (rG4)-targeting L-RNA aptamers by rG4-SELEX
-
"Aptamer sequences can appear in either sense or antisense strands within the sequencing data. To analyze these data, we used the SnapGene software (GSL Biotech) following the steps below. "
Umar, M.I., Chan, CY. & Kwok, C.K. Development of RNA G-quadruplex (rG4)-targeting L-RNA aptamers by rG4-SELEX. Nat Protoc 17, 1385–1414 (2022). https://doi.org/10.1038/s41596-022-00679-6
- Modeling mRNA-based vaccine YFV.E1988 against yellow fever virus E-protein using immuno-informatics and reverse vaccinology approach
-
"Vaccine peptide 3D structure was predicted and validated based on the Ramachandran plot. Finally, we designed the vaccine construct for simulating restriction cloning using the SnapGene tool. "
Khan NT, Zinnia MA, Islam ABMMK. Modeling mRNA-based vaccine YFV.E1988 against yellow fever virus E-protein using immuno-informatics and reverse vaccinology approach. J Biomol Struct Dyn. 2023 Mar;41(5):1617-1638. doi: 10.1080/07391102.2021.2024253. Epub 2022 Jan 7. PMID: 34994279.
- Development of a novel PTD-mediated IVT-mRNA delivery platform for potential protein replacement therapy of metabolic/genetic disorders
- "Each IVT-mRNA (5′ UTR-SCO2-3′ UTR, simplified as IVT-mRNA of SCO2/5′ UTR-β-globin-3′ UTR, simplified as IVT-mRNA of β-globin) was prepared by the T7 polymerase-based in vitro transcription method and purified by phenol-chloroform after linearization of the recombinant plasmid vector by digestion with the XbaI restriction enzyme. IVT-mRNA maps are also shown via SnapGene software."
Miliotou AN, Pappas IS, Spyroulias G, Vlachaki E, Tsiftsoglou AS, Vizirianakis IS, Papadopoulou LC. Development of a novel PTD-mediated IVT-mRNA delivery platform for potential protein replacement therapy of metabolic/genetic disorders. Mol Ther Nucleic Acids. 2021 Sep 20;26:694-710. doi: 10.1016/j.omtn.2021.09.008. PMID: 34703653; PMCID: PMC8517095.
- Design of a novel multiple epitope-based vaccine: An immunoinformatics approach to combat SARS-CoV-2 strains
-
"Results: The constructed vaccine showed good results of worldwide population coverage and promising immune response. This constructed vaccine was subjected to in-silico immune simulations by C-ImmSim. Chimeric protein construct was cloned into PET28a (+) vector for expression study in Escherichia coli using snapgene."
Naveed M, Tehreem S, Arshad S, Bukhari SA, Shabbir MA, Essa R, Ali N, Zaib S, Khan A, Al-Harrasi A, Khan I. Design of a novel multiple epitope-based vaccine: An immunoinformatics approach to combat SARS-CoV-2 strains. J Infect Public Health. 2021 Jul;14(7):938-946. doi: 10.1016/j.jiph.2021.04.010. Epub 2021 May 4. PMID: 34119848; PMCID: PMC8093003.
- Immunoinformatics prediction of overlapping CD8+ T-cell, IFN-γ and IL-4 inducer CD4+ T-cell and linear B-cell epitopes based vaccines against COVID-19 (SARS-CoV-2)
-
"Fig 2 - The whole genome sequence (GenBank number MN908947.2) of COVID-19 built using Snapgene showing the position of the promising CD4 + T-cell epitopes and B-cell epitopes. The CD4 + T-cell epitopes were named from E1-CD4 to E19-CD4 representing alphabetically the epitopes as found in Table 2, the B-cell epitopes number E1-Bcell to E8-Bcell represented as found in Table 3."
Fatoba, Abiodun & Maharaj, Leah & Teniola, Adeleke & Okpeku, Moses & Adeniyi, Adebayo & Adeleke, Matthew. (2021). Immunoinformatics prediction of overlapping CD8+ T-cell, IFN-γ and IL-4 inducer CD4+ T-cell and linear B-cell epitopes based vaccines against COVID-19 (SARS-CoV-2). Vaccine. 39. 10.1016/j.vaccine.2021.01.003.
- The Essential Facts of Wuhan Novel Coronavirus Outbreak in China and Epitope-based Vaccine Designing against COVID-19
-
"The optimized DNA sequence was then taken and SgrA1 and SphI restriction sites were attached to the N-terminal and C-terminal sites, respectively. Finally, the SnapGene restriction cloning module was used to insert the newly adapted DNA sequence between the SgrA1 and SphI restriction sites of pET-19b vector [170]-[174]."Bishajit Sarkar, Md. Asad Ullah, Fatema Tuz Johora, Masuma Afrin Taniya, Yusha Araf bioRxiv 2020.02.05.935072; doi: https://doi.org/10.1101/2020.02.05.935072
- Signal hotspot mutations in SARS-CoV-2 genomes evolve as the virus spreads and actively replicates in different parts of the world
-
"Nucleotide sequences of isolates from China, India, and Europe were analyzed with the program Snapgene (GSL Biotech) by using the algorithm MUSCLE (MUltiple Sequence Comparison by Log-Expectation). Amino acid sequences were also analyzed with the program Snapgene."
Weber, S., Ramirez, C., & Doerfler, W. (2020). Signal hotspot mutations in SARS-CoV-2 genomes evolve as the virus spreads and actively replicates in different parts of the world. Virus research, 289, 198170. https://doi.org/10.1016/j.virusres.2020.198170
- A Novel Technique for Constructing Infectious Cloning of Type 3 Porcine Circovirus
-
"In this study, according to the linear sequence of PCV3 DNA published on GenBank, the sequence was rearranged with SnapGene gene-editing software, and after rearrangement, the HindIII restriction endonuclease site was added to the end of the linear DNA, so that both ends have the same restriction endonuclease site."
Jiang Z, Wu J, Jiang M, Xie Y, Bu W, Liu C, Zhang G, Luo M. A Novel Technique for Constructing Infectious Cloning of Type 3 Porcine Circovirus. Front Microbiol. 2020 Jun 9;11:1067. doi: 10.3389/fmicb.2020.01067. PMID: 32582064; PMCID: PMC7296095.
- Extending SynBioHub's Functionality with Plugins
-
"This framework was tested by the development of three example plugins, one of each type as follows: one allowing the submission of SnapGene files, one visualizing the course of different genetic parts, and one preparing plasmid maps for download."
Mante J, Zundel Z, Myers C. Extending SynBioHub's Functionality with Plugins. ACS Synth Biol. 2020 May 15;9(5):1216-1220. doi: 10.1021/acssynbio.0c00056. Epub 2020 Apr 23. PMID: 32275821.
- ER arrival sites associate with ER exit sites to create bidirectional transport portals
-
"Cultures were grown in rich glucose medium (yeast extract, peptone, dextrose), synthetic glucose medium (SD), or nonfluorescent synthetic glucose medium (NSD; Bevis et al., 2002) in baffled flasks at 30°C with shaking at 200 rpm. P. pastoris gene sequences were obtained from the NCBI database. Molecular biology procedures were simulated and recorded using SnapGene software."
Sudeshna Roy Chowdhury, Chumki Bhattacharjee, Jason C. Casler, Bhawik Kumar Jain, Benjamin S. Glick, Dibyendu Bhattacharyya; ER arrival sites associate with ER exit sites to create bidirectional transport portals. J Cell Biol 6 April 2020; 219 (4): e201902114. doi: https://doi.org/10.1083/jcb.201902114
- Maturation-driven transport and AP-1–dependent recycling of a secretory cargo in the Golgi
-
"Video 7 shows fast 4D imaging of terminally maturing cisternae as both the cargo and the late Golgi markers depart. Data S1 ZIP file contains annotated SnapGene files for all of the plasmids used in this study."
Jason C. Casler, Effrosyni Papanikou, Juan J. Barrero, Benjamin S. Glick; Maturation-driven transport and AP-1–dependent recycling of a secretory cargo in the Golgi. J Cell Biol 6 May 2019; 218 (5): 1582–1601. doi: https://doi.org/10.1083/jcb.201807195
- TANGO1 and Mia2/cTAGE5 (TALI) cooperate to export bulky pre-chylomicrons/VLDLs from the endoplasmic reticulum
-
"Real-time PCR was performed with Light Cycler 480 SYBR Green I Master (Roche) according to the manufacturer’s instructions. SnapGene software (GSL Biotech) was used for molecular cloning design."
António J.M. Santos, Cristina Nogueira, Maria Ortega-Bellido, Vivek Malhotra; TANGO1 and Mia2/cTAGE5 (TALI) cooperate to export bulky pre-chylomicrons/VLDLs from the endoplasmic reticulum. J Cell Biol 9 May 2016; 213 (3): 343–354. doi: https://doi.org/10.1083/jcb.201603072
- Remodeling of secretory compartments creates CUPS during nutrient starvation
-
"SnapGene software (GSL Biotech; available at www.snapgene.com) was used for molecular cloning procedures. "
David Cruz-Garcia, Amy J. Curwin, Jean-François Popoff, Caroline Bruns, Juan M. Duran, Vivek Malhotra; Remodeling of secretory compartments creates CUPS during nutrient starvation. J Cell Biol 22 December 2014; 207 (6): 695–703. doi: https://doi.org/10.1083/jcb.201407119
SnapGene機能詳細
SnapGeneは、日常的に行う遺伝子クローニングの作業手順を向上させるために開発されています。
分子クローニング
- 制限酵素クローニング
- Gateway®クローニング
- Gibson Assembly®
- NEBuilder HiFi® アッセンブリー
- In-Fusion®クローニング
- TA-GCクローニング
- TOPO®クローニング
- Golden Gate® アッセンブリー
プライマー
- プライマーの設計
- 2つのオリゴをアニーリングして二重鎖産物の形成
PCRおよび変異導入
- シミュレートPCR
- オーバーラップ伸長PCR
- プライマー指向変異導入
- サイレント変異導入による制限酵素サイトの追加または削除
ファイルのフォーマットの変換
- アライメントフォーマット
- ApE
- CLC Bio
- Clone Manager
- DNA Strider
- DNADynamo
- DNASIS
- DNAssist
- DNASTAR Lasergene®
- DS Gene
- EMBL (ENA)
- EnzymeX
- GenBank / DDBJ
- Gene Construction Kit®
- Geneious
- GeneTool
- Genome Compiler
- Jellyfish
- MacVector
- pDRAW32
- Serial Cloner
- Swiss-Prot
- Vector NTI®
- Visual Cloning
酵素セット
- 制限酵素セット
- 定義済み制限酵素セット - 企業またはカッターによる
- カスタム制限酵素セットの作成
- 詳細な制限酵素情報の表示
- メチル化感受性とこれに関連するエラー回避に対する豊富なサポート
翻訳
- 翻訳されたフィーチャーの表示および修正
- オープン・リーディングフレーム(ORF)
- 全シークエンスの翻訳
- 遺伝子融合のリーディングフレームの確認
- アミノ酸配列への翻訳 (DNAから)
- 逆翻訳(アミノ酸配列から)
アライメント
- DNAシークエンスをリファレンスシークエンスとアライメント
- クローニングや変異導入の検証
- cDNAを染色体にアラインメント
- ペアワイズとマルチシークエンスのDNAとアミノ酸のアラインメント
- アライメント・アルゴリズムの選択 - Clustal Omega、MAFFT、MUSCLE
- T-Coffee
- コンティグ・アセンブリ
可視化
- 複数のDNAシークエンス図の表示
- 大きいシークエンスのサポート - 染色体サイズのシークエンスの閲覧
- DNAやアミノ酸のシークエンスの修正
- 色コードシークエンス
- RNA二次構造図の表示
履歴の追跡
- 包括的な「取り消し」機能
- 産物のグラフィック履歴の表示
- オプションの色の履歴を使用すると、シークエンスに対する最新の変更の識別が可能
アガロースゲル電気泳動の泳動パターンをシミュレート
- 制限酵素処理のシミュレート
- PCR反応の増幅のシミュレート
- MWマーカー情報の大規模な収集
全般
- プラットフォーム間の互換性 - Windows、macOS、Linux
SnapGeneシステム条件
SnapGeneのシステム条件は以下の通りです。インストール可能台数やライセンス許諾についてはFAQ:ライセンスについて をご参照下さい。
Windows
SnapGene 6.1 - 7.0: Windows 10* ~ 11
SnapGene 6.1 - 7.0: Windows 10 ~ 11
SnapGene 5.1 - 6.0: Windows 7 ~ 11
ご注意下さい:64bitのみに対応しており、31bit には対応していません。
Microsoft Surface Pro XやLenovo Yoga 5Gを含むARMベースのWindowsコンピュータでは動作しません。
mac OS
SnapGene 7.1: macOS 11 以降
SnapGene 6.1 - 7.0: macOS 10.14 以降
SnapGene 5.3 - 6.0: macOS 10.12 以降
SnapGene 5.2.5 以前: macOS 10.11.
ご注意下さい:SnapGene6 以前のバージョンはARM版には対応していません。
Linux
SnapGene 7.1: Ubuntu 22.04, CentOS 8 Stream or RedHat 8.6.
SnapGene 6.1 - 7.0: Ubuntu 20.04, CentOS 8 (2105) or RedHat 8.4.
Snapgene 5.3 - 6.0: Ubuntu 18.04, Centos 7 (1810) or RedHat 7.6.
SnapGene 4.0 - 5.2: Ubuntu 16.04, RedHat 6.6 & 7.2.
ご注意下さい:ARM版には対応していません 。
HD空き容量 各OS共通
メモリ容量:1 GB RAM
HD空き容量:250 MB
ディスプレイ: 1440 x 900以上
SnapGene製品カタログ
SnapGeneのデジタルカタログをダウンロードいただけます。