遺伝子工学実験支援ソフト:SnapGeneリリース ノート
Release Note : SnapGene8.0 新機能
SnapGene 8.0では、主なデータビューアの外観と操作性が一新され、一括アノテーションやデータ操作の新機能、参照配列へのアラインメントの編集方法が追加されました。
SnapGene チュートリアル:SnapGene の基本操作 で、SnapGene8.0 を使用して SnapGene の機能を紹介しています。こちらのチュートリアルも合わせてご参照ください。
複数ファイルの一括処理
フォルダパネルで選択した複数のファイルを一括で処理することができるようになりました。
フォルダパネルで複数のファイルを選択すると、選択した複数ファイルに共通のフィーチャーの検出、フィーチャーやプライマーの一括インポート、アライメント、アガロースゲルの一括シミュレートを簡単に実行できます。


視覚的で直感的に操作できるインターフェイス
ビューを切り替えるタブ1 がパネルの下方から上方に表示されるようになり、またツールバー2 のアイコンがシンプル化され視覚性が向上しました。表示方法やレイアウトに関する設定をトップツールバー3 から実行できるようになりました。

マップビュー | Map View

フィーチャービュー | Feature View
フィーチャー名がリスト表示され、右向きシェブロン [ ] をクリックすると詳細情報が表示されます.

プライマービュー | Primer View

コンテンツの分割表示 | Split Contents
ビューの切り替えボタンがパネル下部から上部に配置されます.
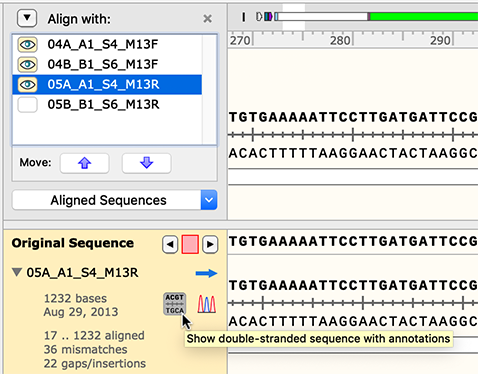
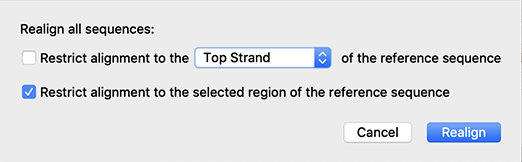
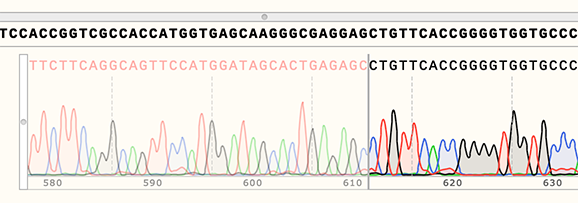
リファレンスDNA配列へのアラインメントの編集
リファレンス DNA シークエンスにアライメント(Align to a Reference DNA Sequence) 機能を使用する際に、サンガーシーケンシングのトレースのギャップ位置を編集して配列を検証できるようになりました。
ファイルとフォルダのインタラクションの強化
フォルダパネルに表示されるファイルがより視覚的になりました。未保存のファイル名にマークが表示され、一見して未保存ファイルを把握できるようになりました。また未保存ファイルがある場合は、ファイルリストの上部にアラート( "You have unsaved changes" )が表示され、変更が保存されていないファイルがあることを知らせます。直感的に未保存ファイルを選択でき、更に効率的なデータ管理が可能となりました。

フォルダパネル
ファイルに変更などが加えられた後、保存されていないファイル名の右横に黄色い丸印のアークが表示されます
.
SnapGene8.0 新機能・機能強化詳細
- 複数ファイルの一括操作
- フィーチャーをインポートしたり、複数のDNAファイルに共通するフィーチャーを検出します。
- リストまたはSnapGene、VNTI、Clone Managerファイルから複数の配列にプライマーをインポートします。
- 複数の塩基配列ファイルを環状、直鎖状、またはフリップします。
- メチル化、トポロジー、鎖の有無など、複数の配列のファイル属性を変更します。
- 複数のDNAファイルに対して表示する酵素を選択します。
- 複数の配列のDNAまたはRNA末端を編集します。
- 複数配列のDNA、RNA、タンパク質を作成します。
- 複数のファイルの履歴をトリミングします。
- プロジェクトにおけるファイル管理機能の強化
- フォルダを選択する機能を追加し、部分的なフォルダ選択の表示を追加します。
- 選択範囲全体をすばやくクリア、またはファイルの種類ごとに選択を解除します。
- 指定されたファイルタイプのファイル数の右のXをOpt/Altでクリックすることで、他のタイプのファイルの選択を解除することができます。
- プロジェクトウィンドウの最後のタブを閉じたときに、ファイル/フォルダの選択がクリアされるようになりました。
- ヘルプリンクが追加されました。
- 未保存の変更の視覚化と操作性の向上
- フォルダパネルとタブのファイルの横に黄色い点が表示されます。
- プロジェクトフォルダツリーの上部にアラートを表示します。
- プロジェクト内の未保存のファイルをすべて保存、すべて破棄、または選択するオプションが追加されました。
- マップビューとシーケンスビューの変更
- タブはパネル上部に、選択バーはパネル下部に表示されるようになりました。
- シーケンスビューのミニマップはパネル下部表示されるようになりました。
- 選択された酵素セットのインジケータは選択バーに表示されるようになりました。
- 左下のズームボタンを検索ボタンに置き換え、検索コントロールを切り替えられるようにしました。ズーム機能「表示メニュー > ズームコントロール」から、表示/非表示の切り替えが可能です。
- パネル下部にあった「説明パネル(Description Panel)」チェックボックスを、パネル上部の「Info 」ボタンに変更にしました。
- 左上の 「言語(Language) 」ボタンを 「設定(Settings) 」ボタンに変更しました。言語の切り替えは設定ボタンから行います。
- サイドツールバーをコンパクトにし、使用頻度の低い機能をサイドメニューや右上の表示ボタンに配置しました。表示オプションの設定に関係なく、一貫した予測可能なアイコンを表示するようにしました。
- 履歴表示の右上にあるコントロールと、履歴がない場合に表示されるメッセージのルック&フィールを更新しました。
- シークエンスが 「Confirmed Experimentally(実験的な確認)」であることを示す緑色のチェックマークアイコンを右上に追加しました。
- フィーチャーが表示されない際にマップビューで表示されるアラートの表示位置が右上から右下になりました。また、アラートを閉じることができるようにしました。
- フィーチャービューとプライマービューの変更
- 個々のフィーチャーとプライマーの展開と折りたたみができるようになりました。
- 検索フィールドがパネル上部に表示されるようになりました。
- 検索に一致した項目がテーブル内でハイライトされるようになりました。
- 列を逆方向にソートする機能を追加しました。
- カラーピッカーを使用して注釈の色を素早く変更する機能を追加しました。
- チェックボックスを追加し、選択範囲を変更できるようになりました。
- 代替開始コドンのツールチップを追加しました。
Release Note : SnapGene7.2 新機能
SnapGene 7.2 では、プライマーのホモ二量体構造を可視化する機能が追加されました。また、タブをドラッグ&ドロップによって別ウィンドウとして表示することが可能になり、プロジェクト内のファイルをインタラクティブに操作できるようになっています。
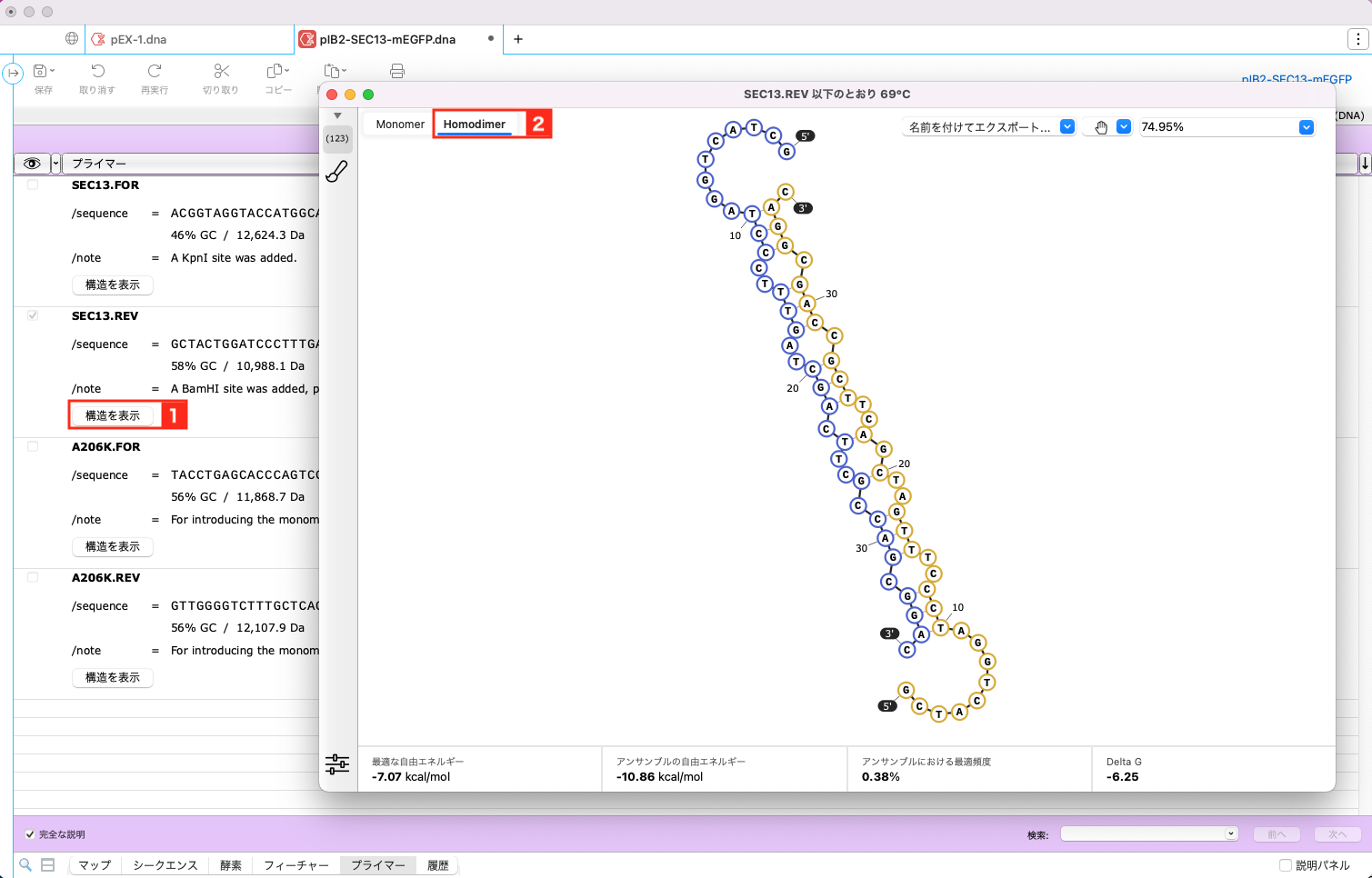
プライマーのホモ二量体構造の可視化
プライマータブに「構造を表示」ボタン1 をクリックすると、ウィンドウが開き選択したプライマーの二次構造が表示されます。ホモ二量体構造を表示するには、Homedimerタブ2 をクリックします。
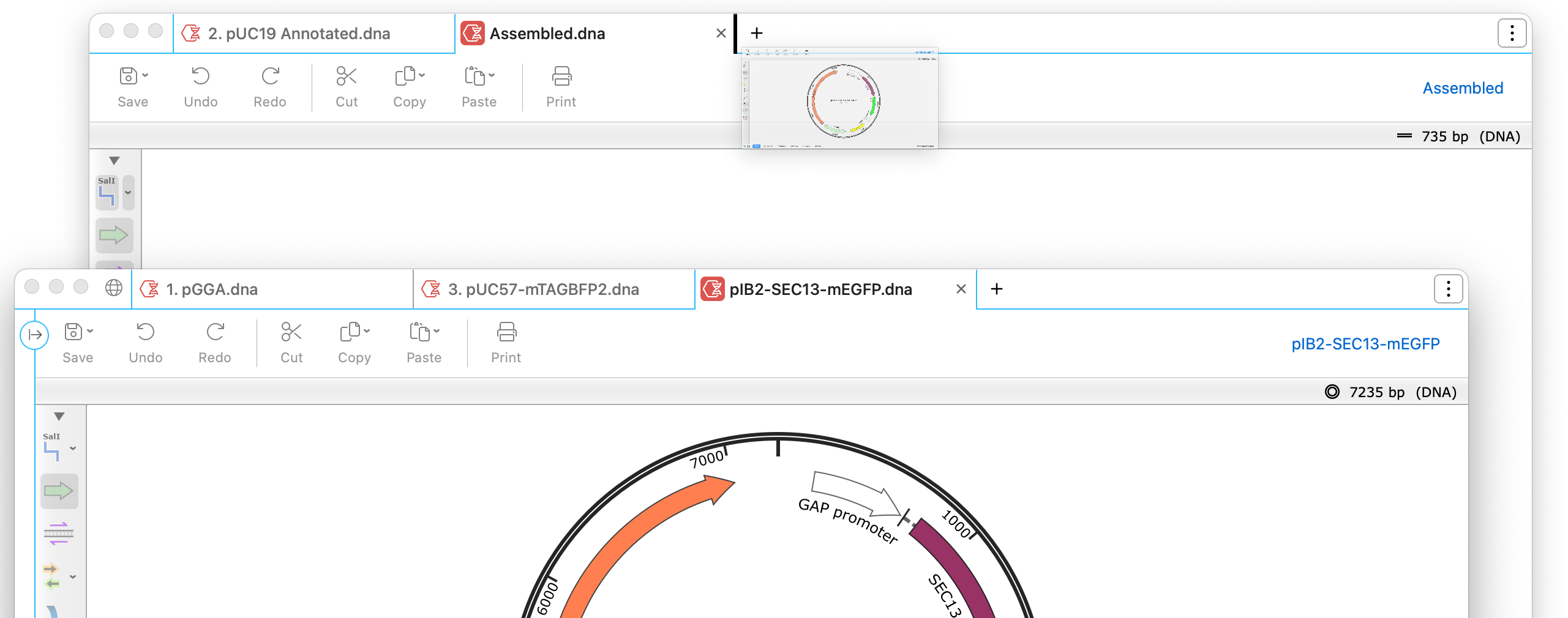
タブのドラッグ&ドロップ移動
タブをドラッグ&ドロップし、別のウィンドウで開く機能が追加されました。また、ドラッグ&ドロップで別のウィンドウにタブを挿入することも可能です。
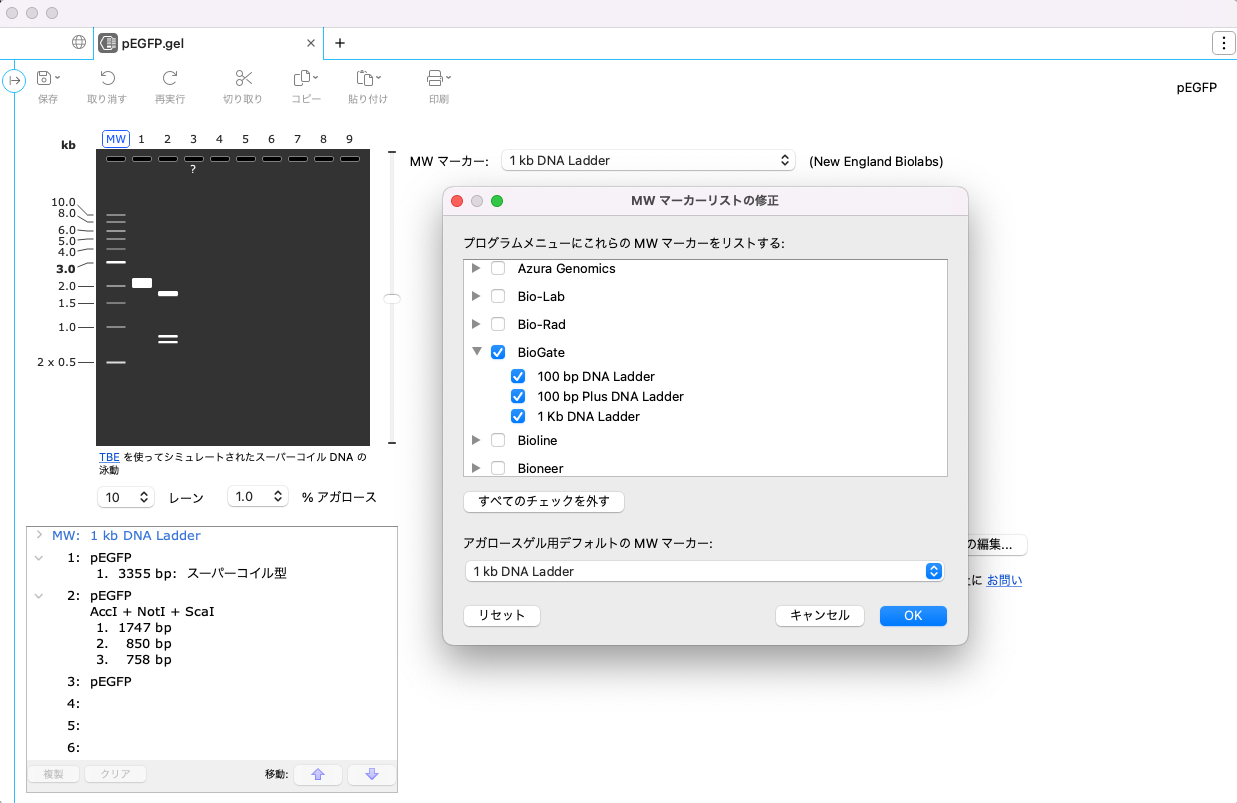
アガロースゲル電気泳動像シミュレーション機能追加
MWマーカーのサプライヤーに、BioGate、GeneAll、Hylabs が追加され、ゲルラダーを複数のサプライヤーから選択できるようになりました。
SnapGene7.2 新機能・機能強化詳細
- プライマーホモダイマー(ホモ二量体)
- Vienna RNAパッケージのRNAcofoldツールを用いてプライマーホモダイマー(ホモ二量体)構造を計算します。
- 配列または信頼度で色分けしてプライマーホモダイマーを視覚化します。
- タブ付きファイルウィンドウの機能強化
- ファイルタブをドラッグ&ドロップして別ウィンドウで開く、または既にあるウィンドウに挿入できるようになりました。
- タブ付きファイルウィンドウが "Project "パネルなしで存在できるようになリマした。
- 上部ツールバーに現在のファイル名を追加しました。
- 選択タブの視認性が向上しました。
- プロジェクト・インターフェイス内のデータ管理の強化
- ファイルをサブフォルダに直接インポートすることが可能になりました。
- ファイルパネルのアクションメニューに「すべてのフォルダを展開/折りたたむ(Expand/Collapse)」コマンドを追加しました。
- ファイルパネルでファイルを右クリックしたときに表示されるメニューに「ファイルを開く(Open File)」コマンドを追加しました。
- ダブルクリックでプロジェクト内のPDFが開くようになりました。
- クロマトグラムトレースファイルの表示設定の強化
- タブウィンドウ内でピークが引き伸ばされないようにデフォルトの高さを調整します。
- 品質データ上でマウスを動かすとピーク情報を表示します。
- トレースファイルのシーケンス上でマウスを動かすと、ベースハイライトが表示されるようになりました。
- In-Fusionクローニングのデフォルトを更新し、複数のフラグメントがある場合に20 bpのフラグメントを使用するようにしました。
- アガロースゲルシミュレーションにBioGateラダー(100 bp、100 bp Plus、1 KB)を追加しました。
- アガロースゲルシミュレーション用GeneAll GENESTAラダー(100 bp、250 bp、1 kb)を追加しました。
- アガロースゲルシミュレーション用Hylabsラダー(50bp、100bp、1KB+)を追加しました。
- 共通機能データベースの更新
- Anderson Constitutiveプロモーターセットを追加しました。
- 代替Bxb1インテグラーゼおよび関連部位を追加しました。
- mCyRFP、mScarlet3、StayGold、mStayGold蛍光タンパク質フィーチャーを追加しました。
- Csy4とヒトU6プロモーター配列を更新しました。
- サンプルプロジェクトにssDNAファイルを追加
Release Note : SnapGene7 新機能
2024/3/19 SnapGene7 がリリースされました。SnapGene7 で新たに導入されたタブ型インターフェースによって、データ管理機能が強化されました。ファイルやフォルダをタブで簡単に移動でき、配列やメタデータに基づくファイルの検索を新しいフォルダパネルから行うことができます。その他、新しいプライマー挿入アノテーションや様々な使い勝手の向上が図られています。
SnapGene 7.0で新たに導入されたタブ型インターフェースによって、データ管理機能が強化されました。ファイルやフォルダをタブで簡単に移動でき、配列やメタデータに基づくファイルの検索を新しいフォルダパネルから行うことができます。その他、新しいプライマー挿入アノテーションや様々な使い勝手の向上が図られています。
フォルダパネルによるファイルおよびフォルダ管理
ファイルシステム上の任意のフォルダを選択すると、フォルダパネルにプロジェクトフォルダとしてサブフォルダとファイルが表示されます。標準的なファイル操作を実行し、サポートされているすべてのファイルタイプをすばやくブラウズできます。
フォルダパネル機能
フォルダ Open Folder ボタンをクリックし、システムにある任意のフォルダ(プロジェクトフォルダ)を選択します。
選択したフォルダが、プロジェクトとして表示されます。

別のプロジェクトフォルダを開く場合、フォルダ名の右横にある (3点リーダー)をクリックし、「Open Project Folder」 を選択します。

新しいフォルダを追加する場合は、フォルダパネル上で右クリックし、メニューから「新しいフォルダ」を選択します。

タブ付きインターフェイス
新しいタブ付きインターフェイスを使用して、ドキュメント間を移動したり、比較のためにドキュメントを別のウィンドウにポップアウトすることができます。
タブ機能
保存されていないファイル は、タブ名の右側に・(黒丸)が表示されます。
は、タブ名の右側に・(黒丸)が表示されます。![]() はインポートして開いたファイルになります。これらのファイルは、保存しないとフォルダパネルに表示されません。
はインポートして開いたファイルになります。これらのファイルは、保存しないとフォルダパネルに表示されません。

ファイル検索
プロジェクト内のファイルを、名前やタイプ、配列、フィーチャーアノテーションなどの属性から素早く検索できます。
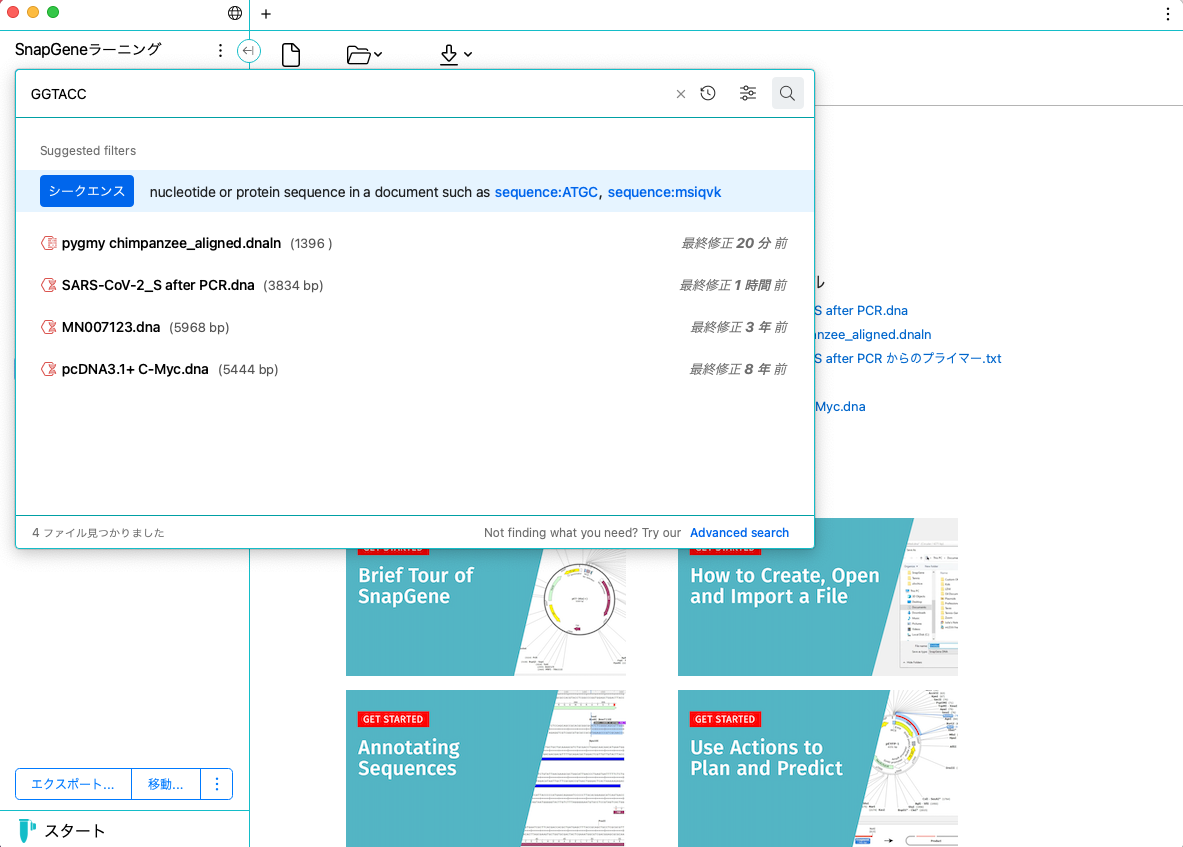
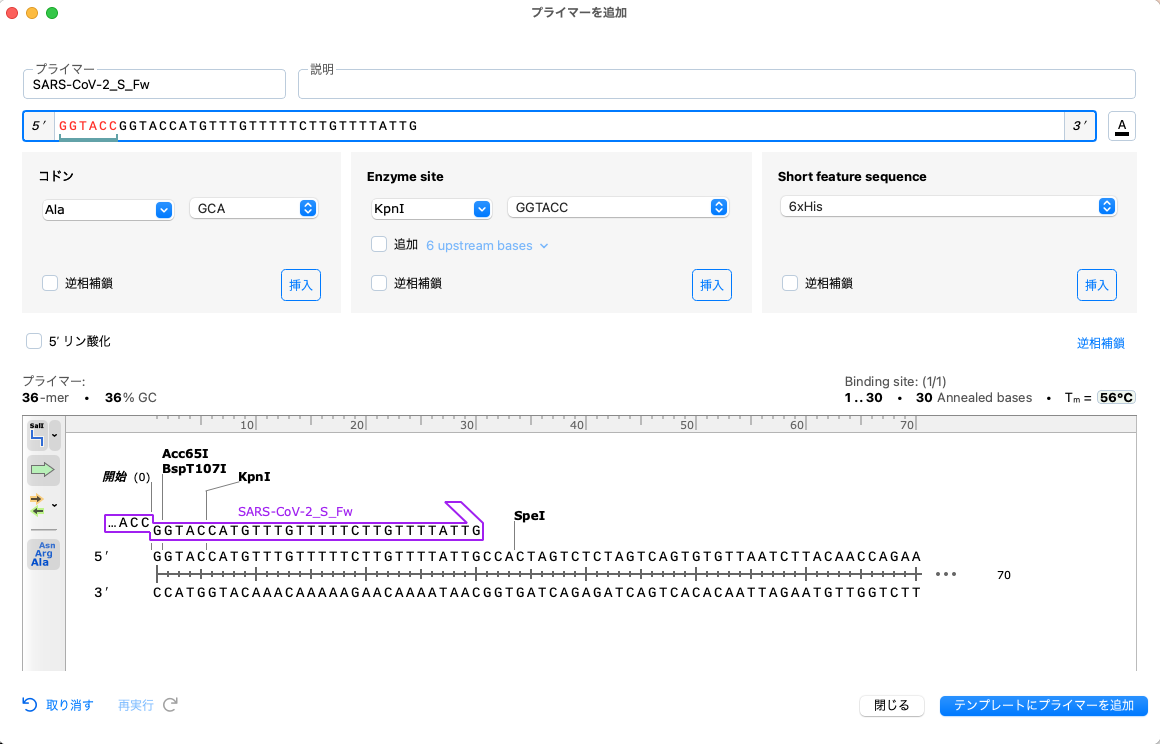
プライマーアノテーション
プライマーに追加された酵素、コドン、短いペプチド配列などの挿入物が、プライマーを追加ダイアログで視覚化されます。また、自動設計されたプライマーに対しても、関心のある領域(Golden Gate法の制限部位、Gateway クローニング法の AttB配列、ディレクショナル TOPOクローニング法のモチーフ)をアノテーションできるようになりました。
検出したフィーチャーのプレビュー
ファイルパネルにシーケンスマップが追加され、配列から検出されたフィーチャーをプレビューできるようになりました。また、配列に含めるアノテーションを選択できるようになりました。
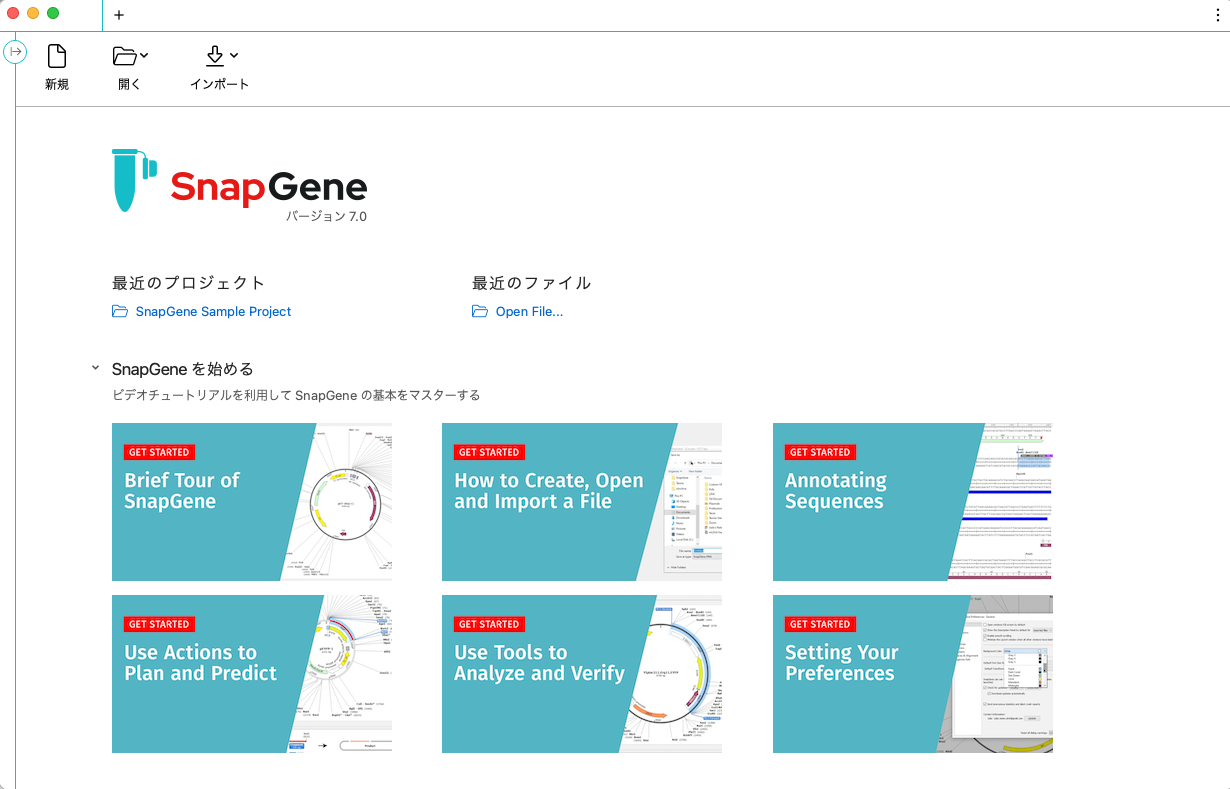
スタートアップダイアログ
スタートアップダイアログから、最近のファイルやフォルダにすばやくアクセスできるようになりました。また、ビデオチュートリアル、ユーザーガイド、SnapGeneアカデミーへのリンクを使用して、SnapGeneの使用方法について詳しく知ることができます。
SnapGene7 新機能・機能強化詳細
新たに追加された機能
- タブ表示によるインターフェイスが導入されました。
- データ管理強化 - ナビゲーションパネルにファイルとフォルダが階層的に表示されるようになりました。
- DNA、RNA、タンパク質、アラインメント、クロマトグラム、ゲルなど、SnapGeneがサポートするすべてのファイルタイプをすばやくブラウズすることができます。
- SnapGeneで標準的なファイル操作を直接実行できます。
- コンテキストベースのクイックリンクにより、一般的なツールに簡単にアクセスできます。
- 選択したプロジェクトフォルダ内のファイルを、ファイルや配列の特徴に基づいて検索できるようになりました。
- プライマーの追加・編集ダイアログおよび選択したクローニングダイアログでプライマーアノテーションを視覚的に表示する機能を追加しました。
- 新規配列作成時に検出されたフィーチャーをプレビューできるようになりました。
- リソースや最近のプロジェクトやファイルに簡単にアクセスできる Get Started が起動時に表示されるようになりました。
- フォルダパネルの配列選択に基づいて、選択したファイルをアラインメント、アセンブリクローニング法、ゲルシミュレーションへの直接入力が可能になりました。
- 複数配列のファイルをフォルダにインポートできるようになりました。
- プライマー配列への挿入をより簡単にできるように、プライマーダイアログを再構築しました。
- プライマーダイアログで、非縮退型認識配列をプライマー配列に挿入するオプションが追加されました。
- プライマーダイアログに表示される結合部位情報を改善しました。
- アガロースゲルのシミュレーション時にメチル化を変更できるようになりました。
- 日本ジェネティクス社のラダーを追加しました。
- SnapGene 内から Dotmatics Bioregister の配列を配列名、Bioregister ID、または類似配列を使って直接検索(BLAST 検索) SnapGene 内から Dotmatics Bioregister を直接読み込み検索することができるようになりました。
その他変更・バグフィックス
- すべてのアガロースゲルレーンにプライマーを適用するオプションを追加しました。
- 選択したフィーチャーから任意の位置まで(または、任意の位置から選択したフィーチャーまで)の選択範囲を拡張する機能を追加しました。
プライマーに5'制限部位を挿入する際に、上流側の塩基を自動的に追加する新しいオプションを追加しました。 - クローニングシミュレーション中、挿入が自動的に追加された際にプライマーアノテーションが作成されるようになりました。
- RNA二次構造設定にコマンドラインプレビューを追加しました。
- アガロースゲルのシミュレーション時に使用される式を調整し、より正確な結果を提供するようにしました。
- Collectionsのトップフォルダに、SnapGeneのCollections UIで表示されないコンテンツがある場合に警告が表示されるようになりました。
- コレクション(Collections)機能をデフォルトで非表示にする設定を追加しました。
- 起動ダイアログが「Get Started」になりました。
- マルチセグメントフィーチャーのエンドポイントを調整する際の問題を修正しました。
- ポイントフィーチャの位置を調整できないことがある問題を修正しました。
- プライマービューでプライマーを追加した日付が表示され、文字化けすることがある問題を修正しました。
- Cmd+Wで "Save Unsaved Changes? "ダイアログを閉じないようにしました。
- FASTAインポーターで、コメント以外の行に数字が含まれるファイルを拒否するように改善しました。
- 一部のGeneiousファイルを開くことができない問題を修正しました。
- インポートされた遺伝コードの編集および遺伝コードの複製をサポートするようになりました。
- シーケンスビューで表示されるリーディングフレームを変更したり、Enzymesビューで酵素をソートしても、WindowsやLinuxでは効果がない不具合を修正しました。
- TaKaRa DL500ラダーを追加しました。
- Azura PureView 100 bp DNA Ladderに欠落バンドを追加しました。
Release Note : SnapGene6.2 新機能
バージョン6.2では、準最適RNA二次構造のサポート、Structureビューのその他の機能強化、Golden Gateアッセンブリー シミュレーションツールとPCRシミュレーション全般の改善、Proteinプロパティでの作業時の柔軟性向上などの機能追加・強化がありました。
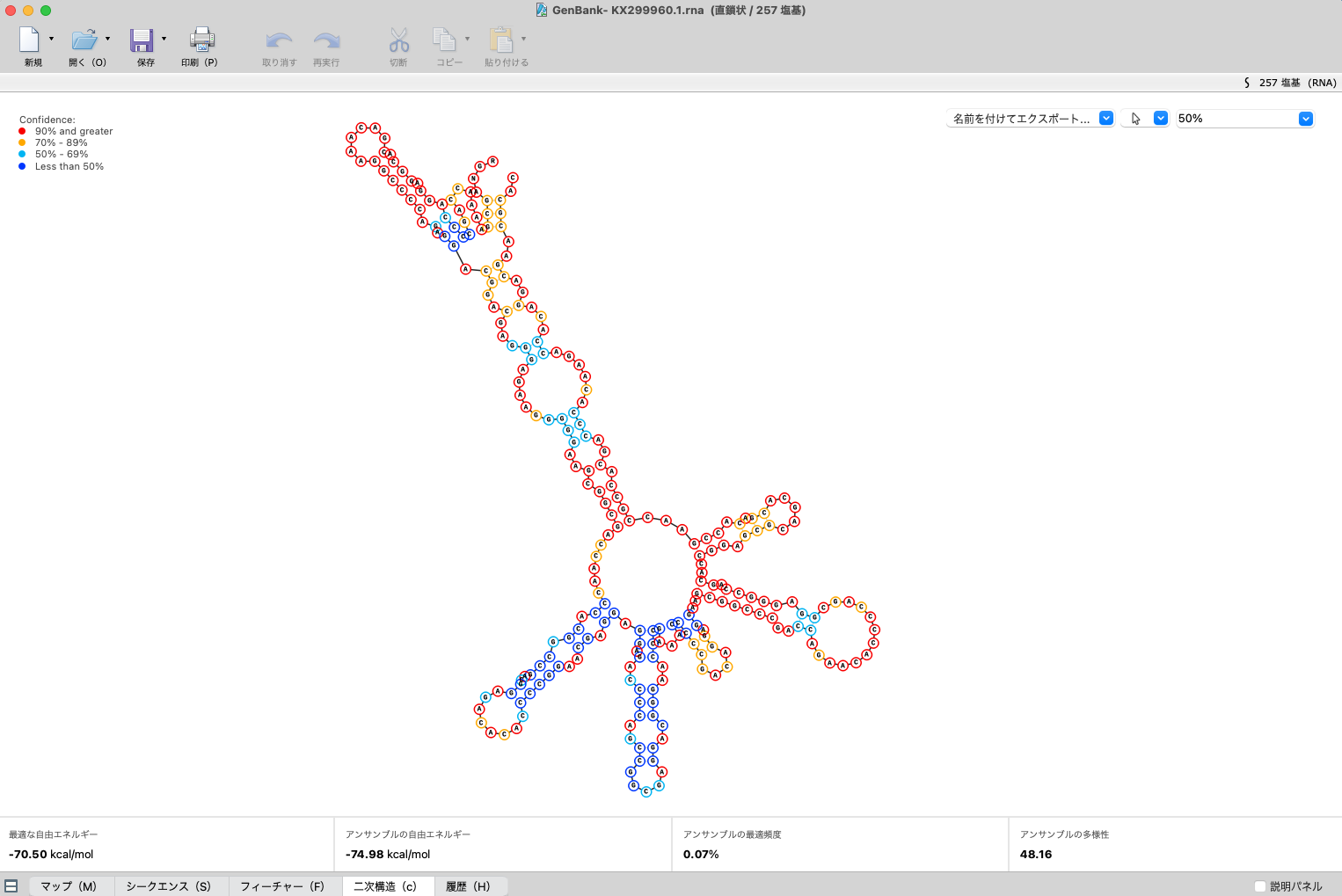
RNA二次構造ビューの機能強化
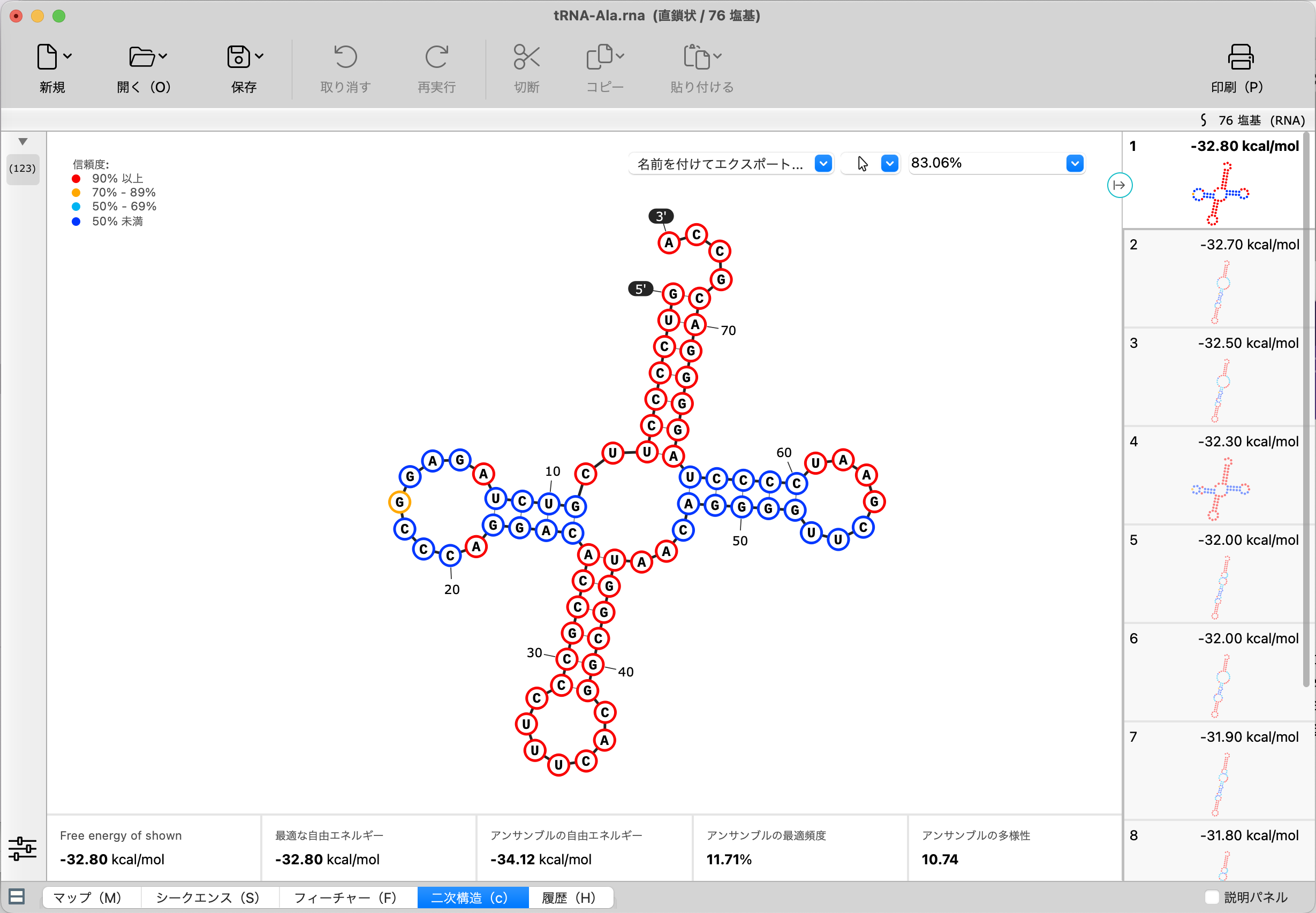
準最適RNA二次構造が表示されるようになりました。すべての構造は、調整された Tm およびその他の設定を使用して再計算されます。可視化および選択機能が強化され、すべてのビューで同期された配列の表示と選択が可能になりました。座標と 5' / 3' 末端のラベルをオプションで表示することができます。詳しくは、SnapGeneラーニング:RNA二次構造予測図 をご覧ください。
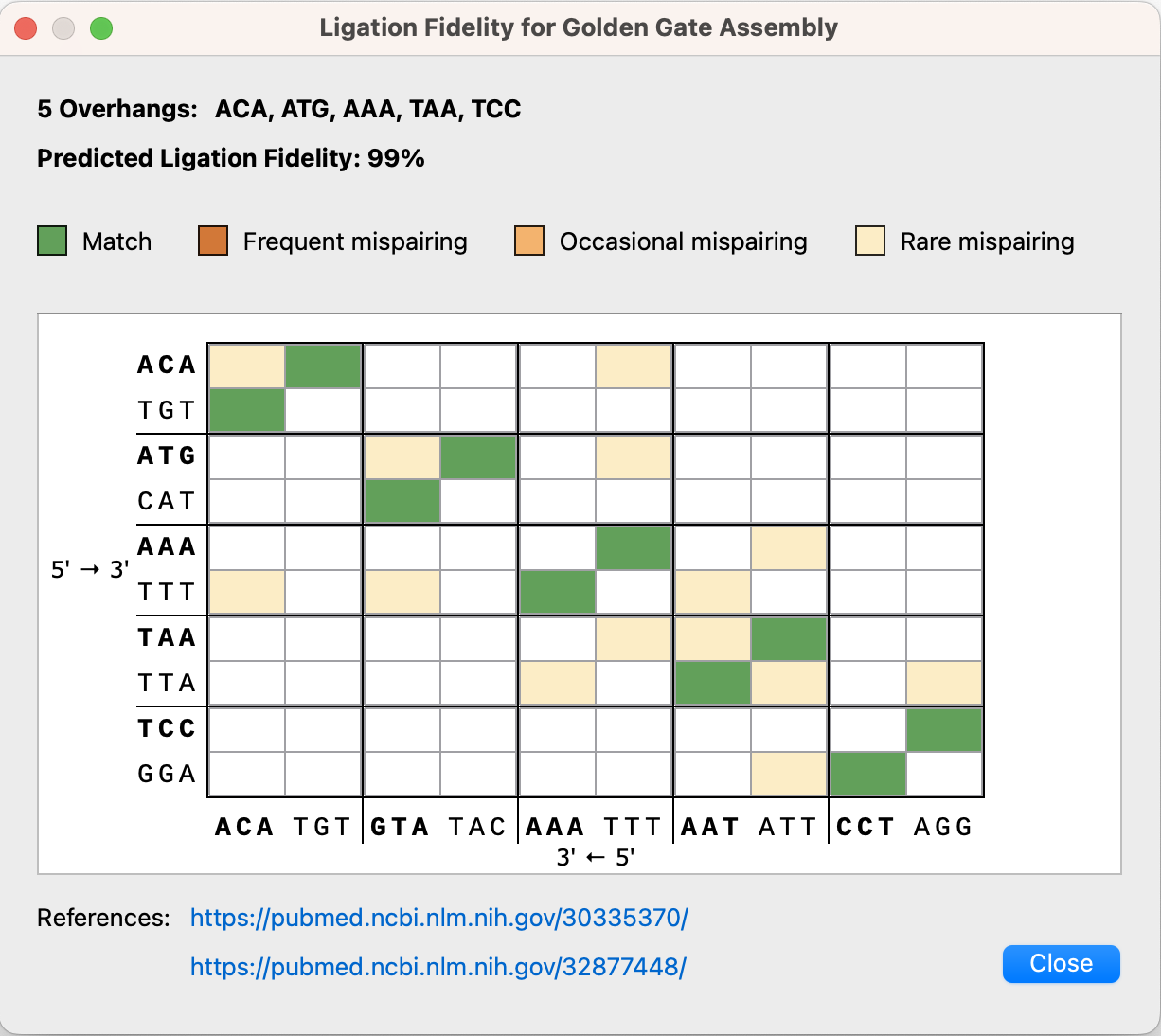
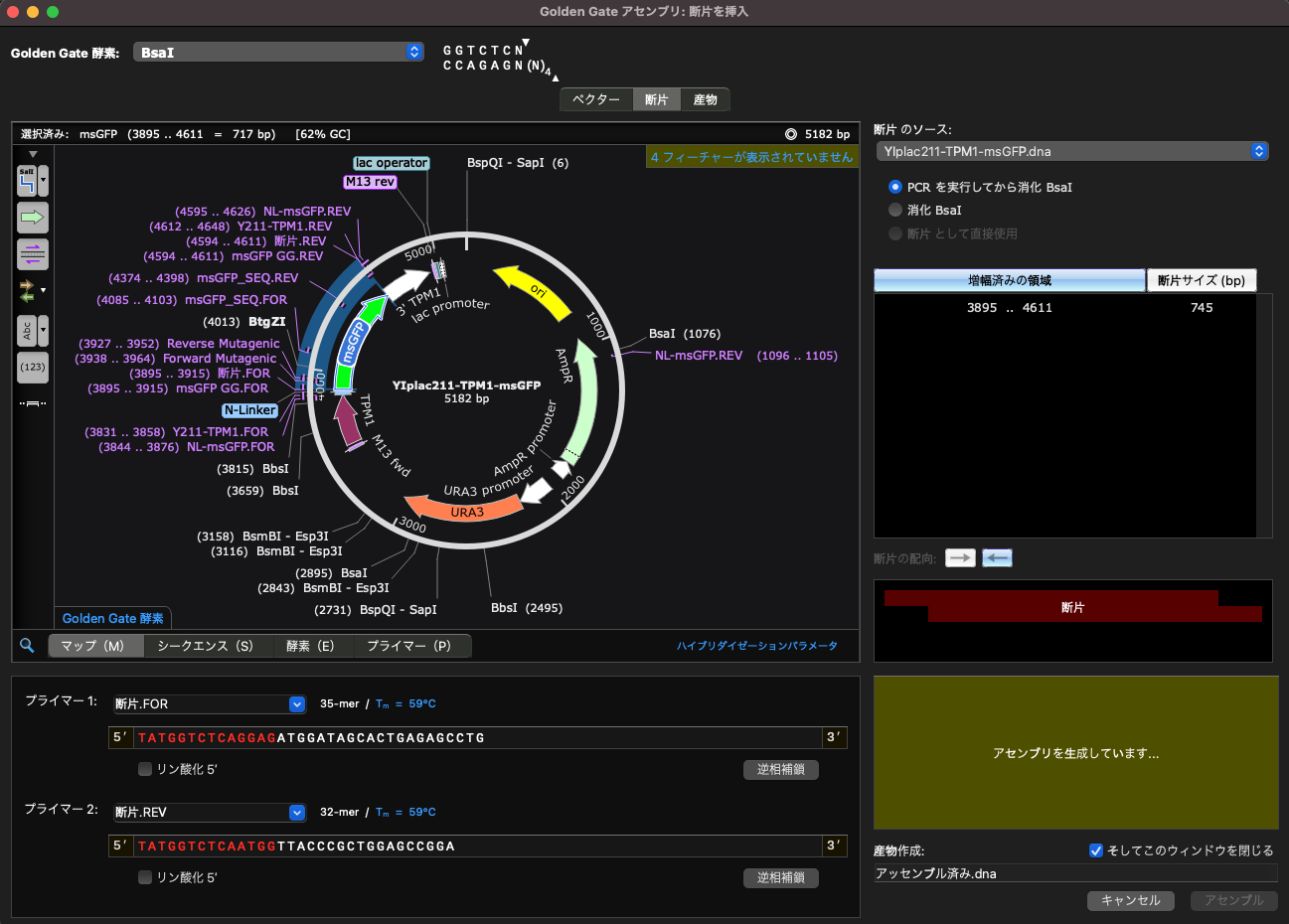
Golden Gate アッセンブリーの改良
Golden Gate アッセンブリーのダイアログの機能が強化されました。ライゲーション フィディリティ マトリックスによる反応全体の忠実度の評価、手動でデザインしたプライマーのオーバーハングの調整、制限酵素を Type IIS に限定せず任意の酵素でベクターをカットできるオプション、推奨された酵素に簡単にアクセスできる設定ダイアログ等が新たに追加されました。
PCRクローニングシミュレーションにおけるプライマー設計の改良
すべてのPCRクローニングシミュレーション(例:PCR、Golden Gate アッセンブリー、Gibson Assembley、In Fusionクローニングなど)において、ハイブリダイゼーション領域の調整ができるようになりました。プライマー自動設計を利用の際、 5’ 末端プライマー伸長が偶然テンプレートと部分的にハイブリダイズした場合に、雑多なフィーチャーが産物に移動しないように領域が自動的に設定されます。

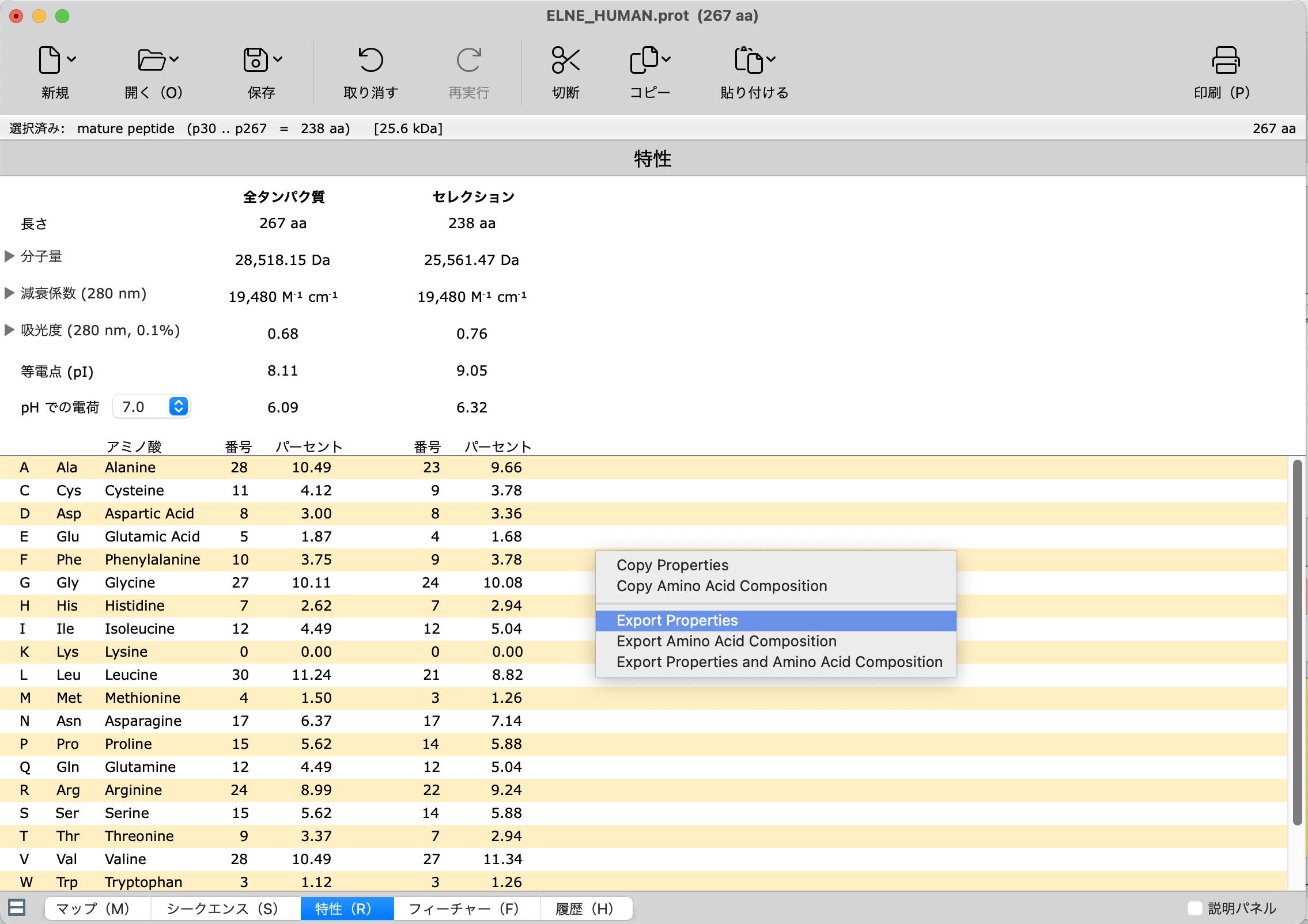
タンパク質のプロパティ(特性)をコピー、エクスポート
タンパク質配列全体または選択した領域の特性およびアミノ酸データを、テキストまたはスプレッドシート(*.csv / *.tsv)形式でコピーまたはエクスポートすることができます。また、それぞれのプロパティをビューから直接コピーすることもできます。
ツールバー、アプリケーション、ドキュメントのアイコンを刷新
トップツールバーにシンプルで分かりやすいなアイコンを採用しました。また、アプリケーションアイコンとファイルアイコンも更新されました。

SnapGene6.2新機能・機能強化詳細
新たに追加された機能
- 準最適RNA二次構造機能を追加しました。
- 調整したTm値などを用いてRNAの構造を再計算することができ流ようになりました。
- Structureビューでの配列の表示と選択が可能になりました。
- Structure viewの右上に、ペイン、回転、スケールをリセットするメニューアクションを追加しました。
- Golden Gate Assemblyツールにライゲーションフィデリティマトリックスを追加し、全体的な反応フィデリティを評価できるようになりました。
- Golden Gate Assemblyベクターを、Type IIS酵素だけではなく任意の酵素で切断できるようになりました。
- Golden Gate Assemblyのシミュレーション中に手動で設計したプライマーのオーバーハングの調整が可能になりました。
- すべてのPCRクローニングシミュレーションのハイブリダイゼーション領域の調整が可能になりました。自動プライマー設計を使用する場合、ハイブリダイゼーション領域は自動的に設定され、偶然に5'プライマー延長がテンプレートに部分的にハイブリダイズしたときに雑多な機能がプロダクトに移行しないようにします。
- タンパク質配列全体または選択した領域の特性およびアミノ酸データをコピーまたはエクスポートすることができます。
強化された機能
- 隣接する2つのフラグメントが直接使用されるように設定され、隣接する末端が両方とも5'末端リン酸塩を持たない場合、ゴールデンゲート集合のシミュレーションをチェックし、防止します。
- 低DPIディスプレイにおける多くのアイコンの見栄えを改善
タンパク質アラインメントの選択バーに選択されたMWを表示するようにしました。 - 5'末端リン酸塩を欠く線状フラグメントをライゲーションする際に表示されるエラーメッセージを改善しました。
- Golden Gate Assemblyダイアログを更新し、推奨酵素に簡単にアクセスできるようになりました。
- FASTA エンコードされた配列を新規ファイル作成ダイアログに貼り付ける際のデフォルトの配列名を改良しました。
- アプリケーション、ファイル、その他様々なアイコンを、シンプルでより分かりやすいアイコンに変更しました。
- ゴールデンゲートクローニングのミニ・オーバービューにおいて、隣接するオーバーハングの相補的塩基を表示するように変更しました。
Release Note : SnapGene6.1 新機能
バージョン6.1では、Golden Gateアッセンブリーのシミュレーション時のプライマー設計を簡単にしました。また、ssRNA配列の新しい二次構造ビューが搭載されています。
Golden Gate アッセンブリー
SnapGene 6.1では、Type IIS制限部位を適切に配置したPCRプライマーの自動設計や、Golden Gate アッセンブリーのシミュレーションを行うツールが提供されています。Golden Gateのプライマーとオーバーハングを自動的に設計し、反応の忠実度を最適化することで、成功の可能性を最大限に高めることができる新しいツールです。
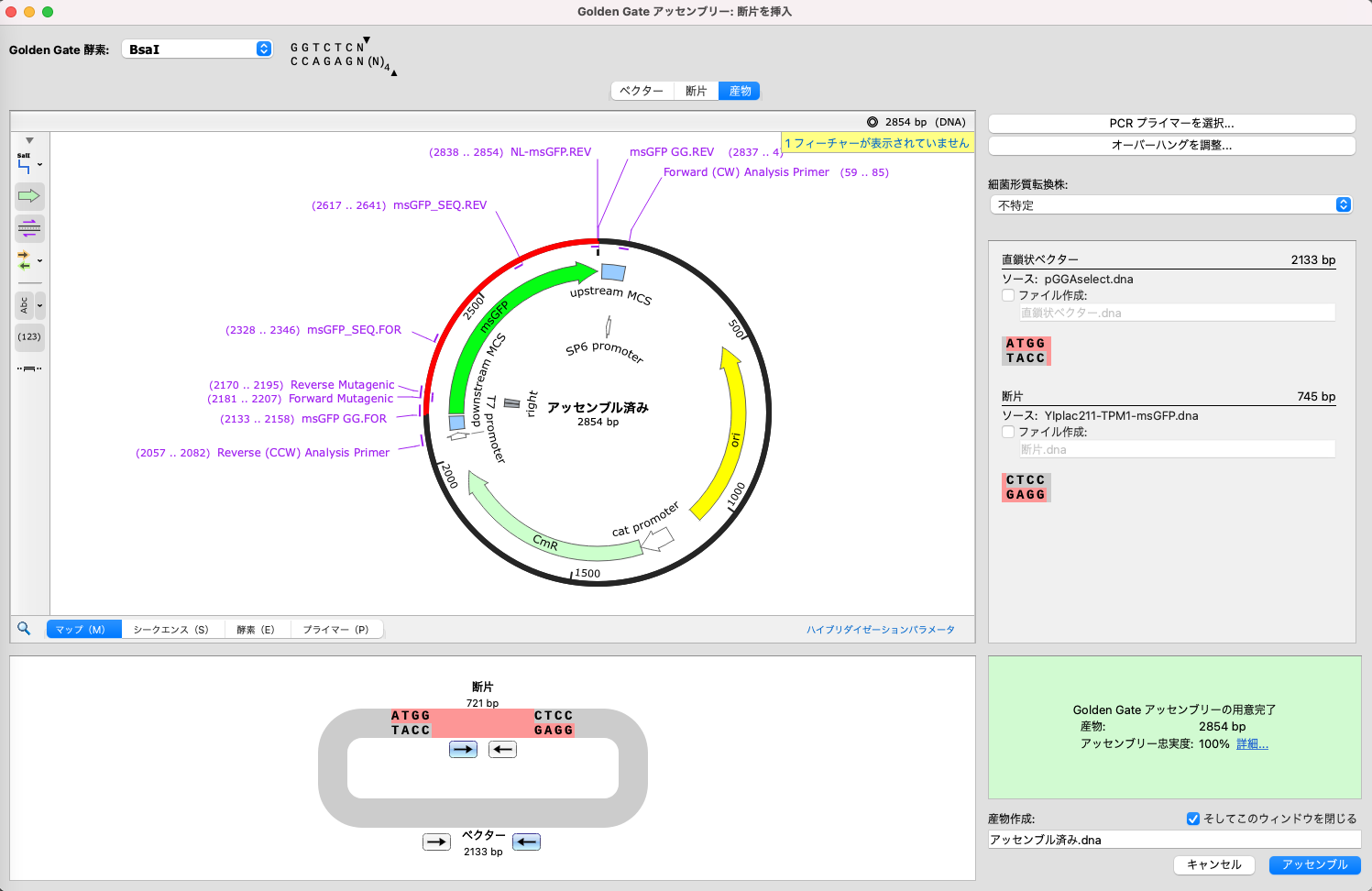
How is Golden Gate Fidelity Predicted in SnapGene?
RNA二次構造
SnapGene6.1では、ViennaRNA を使って一本鎖RNA配列(ssRNA)二次構造を計算します。「二次構造ビュー」では、ViennaRNA で計算された最適な構造予測図が表示され、一本鎖RNA配列がどのように折り畳まれるかを視覚化します。SnapGene は、3000塩基までのssRNA配列の二次構造の予測を表示することができます。
ダークモード
ユーザーインターフェイスがダークモードに対応し、デフォルトで OS のダークモードまたはライトモードの設定に合わせられるようになりました。
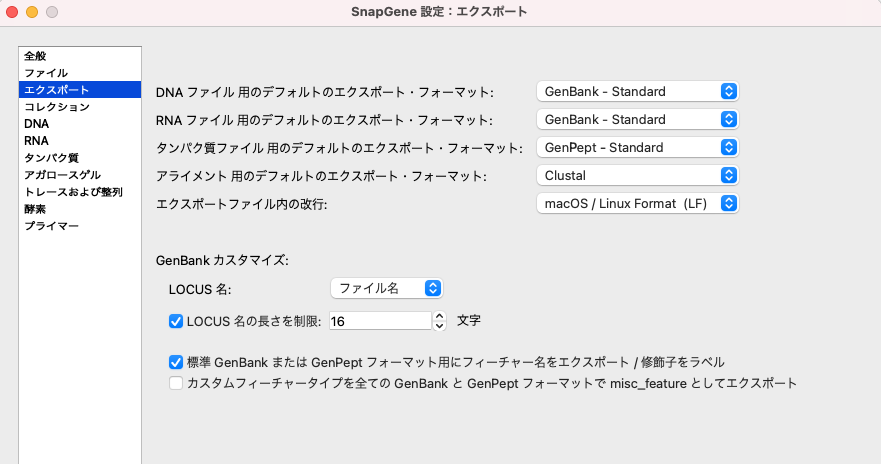
エクスポート機能
環境設定のエクスポートパネルで、LOCUSフィールド識別子フィーチャーのエクスポートオプションなど、GenBankへのコンテンツのエクスポート方法をカスタマイズできます。
Release Note : SnapGene6 新機能
What's New in SnapGene | SnapGene6に追加された機能
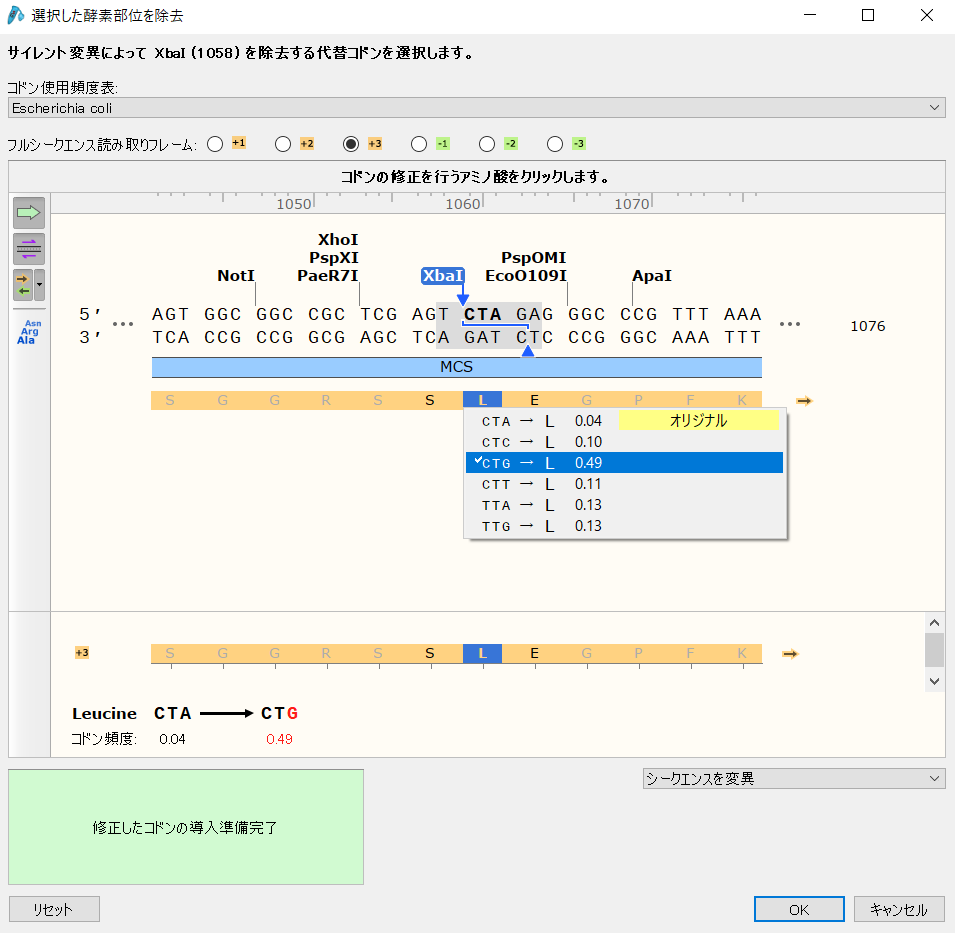
- SnapGene5では、サイレント変異を導入する場合は教科書などに記載されているコドン表を見ながら入力する必要がありました。しかし、SnapGene6ではその候補をコドン使用率とともに提示してくれます。これにより置換候補の優先順位をつけることが容易になりました。
- SnapGene6では、フィーチャータイプのカスタム編集が可能になりました。これにより実験者の好みや表現の幅に沿った配列編集が可能になりました。
- 配列の一致度を確認するペアワイズアラインメントでもフィーチャーの表示や編集ができるようになり、アライメント後の画面も見やすくなりました。これにより、任意のフィーチャーごとの比較がより簡便かつ迅速にできるようになりました。
- クローニングのシミュレーション機能では、フラグメントの追加、削除、順序変更ができるようになり、複数のフラグメントを連続挿入のシミュレーションがより扱いやすくなりました。これにより融合タンパク質のコンストラクト作成やプロモーター領域の改良コンストラクト作成などのシミュレーションがより容易になりました。
サイレント変異導入による制限酵素サイトの追加または削除
コーディング配列からサイレント変異を導入できるようになりました。これにより、制限酵素サイトの追加または削除がより簡便になりました。
新しいフィーチャータイプの追加
これまで使用可能だったフィーチャータイプは、GenBankで使用されているものに限定していました。今回のアップデートでは、GenBank以外のフィーチャータイプも追加しています。また、オリジナルなフィーチャータイプを追加することもできます。そして、それらのフィーチャーをインポート・エクスポートして、他の人や他のコンピュータと共有することも可能です。さらに、デフォルトのフィーチャーカラーが変更できるようになりました。
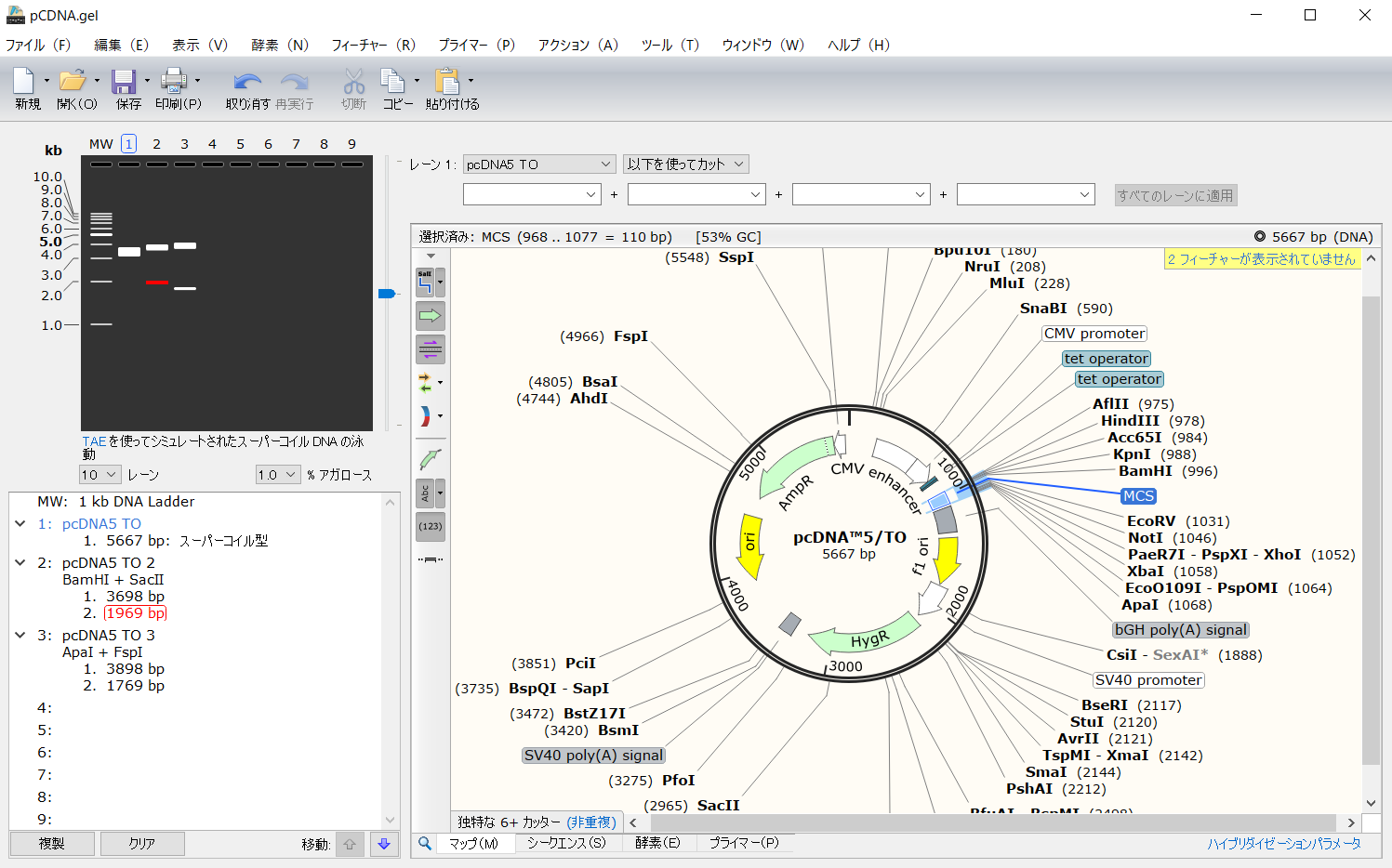
アガロースゲルファイルの登場
アガロースゲルのシミュレーション結果をファイルとして保存することができるようになりました。拡張子は “.gel” です。実際のゲルを見た後に編集を加えることが可能ですし、さらに他の人と共有することも可能です。
アラインメントにおけるフィーチャー編集機能の追加
アライメントでフィーチャーを表示する機能は、マルチプルシーケンスアラインメントでのみでした。今回のアップデートでは、ペアワイズアラインメントでもフィーチャーの表示ができるようになりました。また、フィーチャーの編集も可能となっています。追加・編集・検索は、アライメントビューまたは新規のフィーチャービューのどちらからでも可能です。
クローニングシミュレーション機能の強化
これまで、複数のフラグメントを連続してベクターに挿入する時は、コンストラクトどこに何を追加するかを決める必要があり順序変更などはできませんでした。また、ミスを修正するためには編集画面を閉じて最初からやり直す必要もありました。今回のアップデートでは、フラグメントの追加、削除、順序変更ができるようになり、複数のフラグメントを連続挿入がより使いやすくなりました。また、ベクターを反転させる機能も追加されています。
SnapGene6新機能・機能強化詳細
新たに追加された機能
- サイレント変異によるコーディング配列に酵素サイトを追加するツールを追加しました。
- サイレント変異によるコーディング配列から酵素サイトを削除するツールを追加しました。
- GenBankフィーチャータイプの標準セットに含まれていないカスタムフィーチャータイプに対応しました。
- 標準的なGenbankフィーチャータイプのデフォルトカラーの変更を可能にしました。
- アガロースゲルのシミュレーションを.gelファイルとして保存・読み込みできるようになりました。
- ペアワイズアラインメント内のフィーチャーのサポートを追加しました。
- アラインメント内のフィーチャーを追加、編集、削除できるようになりました。
- ペアワイズおよびマルチプルシーケンスアラインメントの表示時にフィーチャーテーブルを追加しました。
- アラインメントを検索してフィーチャーを見つけることができるようになりました。
- Gibson, InFusion, NEBuilder HiFi Assemblyのシミュレーションツールが強化され、ベクターを反転できるようになりました。
- クローニングシミュレーションツールが強化され、配列の追加、削除、並び替えに対応します。
- 酵素の認識配列に対する切断回数と相対的な位置に基づいて、選択された酵素のセットをフィルタリングするコントロールが含まれています。
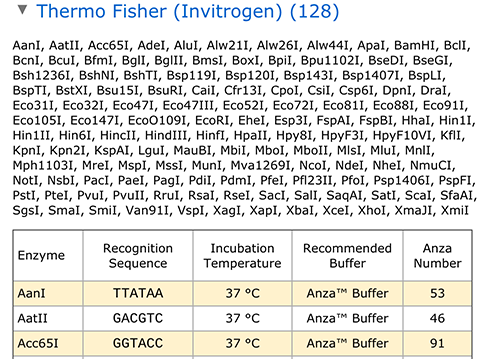
- 11種類の制限酵素に対応しました。AgsI、ArsI、BssSI、BsuI、BtsI、HpySE526I、LmnI、LpnPI、Mox20I、PstNI、TseFIの11種類の制限酵素に対応しました。
- デフォルトのCodon Usage Tableを設定できるようになりました。
- DNA Calculationsダイアログに、ng/uLをnMに変換するツールを追加しました。
- DNA Calculationsダイアログに塩基数を追加しました。
強化された機能
- "Blocks of 3"シーケンスビューのフォーマットを改良し、ヌクレオチドトリプレットをリーディングフレームに合わせられるようにしました。
- RNA配列のインポートまたは新規作成時にPsiをUに変換するようになりました。
- ペアワイズおよび多重整列を印刷する際に、オプションで配列名を配列と一緒に表示することをサポートします。
- Gatewayクローニング用ベクターpEXP3-DESTとpEXP4-DESTを追加し、pEXP-DEST Vectorsの説明の相互参照を更新しました。
- 32の酵素のコメントを更新しました(複数のサイトを持つことが必須ではないものの、これによって結果的に切断が促進されます):AarI, AciI, AcuI, AloI, BaeI, BbeI, BfiI, Bpu10I, BsaWI, BsaXI, BsmFI, BsrFI, BveI, Cfr9I, Cfr10I, Cfr42I, CseI、EarI、Eco57I、EcoRII、FaqI、HgaI、HpaII、LweI、Mly113I、PdiI、PpiI、SatI、Sau3AI、SfaNI、SmlI、XmaI
- 編集中のファイルを、他のSnapGeneインスタンスに対して読み取り専用に設定するロック機構を追加しました。ただし、ロック機構は大きなファイルでは使用できず、遅延時間も短いため、ファイルを同時に開いた場合には効果がありません。
- New Protein Fileにコピー&ペーストしたり、タンパク質配列中の残基を挿入または置換しても、機能やカスタム番号が保持されるようになりました。
- プライマーデータをエクスポートする際に、結合部位を含めることができるようになりました。
- コピーした相補的なプライマーペアをペーストしてアガロースゲルのレーンを構成する機能を追加しました。
- Restriction Cloning中に酵素がブロックされた場合に配列のメチル化を変更する方法の説明を改善しました。
- 曖昧なアミノ酸を表現するための訳語を"?"から "X/Xaa "に変更しました。
- ssDNA配列において、選択した領域のTmを追加しました。
- コマンドラインインターフェースからマップをエクスポートする際に、解像度(ppi)の調整と透過画像の作成に対応しました。
修正箇所
- タイプIISの酵素セットにTaqIIを追加しました。
- 6+ Cutters酵素セットにニッキング酵素を追加しました。
- TaqIIの二次認識配列は切断につながらないため削除しました。
- 酵素BmeT110IおよびBmgT120Iの切断位置を修正しました。
- Restriction Enzymes ウィンドウで以前表示していた酵素に戻る際に、最後に表示していた酵素のバリアントを復元します。
- 大規模な祖先配列のHistoryビューのフィーチャーを省略することで、ファイルサイズを削減し、ロードを高速化しました。
- すべての翻訳可能なフィーチャータイプに対して、次の非標準修飾子のサポートを追加しました:calculated_mol_wt、codon、codon_start、exception、protein_id、transl_except、transl_table、translation
- 非標準の修飾子が、インポートおよびエクスポート時に保持されるようになりました(以前は /note に変換されていました)。
- タンパク質配列中のCDSフィーチャーで、/transl_tableおよび/codon_start修飾子のサポートを追加しました。
修正箇所
- 複数のフィーチャーを選択した場合に、そのフィーチャーが介在すると「Copy Amino Acids」や「Make Protein」が失敗する問題を修正しました。
- macOSで、空のSnapGeneウィンドウが表示され、SnapGeneを再起動しないと閉じることができないことがある問題を修正しました。
- 環境設定で、補助的なチェックボックスを無効にするようにしました。
- 複数のセグメントを持つSite featureのマウスオーバー効果を修正しました。
- protein point feature typeをmisc_feature、unsure、variationに設定できない問題を修正しました。
- 点状フィーチャーを追加する際にタイプを変更すると、色が手動で調整されていない場合、フィーチャーの色が自動的に変更されるようになりました。
- 直鎖状のDNA配列のどちらかの端を共有結合で閉じるように修正した場合、UがTに変換されない問題を修正しました。
- 株式会社ニッポンジーンの酵素のリンクを修正しました。
- 配列調整ツールで追加された未保存のファイルが誤った名前でリストアップされる問題を修正しました。
- アラインメントでランオンの翻訳が表示されないことがある問題を修正しました。
- DNAファイルで最後の塩基までスクロールできるようになりました。
- 一部のタンパク質ファイルでfeaturesタブを開いたときに表示されていた不適切なメチル化メッセージを削除しました。
- Choose Alternative Codons "ツールを使用した際に、プライアンシーが表示されない問題を修正しました。
- アガロースゲルのシミュレーションでレーンをMWマーカー使用に切り替えた際に表示される様々な問題を修正しました。
- タンパク質配列にポイントフィーチャーが追加されない問題を修正しました。
- Mutagenesisツールを使用する際、プロダクト名の指定を必須とするようにしました。
- ヒストリーカラーが表示されている場合、塩基が常に表示されるようにしました。
- シーケンスビューで、隣接する翻訳済みフィーチャーの共有コドンを常に表示するように修正しました。
- Align to Reference DNA sequence(リファレンスDNAシークエンスへのアライン)を修正し、配列の編集を元に戻すと、アラインされた配列が非表示にならないようにしました。
- 環状配列において、数値原点に巻きついたORFを確実に表示するように修正しました。
- Linuxで起動ダイアログが表示されているときに、一部のキーボードショートカットが動作しない問題を修正しました。
- この環境設定がオンになっている場合、デフォルトで説明パネルが正しく表示されるようになりました。
- Choose Alternative Codons(代替コドンを選択)ツールのナビゲーションボタンが正しく有効になるように修正しました。
- Region featureが表示されていない場合に、タンパク質配列のFeatureを検索する際の問題を修正しました。
- Features ビューでタブを切り替えると、修飾子の選択が失われるという問題を修正しました。
- 参照配列へのアラインメントを表示中にアンドゥを使用すると、展開されたアラインメント配列が折りたたまれてしまう問題を修正しました。
- 一部の円形配列を線形化できない問題を修正しました。
- プライマーをインポートする際、ウィンドウ上のチェックボックスの選択が遅れることがあった問題を修正しました。
- Next/Previous Aligned Region ボタンの位置を修正し、原点を越えて次の/前の領域にジャンプできるようにしました。
- Vector NTI Expressのデータベースインポートにおいて、Java 8がサポートされていることを示すダイアログを明確にしました。
- Preferences(環境設定) の Files(ファイル)のデフォルトフォルダが、Open Files(ファイルを開く)で無視される問題を修正しました。
- プライマーペアを選択して新規ファイルを作成する際、デフォルトのドキュメント名にプライマー名が含まれていなかった問題を修正しました。
- NEBuilderツールで50bp以下の間隔でプライマーを使用できない問題を修正しました。
- Import SnapGene Online Sequences ツールを使用する際、検索コントロールをクリアした後に配列リストを更新する際の問題を修正しました。
- NEBuilderツールを使用する際に、ベクターに対して既存の重複しないPCRプライマーを使用できない問題を修正しました。
- Agarose Gel (アガロースゲル)ウィンドウにファイルをドラッグ&ドロップできない問題を修正しました。
- コレクション内のドキュメントを切り替えたときに、選択した酵素が表示されない問題を修正しました。
- Edit References (リファレンスの編集)ウィンドウで参考文献を削除した後に残る不要な横線を削除しました。
- Edit References (リファレンスの編集)ウィンドウのデフォルトサイズの改善しました。
- 粘着性オーバーハングを持つ選択範囲の長さを表示する際の問題を修正しました。
- トリミングハンドルをドラッグする際の安定性が向上しました。
- Insert Codon and Choose Alternative Codons (コドンの挿入と選択)ツールを使用した際に、曖昧なコドンを削除しました。