Golden Gate アッセンブリーをシミュレーション
SnapGene 6.1より Golden Gate アッセンブリーのシミュレーション機能が追加されました。
Golden Gate アッセンブリーは Type IIS 制限酵素を用いたクローニング方法で、余分な配列を含まずシームレスにクローニングを行うことが可能です。このチュートリアルでは、SnapGeneで Golden Gate アッセンブリーをシミュレーションする方法をご紹介します。
参照ニュー・イングランド・バイオラボ・ジャパン株式会社による NEB Golden Gate Assembly Kit (BsaI-HF v2) ページ
https://www.nebj.jp/products/detail/2097
1.【準備】DNA配列を入手する
SnapGeneは、NCBI や Addgene のデータベースから配列をインポートすることが可能です。このチュートリアルでは、SnapGeneオンラインシークエンスから pGGASelect をインポートしてデータを読み込みます。
また、本チュートリアルで使用する YIplac211-TPM1-msGFP.dna のファイルはこちらよりダウンロード可能です。
1-1. データをインポートする
SnapGeneを起動し、「インポート」から「SnapGene オンラインシークエンス」を選択します。
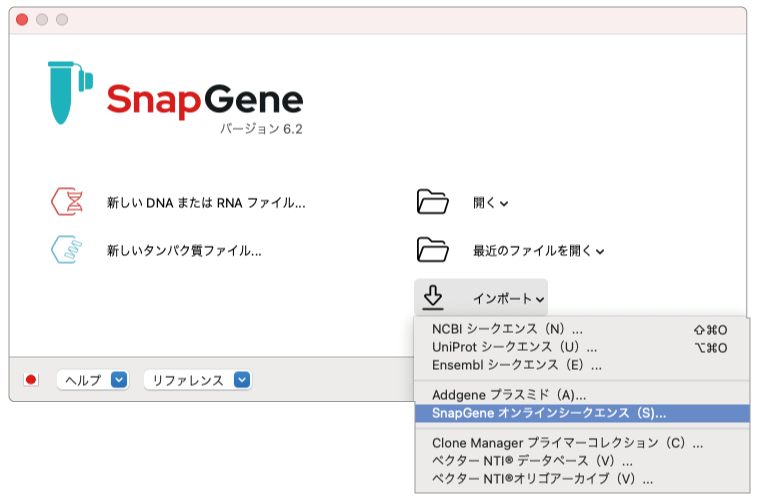
ダイアログの検索フィールドに pGGASelect と入力し検索を開始します。検索結果に表示された pGGASelect を選択し、「インポート」ボタンをクリックします。
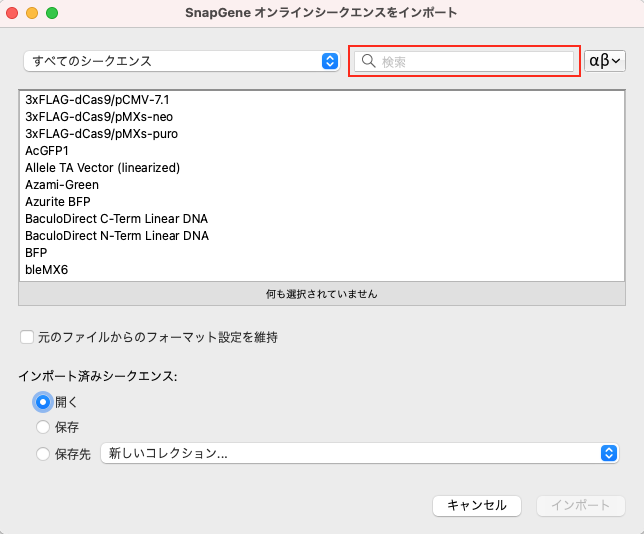
ファイルの読み込みが完了と同時にファイルが開きます。
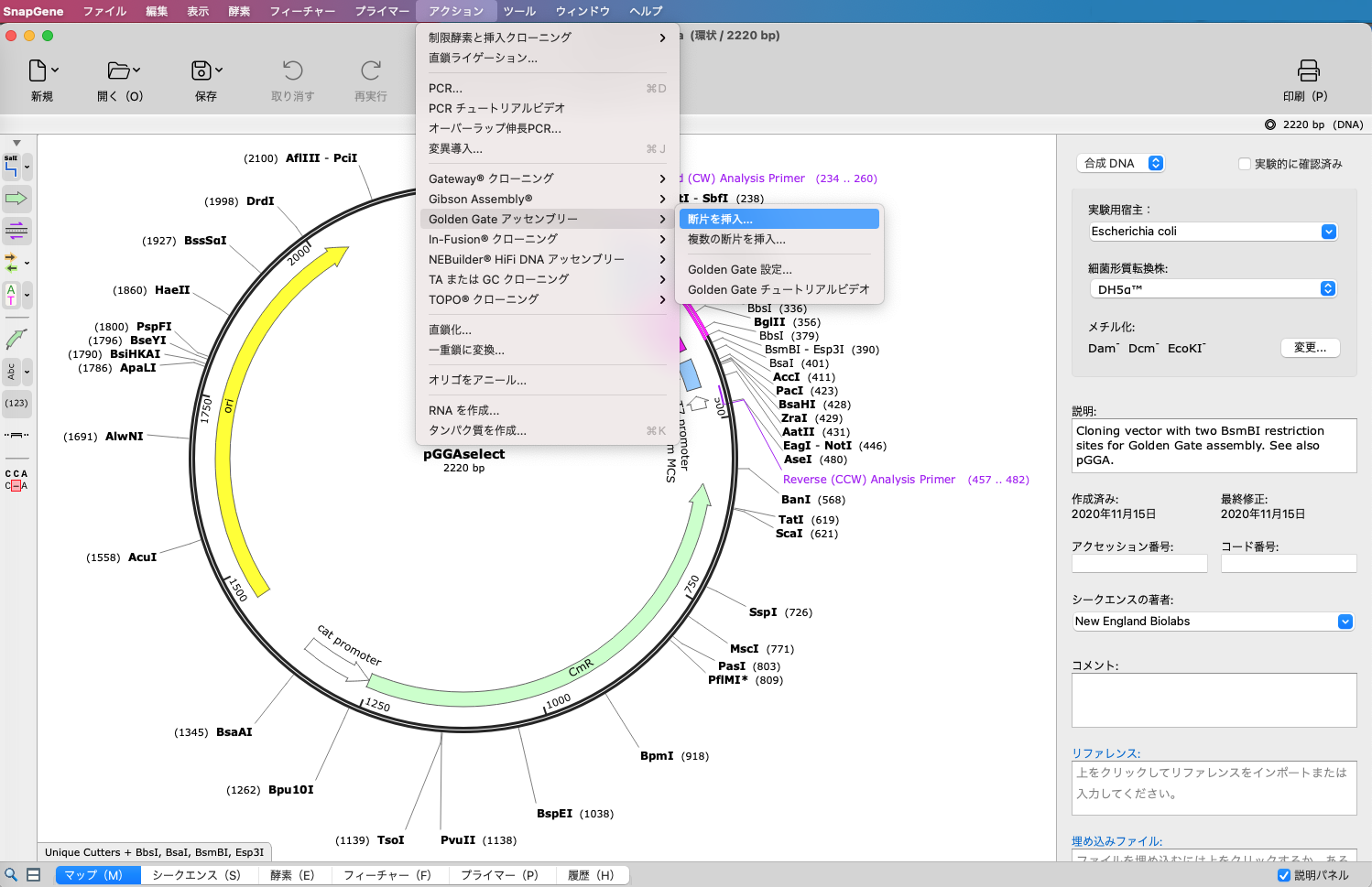
2. Golden Gate アッセンブリーをシミュレーション
ツールバーの 「アクション」 → 「Golden Gate アッセンブリー」 → 「断片を挿入…」 をクリックし、作業ウィンドウを開きます。

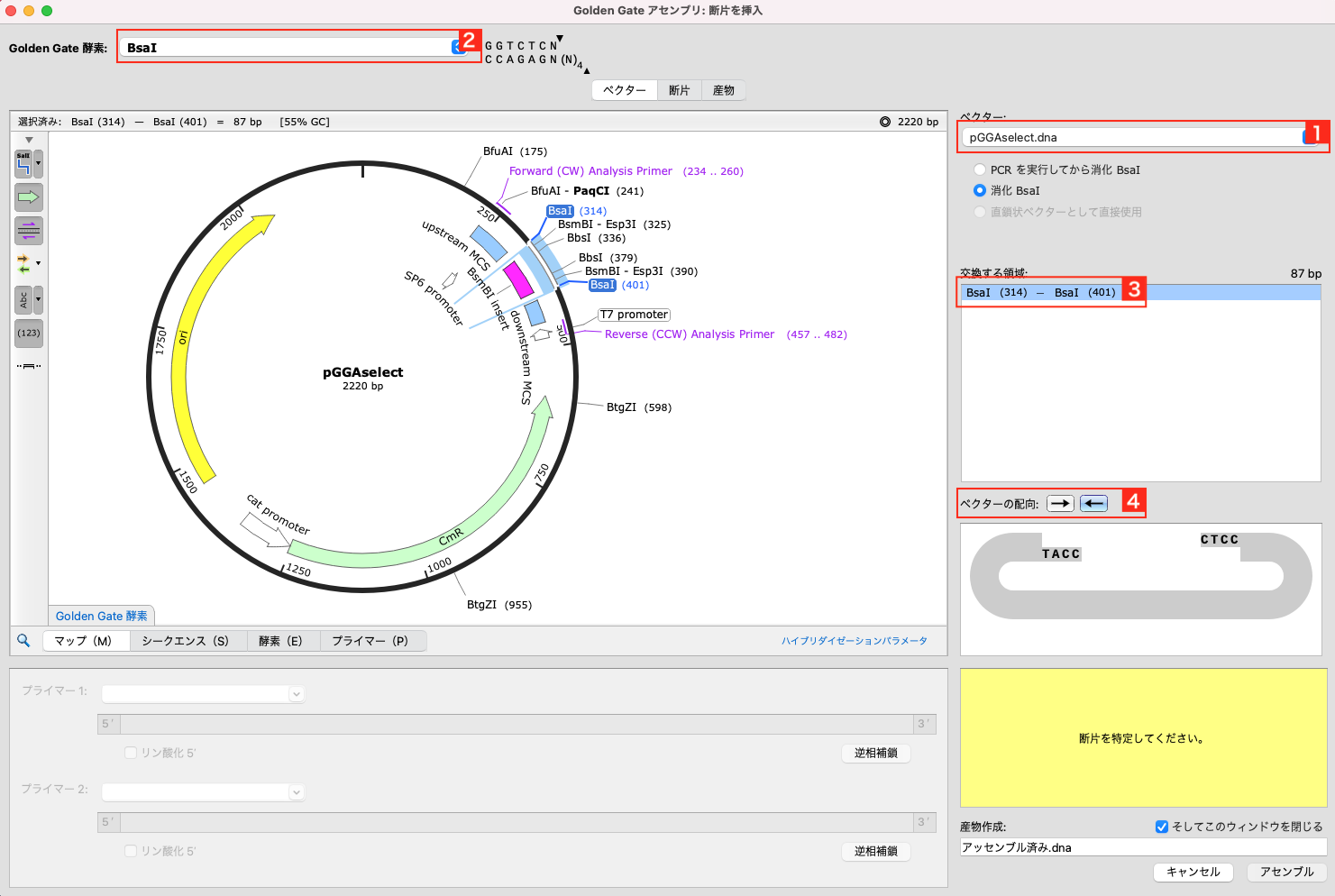
1pGGASelect のベクターが選択されています。もし、pGGASelect が選択されていない場合は、ドロップダウンリストから選択してください。
2デフォルトでは、Type IIS 制限酵素として BsaI が選択されています。別のタイプIIS制限酵素を使用する場合は、ドロップダウンリストから変更してください。 ファイルをダウンロードしても SnapGene で読み込んでいない場合、ドロップダウンリストには表示されません。リストに表示されない場合、ドロップダウンリストの一番下にある「ブラウズ...」からファイルを指定し開きます。
3インサートして置換する領域が自動的に選択されます。
4ベクターの向きを変更する場合は「ベクターの配向」で指定します。
2-1. 断片タブ
断片タブに切り替えます。ソースファイル(YIplac211-TPM1-msGFP.dna) をドラッグ&ドロップするか、「断片のソース」のドロップダウンから、ファイルを選択します。
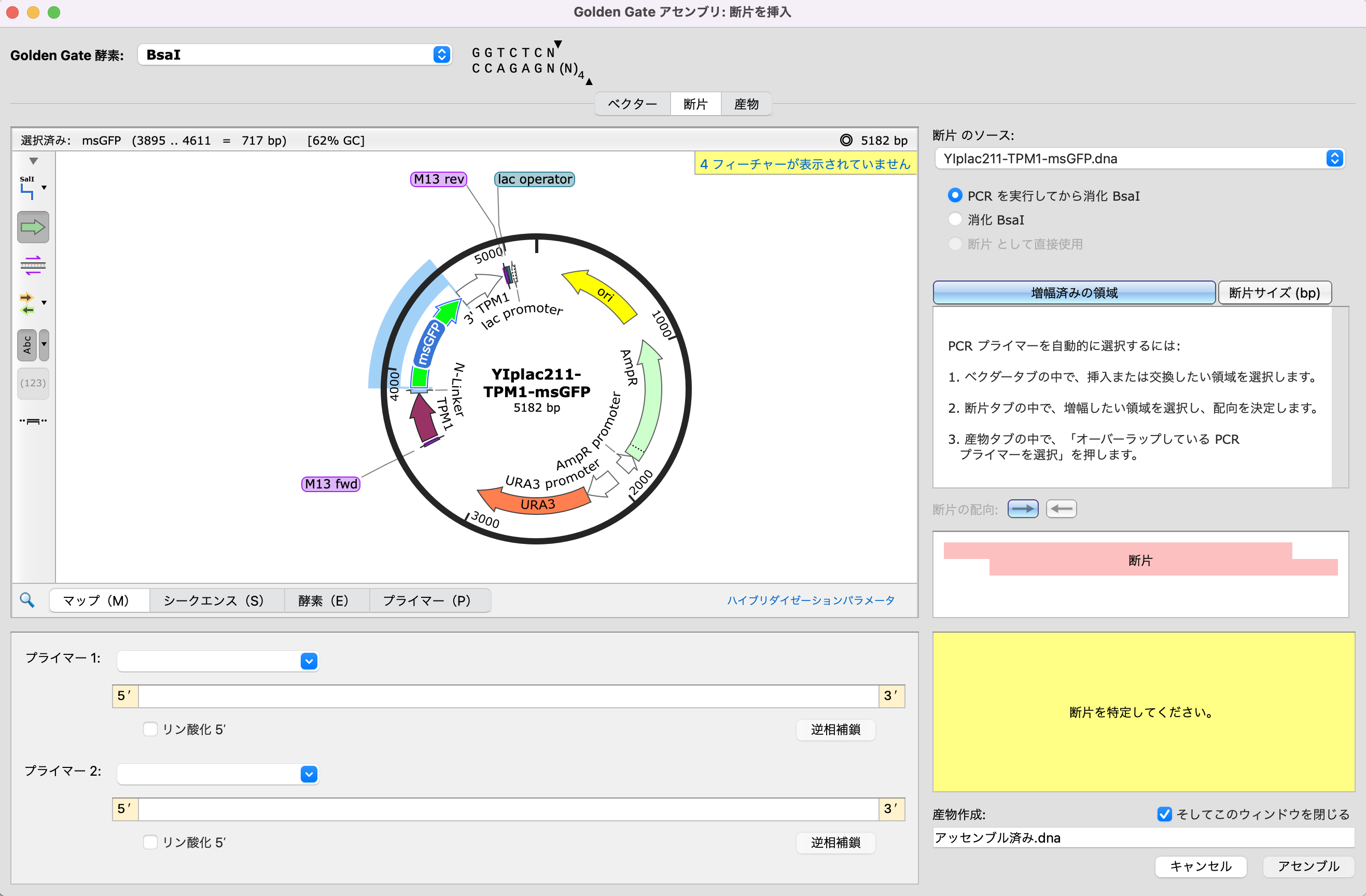
マップビューで、クローンを作成するフィーチャー(ここでは、msGFP)をクリックして選択します。または、シークエンスビューに切り替えて挿入する領域を選択します。
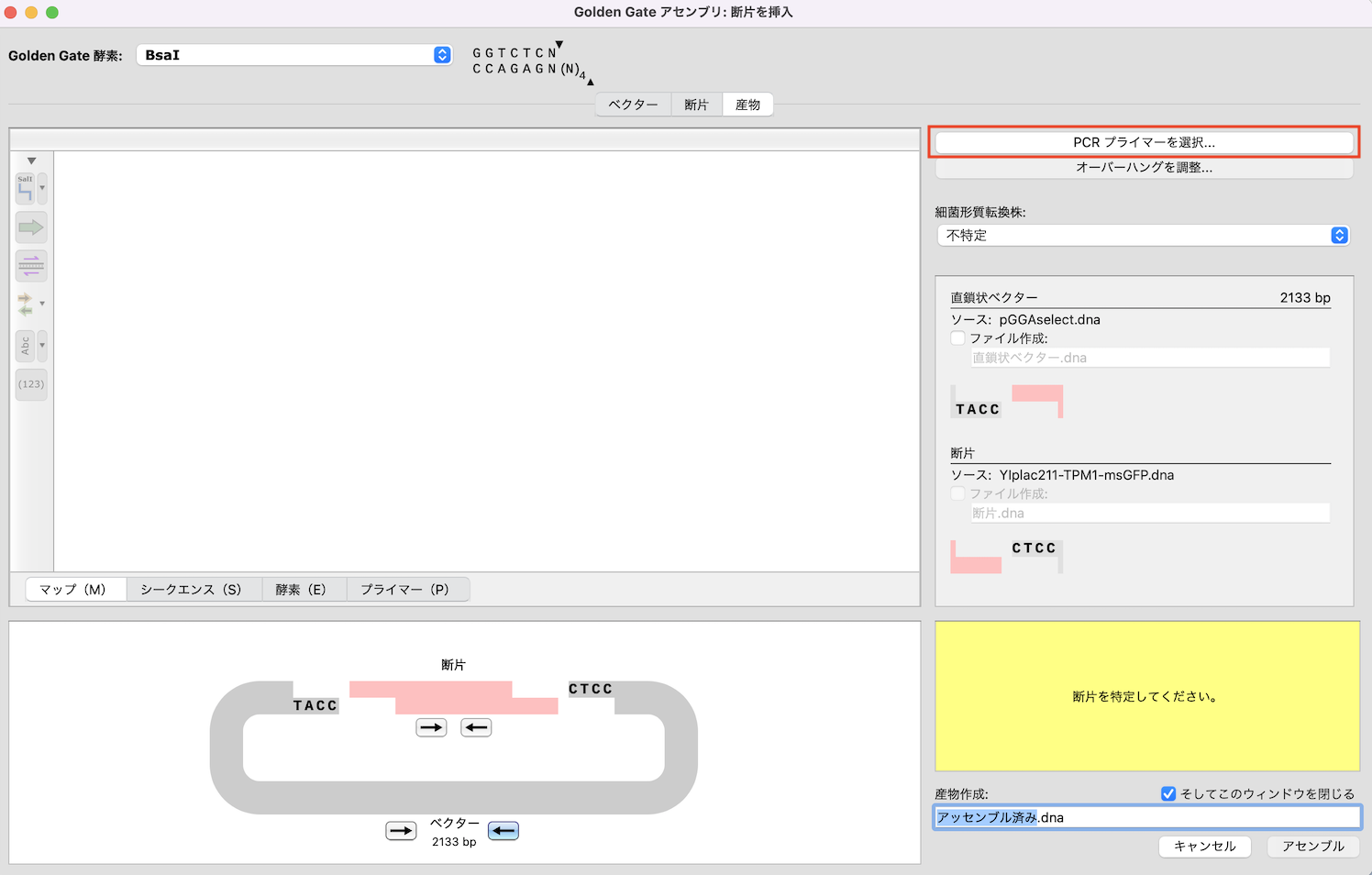
2-2. 産物タブ
産物タブを開き、「PCRプライマーを選択...」をクリックします。
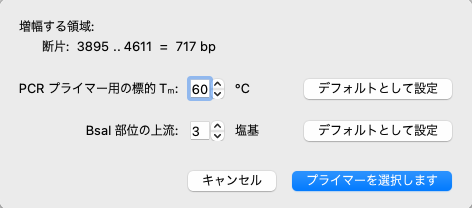
使用しているPCR酵素に応じてプライマーのTm値を設定し、Type IISサイトの 5' に追加する塩基の数を設定します。設定が終わったら、「プライマーを選択します」をクリックします。
産物タブにクローニング後のプラスミドマップが作成されます。
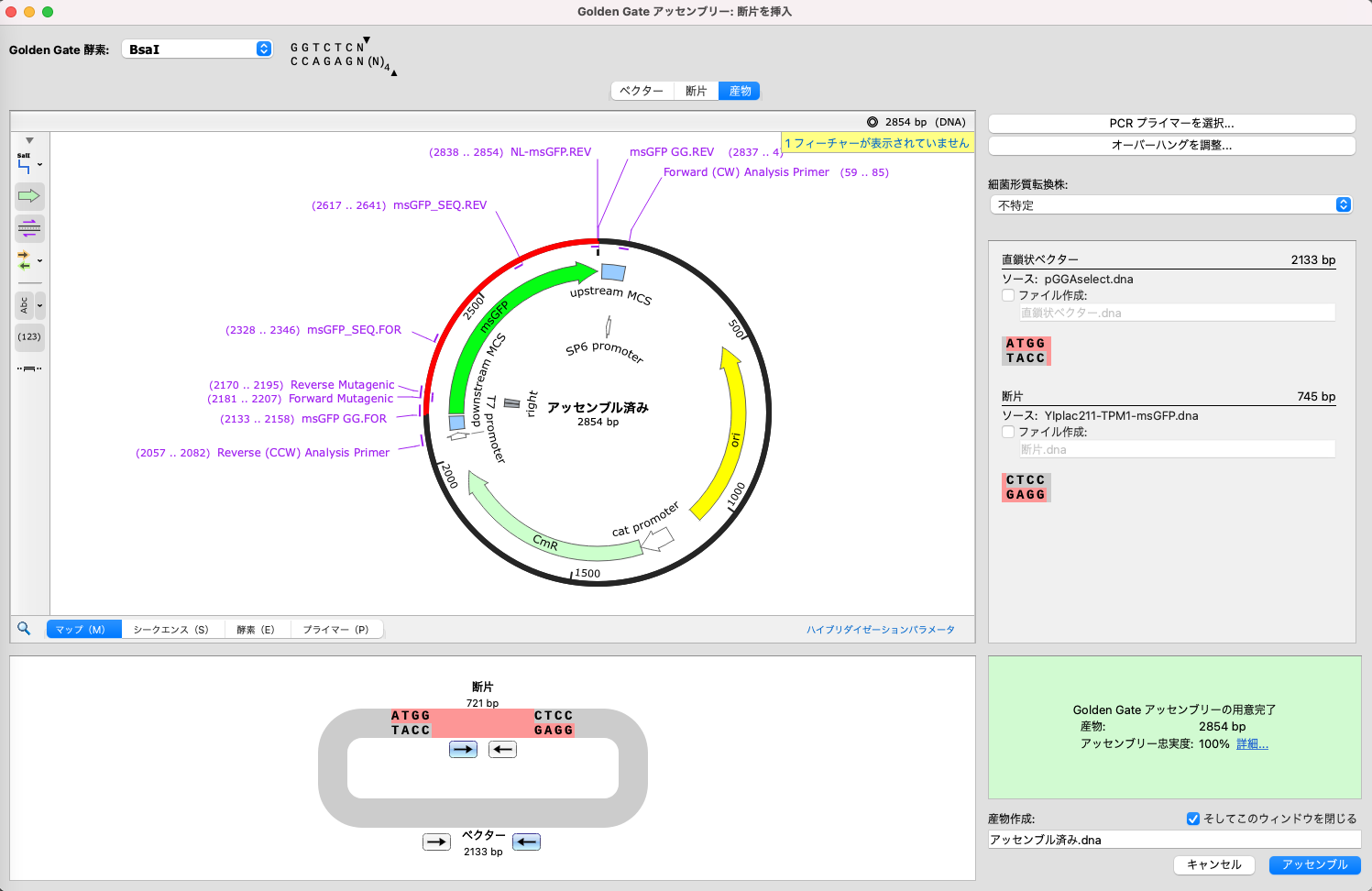
新たに作成されたPCRプライマーはデフォルトで「断片.FOR」「断片.REV」と名付けられます。断片タブに切り替えて、新たに作成したプライマーに名前を変更します。
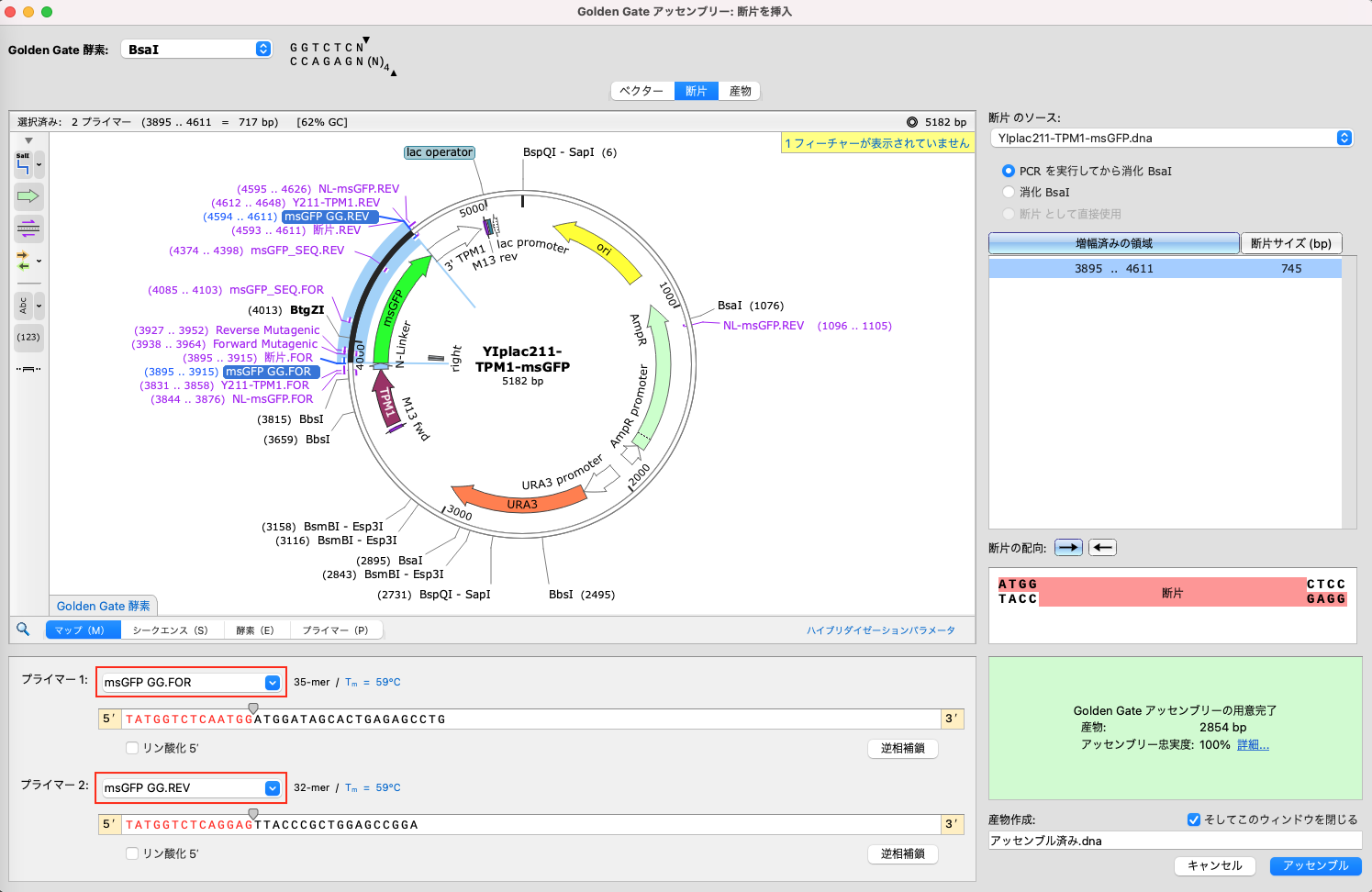
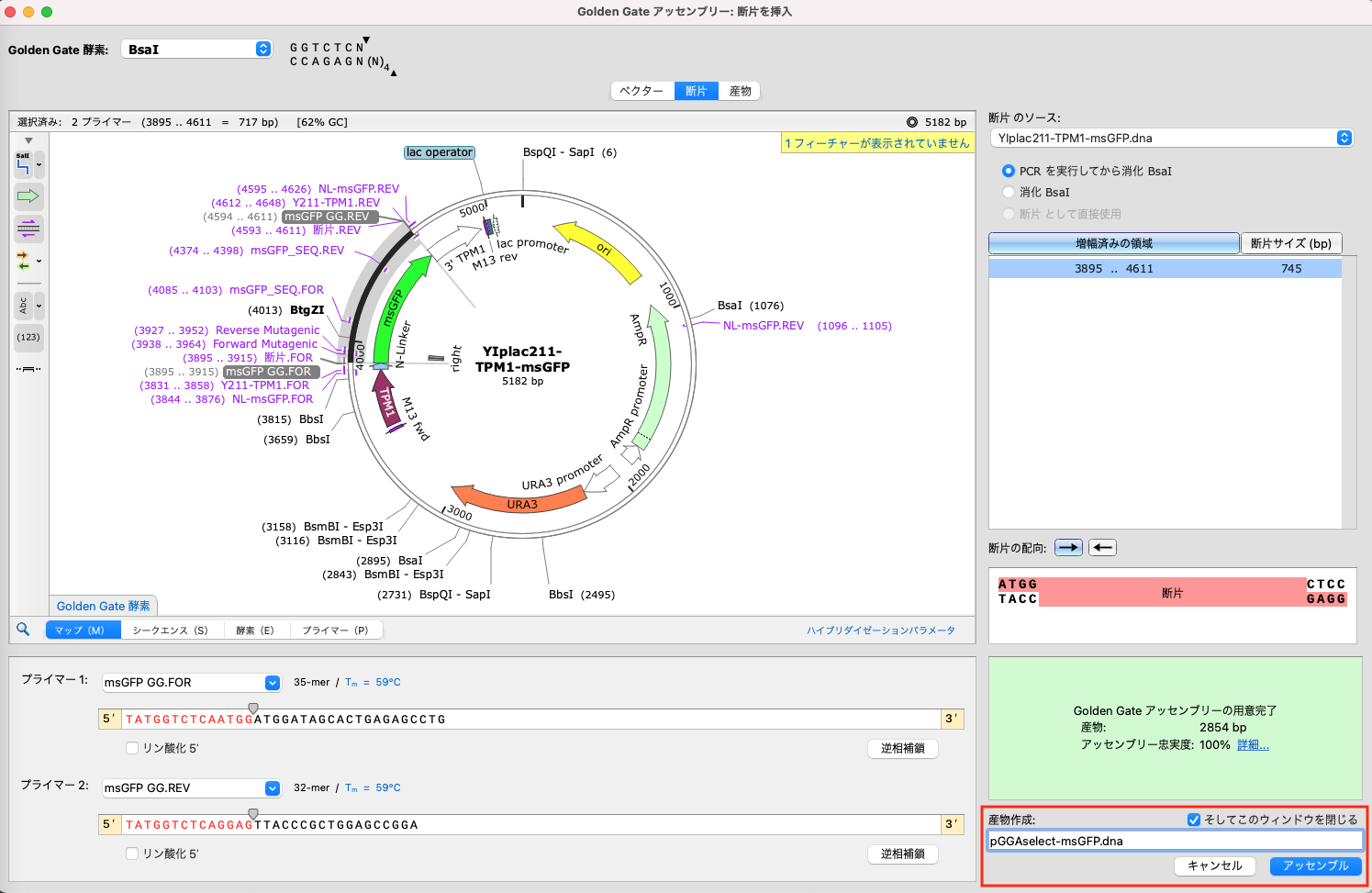
シークエンスビューでクローニング後の配列を確認したり、プライマービューで実際に使用するプライマーの配列を確認します。問題がなければ、右下の「産物作成」で任意のファイル名(今回は “pGGAselect-msGFP” )を付けて「アセンブル」をクリックします。
直鎖状ベクターや断片のファイルを作成する場合は、「ファイル作成」にチェックを入れ任意のファイル名をつけて下さい。
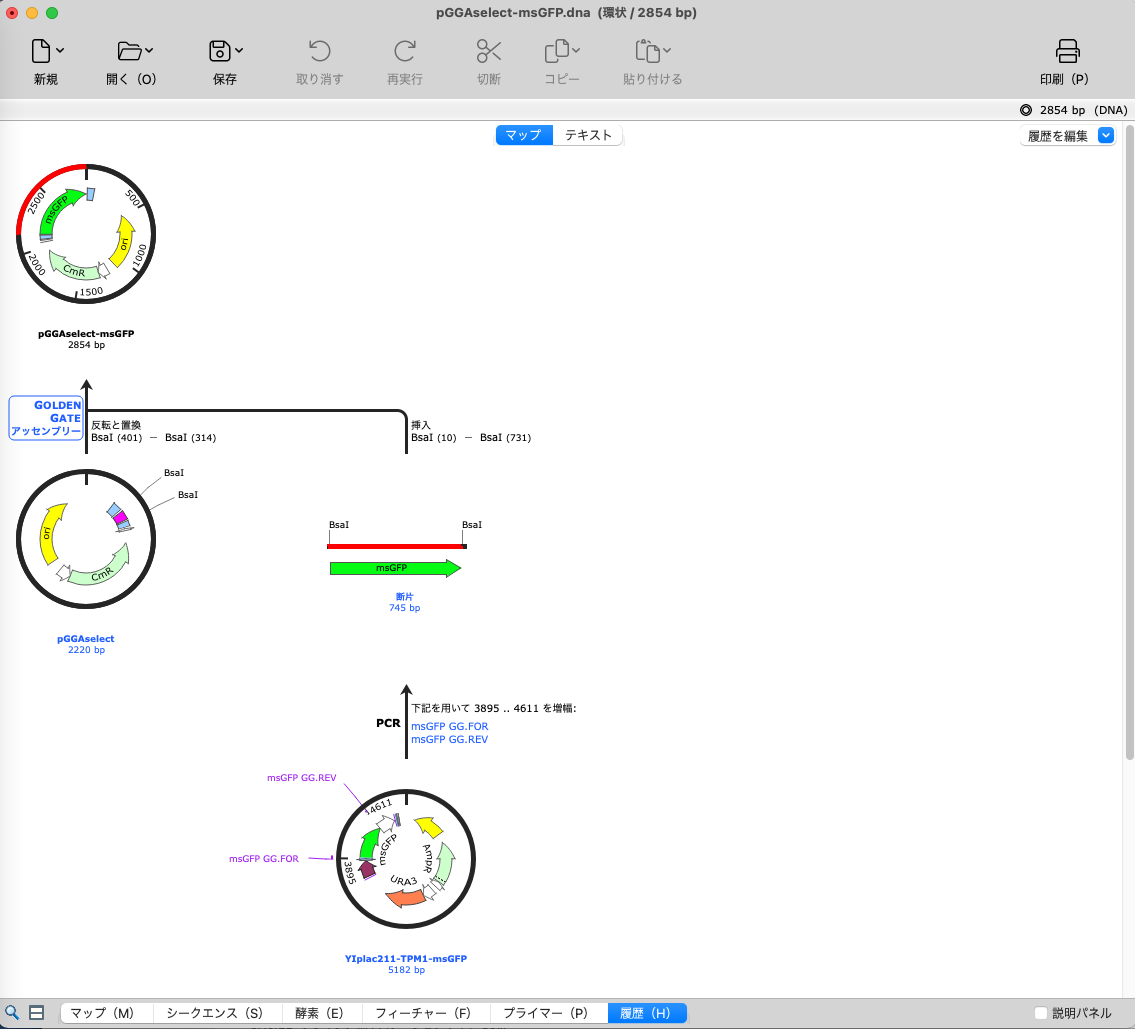
2-3. 履歴ビュー
履歴ビューで Golden Gate アッセンブリーの過程を確認できます。
チュートリアルで紹介した機能や操作方法に関するお問い合わせはお問合せフォームよりお願いします。




