SnapGeneの基本操作
このチュートリアルでは、SnapGeneの基本的な操作方法について学びます。チュートリアルで使用するデータもダウンロードできるようになっており、ハンズオンで実際に操作することでより深く SnapGene の機能や操作方法を学ぶことができるようになっています。
1. SnapGene 画面構成
フォルダパネルの表示/非表示の切り替えは、左上にある矢印アイコン ![]() を使用します。カーソルを画面左に移動させると自動的にフォルダパネルが開きます。
を使用します。カーソルを画面左に移動させると自動的にフォルダパネルが開きます。


1ビュータブ:画面表示の切り替えを行います。
2サイドツールバー:マップやシークエンスのフィーチャーの非表示/表示切替を行います。
3トップツールバー:ビューの分割表示、マップやシークエンスの表示設定を行います。

1-1. フォルダパネル
1. フォルダパネルの 「Open Folder」をクリックし、コンピューターシステム上にある任意のフォルダ(プロジェクトフォルダ)を選択します。

2. 選択したフォルダが、プロジェクトとしてフォルダパネルに表示されます。
フォルダパネルではサブフォルダとファイルが階層的に表示されます。

3. 別のプロジェクトフォルダを開く場合、プロジェクトフォルダ名の右横にある (3点リーダー)をクリックし、「プロジェクトフォルダを開く」 を選択します。

4. 選択したプロジェクトフォルダが開きます。

5. SnapGene 上から新規フォルダを作成することも可能です。新規にプロジェクトファイルを作成する場合は、「Create Project...」を選択します。

SnapGene のインポート機能を使用して開いたファイルが未保存の場合、フォルダパネルに表示されません。フォルダパネルに表示させるには、ファイルを保存する必要があります。
ファイルの内容(シークエンスの塩基置き換えやプライマーの作成など)に変更が加えられた後に保存されていない場合、フォルダパネルの上部にメッセージ:"You have unsaved changes" が表示されます。変更が加えられている未保存ファイルを一括で保存するには、フォルダパネルに表示される "You have unsaved changes" の右横にある (3点リーダー)をクリックし、「すべてを保存」 を選択します。

1-2. タブ
保存されていないファイルは、タブにカーソルを合わせた時または該当するタブを開くとタブ名の右側に・(オレンジ色の丸)が表示されます。

 : ファイルは保存されています。
: ファイルは保存されています。
 : 変更が加えられたファイルです。ファイルは保存されていない状態です。
: 変更が加えられたファイルです。ファイルは保存されていない状態です。
 : インポートしたファイルです。ファイルは保存されていない状態です。
: インポートしたファイルです。ファイルは保存されていない状態です。
右上の (3点リーダー)をクリックすると、現在開いているファイル、または最近使用したファイルの一覧が表示されます。

2. SnapGeneを起動する
本チュートリアルでは、pcDNA3.1+/C-Myc を使用します。下記ページより、pcDNA3.1+ C-Myc.dnaファイルをダウンロードして任意の場所に保存してください。
Mammalian expression vector with a CMV promoter for expressing C-terminally Myc-tagged proteins.
www.snapgene.com
SnapGeneを起動すると次のような画面になります。

3. ファイルを読み込む
画面左上の 「開く」ボタンのメニューから「ファイルを開く」を選択し、ダウンロードしたプラスミドデータ(pcDNA3.1+ C-Myc.dna)を読み込みましょう。

ファイルを指定する画面が出てきたら、ダウンロードしたファイルを選択して「開く」を押してください。次のような画面になっていれば、読み込み成功です。

4. ツールバー
サイドツールバー1 では主にマップやシークエンス上に表示する項目、トップツールバー2 ではマップやシークエンスの表示方法を設定します。ツールボタンの機能が無効 (![]() ) になっている場合は、ボタンをクリックすると有効 (
) になっている場合は、ボタンをクリックすると有効 (![]() ) になります。
) になります。

4-1. サイドツールバーの詳細
サイドツールバーから、マップやシークエンスにフィーチャー、制限酵素や ORF などの表示切替(表示/非表示)や表示形式の設定を行います。
 制限酵素の表示切替
制限酵素の表示切替
制限酵素の表示切替を行います。右側にある矢頭をクリックすると、制限酵素の種類を選択できます。

 フィーチャーの表示切替
フィーチャーの表示切替
フィーチャー(CMV promoter など)の表示切替を行います。
 プライマーの表示切替
プライマーの表示切替
プライマー情報を含んでいる配列データの場合は、プライマーも表示されます。プライマーの表示切替(表示/非表示)を行います。
 プライマー表示機能 :無効
プライマー表示機能 :無効 プライマー表示機能 :有効
プライマー表示機能 :有効 翻訳とORFの表示切替
翻訳とORFの表示切替
オープンリーディングフレーム(ORF)の表示切替を行います。デフォルトでは、「開始コドン(ATG)があり、最小アミノ酸数が75以上」の場合に ORF と判断するよう設定されています。右側にある矢頭をクリックすると、表示する ORF の設定を変えることができます。「翻訳オプション」で ORF と判断する条件を変えることもできます。
 色の表示切替とGC含有の表示
色の表示切替とGC含有の表示
任意のフィーチャーに色を付けたり、GC含有量に応じた色分けを表示させることができます。右側にある矢頭をクリックすると、詳細設定ができます。
 位置の表示切替
位置の表示切替
制限酵素やプライマーの塩基の位置の表示切替を行います。
 アライメントの表示
アライメントの表示
選択されている配列ファイルを参照配列とし、配列のアライメントを行います。
4-2. トップツールバーの機能
トップツールバーからマップやシークエンスの表示方法などを設定します。Display メニューにあるいくつかの機能は、サイドツールバーに用意されている機能と重複します。
マップビュー
 コンテンツの分割表示
コンテンツの分割表示
2つのビューを画面上下に分割して表示します。
 環状マップの表示
環状マップの表示
環状マップを表示します。
 水平マップの表示
水平マップの表示
水平マップを表示します。環状マップでは見にくい時に使用すると便利です。
 ディスプレイ
ディスプレイ
サイドツールバーにある表示/非表示機能は、トップツールバーの Display から選択することも可能です。

![]() : 酵素を表示
: 酵素を表示
![]() : フィーチャーを表示
: フィーチャーを表示
![]() : プライマーを表示
: プライマーを表示
![]() : Show Translations and ORFs
: Show Translations and ORFs
![]() : Show Colors
: Show Colors
![]() : Show Locations
: Show Locations
![]() : Show Alignments
: Show Alignments
シークエンスビュー
 ライン幅の自動折り返し表示
ライン幅の自動折り返し表示
ウィンドウのサイズに応じてライン幅を変更して表示します。
 ライン幅の固定
ライン幅の固定
ライン幅を 30bp、50bp、75bp、100bp、150bp、300bp、500bp で固定します。またはライン全てを水平スクロールで表示します。
 ディスプレイ
ディスプレイ
トップツールバーの Display から、サイドツールバーに用意されている機能を選択することも可能です。

![]() : 酵素を表示
: 酵素を表示
![]() : フィーチャーを表示
: フィーチャーを表示
![]() : プライマーを表示
: プライマーを表示
![]() : Show Translations and ORFs
: Show Translations and ORFs
![]() : Show Colors
: Show Colors
![]() : Show Locations
: Show Locations
![]() : Show Alignments
: Show Alignments
Use Alternative Format:シークエンスのフォーマットを切り替えます。
User Fixed Line Width: ライン幅を固定します。ツールボタンの ラインの固定(![]() )と同じ機能です。
)と同じ機能です。
Use 3-Letter Amino Acid Codes : アミノ酸コードを1文字表記と3文字表記に切り替えます。
Show Minimap: シークエンスビューの下部にミニマップを表示します。
5. 画面表示の切替え
画面表示の切替えは上部にあるビュータブで行います。2つのビューを分割して表示することも可能です。ビューを分割で表示するには、トップツールバーの Split Content (![]() ) をクリックします。
) をクリックします。

5-1. シークエンスビュー
シークエンスビューでは、全長の塩基配列を確認することができます。 シークエンスビューでもツールバーを使うことができるので、制限酵素やORFなどの表示切替ができます。

また、塩基配列の編集もすることができます。例えば、777塩基目のグアニン(G)をアデニン(A)に変更する場合は、該当箇所をドラッグして “A” を入力します。

ダイアログが開き、選択した塩基(777塩基目)と変更する塩基がフィールドに入力されているのを確認し、「挿入」ボタンをクリックすると編集が完了します。


編集内容を取り消す場合は、ツールバーにある「取り消す」で元に戻すことができます。
5-2. 酵素ビュー
プラスミドに含まれる制限酵素認識部位の詳細を見ることができます。 チェックボックスをクリックすることで、任意の制限酵素を追加したり除外したりすることもできます。

また、「酵素の選択」から任意の酵素セットを作成することもできます。研究室でよく使う制限酵素セットを作成しておくと便利です。


「酵素の詳細設定」から、ドロップダウンメニューに表示するサプライヤーの優先順位や表示する数を変更することが可能です。


5-3. フィーチャービュー
フィーチャー(T7 promoterなど)の詳細を確認したり、任意のフィーチャーを除外をすることができます。
右向きシェブロン [ ] をクリックすると詳細情報が表示されます。フィーチャーにカーソルを合わせると、右端に編集(![]() )、非表示(
)、非表示(![]() )アイコンが表示されます。
)アイコンが表示されます。
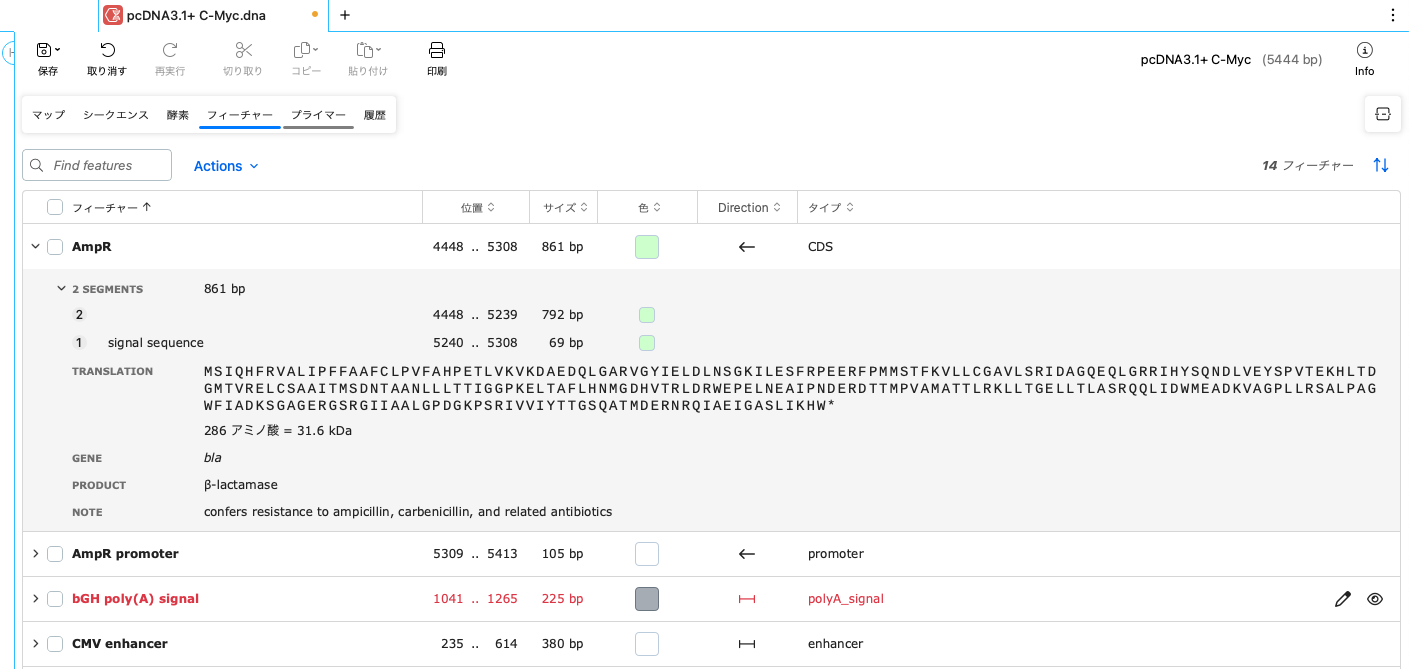
編集アイコン(![]() )をクリックすると、フィーチャー編集ダイアログが開き、フィーチャーを編集することができます。
)をクリックすると、フィーチャー編集ダイアログが開き、フィーチャーを編集することができます。

追加したフィーチャーもフィーチャービューで確認することが可能です。
シークエンスビューで任意の領域をドラッグして、ツールバーの「フィーチャー」から「フィーチャーを追加」をクリックすると、任意をフィーチャーを追加することができます。


5-4. プライマービュー
プライマー情報の詳細を確認することができます。シークエンス用プライマーなどをプラスミドごとに登録しておくと便利です。
SnapGeneファイルにプライマー情報が登録されている場合は、ツールバーの「プライマー」から「プライマーをインポート」をクリックすると、そのプライマーを追加することができます。

また,シークエンスビューで任意の領域をドラッグして、ツールバーの「プライマー(P)」から「プライマーを追加(A)」をクリックすると、任意のプライマーを追加することができます。

追加したプライマーもプライマービューで確認できます。

5-5. 履歴ビュー
編集の履歴を自動で記録し表示します。例えば、先に行った「777塩基目のグアニンをアデニンに変更」もしっかりと記録されています。

Show as Text (![]() ) をクリックすると、テキスト形式による履歴が表示されます。
) をクリックすると、テキスト形式による履歴が表示されます。


























