サンガーシークエンスデータの解析
このチュートリアルでは、実際にデータを利用してサンガーシークエンスデータの解析方法についてご紹介します。
1. 【準備】DNA配列を入手する
本チュートリアルでは、pENTR223-TP53 プラスミドの配列を使用します。NCBI のデータベースで、以下のアクセッション番号を検索してインポートしましょう。詳しい方法は Geneious Prime 基本操作 [1] : NCBI からのインポートを参照してください。
Accession: LT739496
本チュートリアルで使用するシークエンス解析結果のファイルは、以下からダウンロードして Geneious に追加してください。ファイルのインポート方法は Geneious Prime 基本操作 [1] :フォルダの新規作成とファイルのインポートを参照してください。
2. サンガーシークエンスデータの解析方法
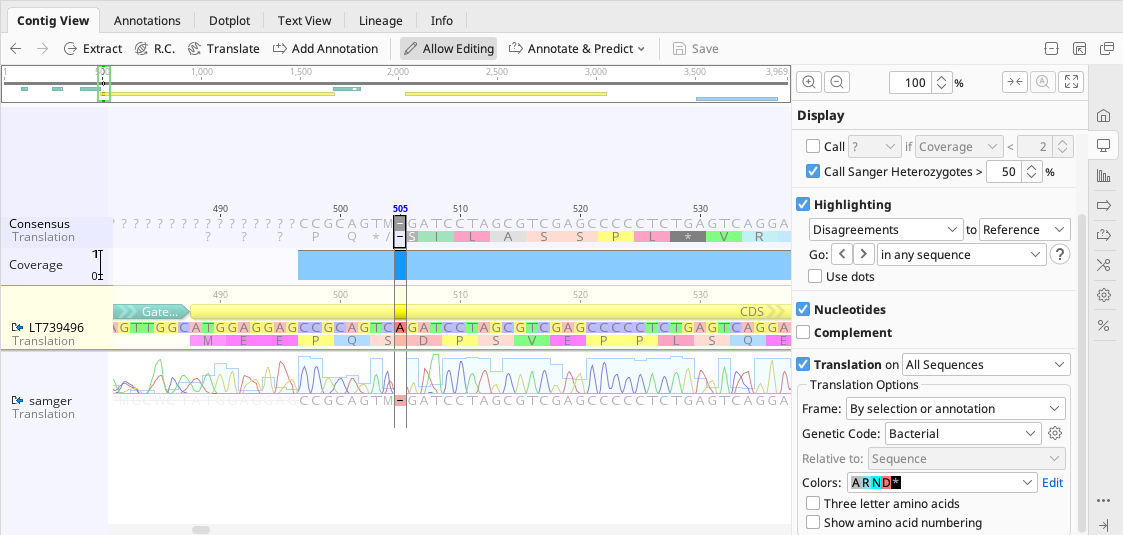
ドキュメントテーブルの ![]() How To Align Chromatograms to a Reference Sequence_Example を選択すると、ドキュメントビューアに塩基の信頼度を示すベースコールクオリティのグラフが表示されます。
How To Align Chromatograms to a Reference Sequence_Example を選択すると、ドキュメントビューアに塩基の信頼度を示すベースコールクオリティのグラフが表示されます。
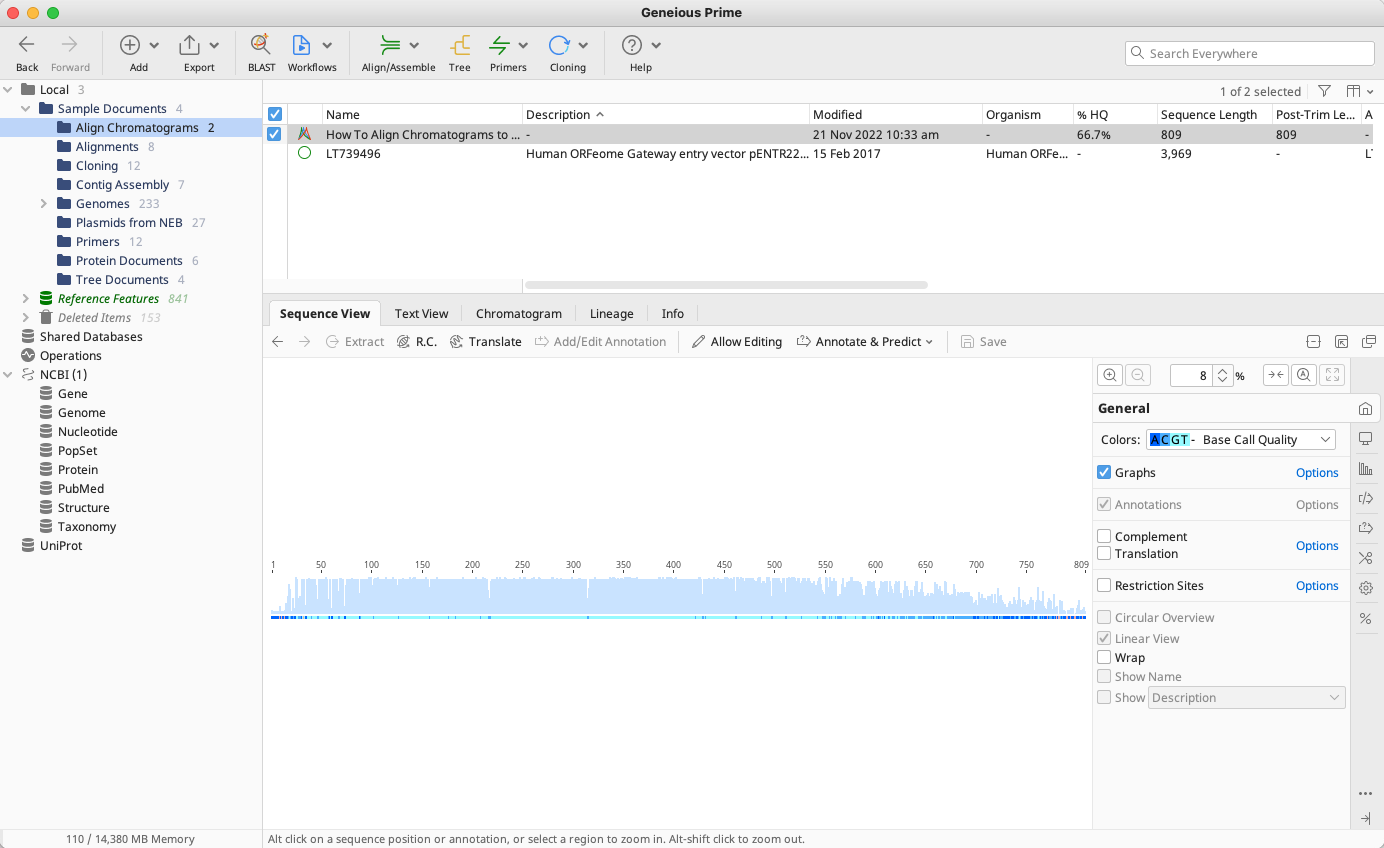
2-1. トリミングの方法
ベースコール全体を見ると、両側のクオリティが低下していることが分かります。この状態で解析を行うと正確な結果が得られない可能性が高いので、データのトリミングを行います。
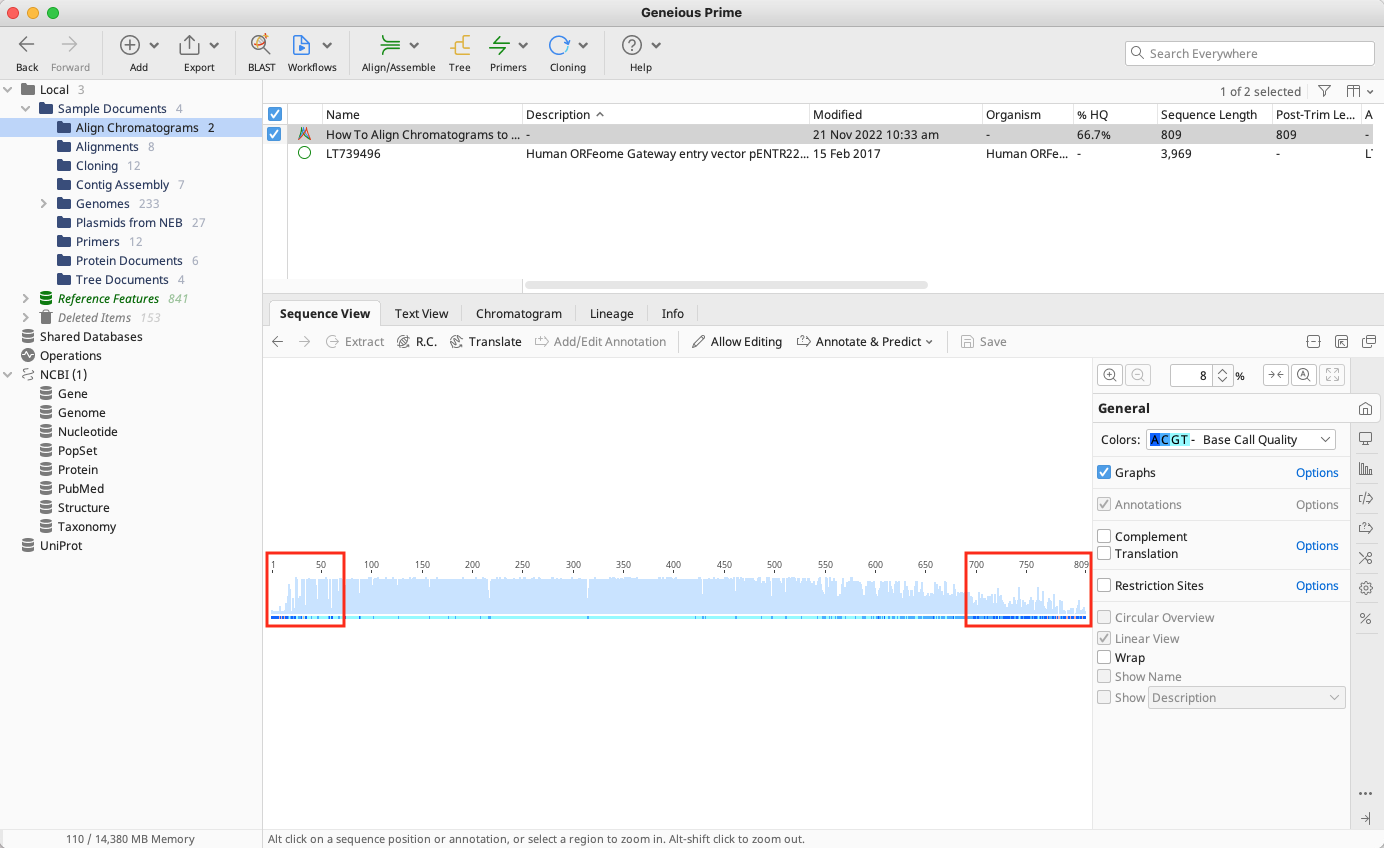
ファイルを選択した状態で、上部の Annotate & Predict から Trim Ends… へと進みます。
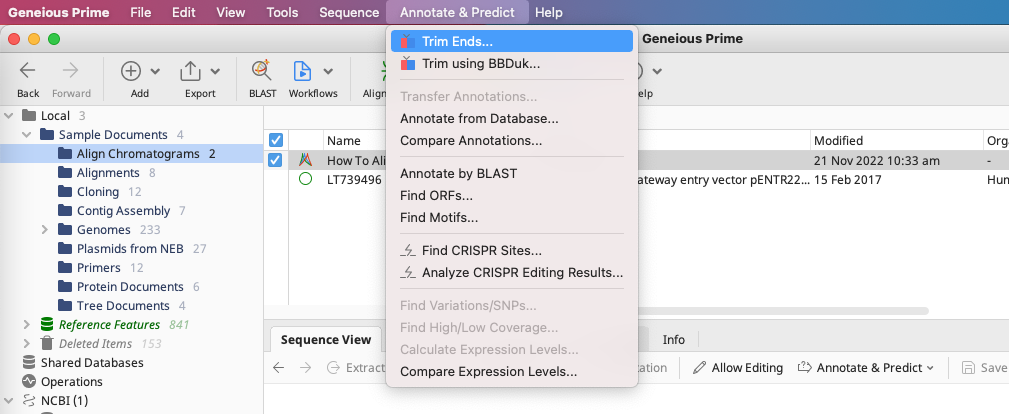
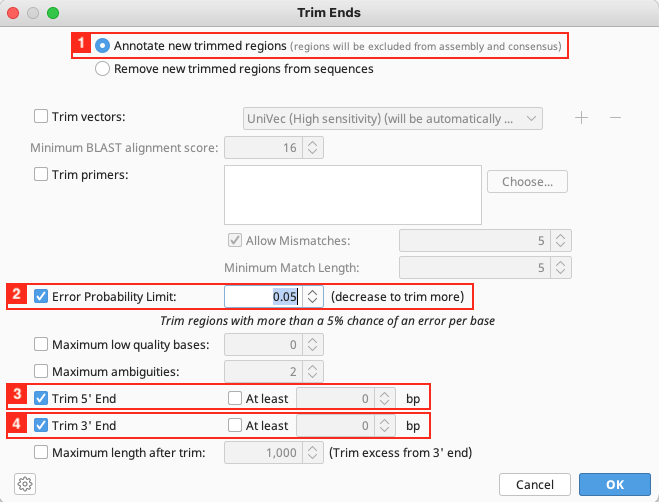
Trim Ends ダイアログで許容できるレベルに設定して OK をクリックするとトリミングは完了し、トリミングされた領域にはマーカーが付きます。 ここではデフォルト設定を利用します。
1Annotate new trimmed regions を選択し、新たにトリミングする領域のアノテーションを行います。
2Error Probability Limit:クオリティの値に基づいたアルゴリズム( modified-Mott algorithm )を使用して末端をトリミングします。各塩基について、塩基のエラー確率をエラー確率のカットオフ値(デフォルト 0.05)から差し引いてベーススコアを算出します。
3 Trim 5’ End:配列の5’末端をトリミングするように設定します。
4 Trim 3’ End:配列の3’末端をトリミングするように設定します。
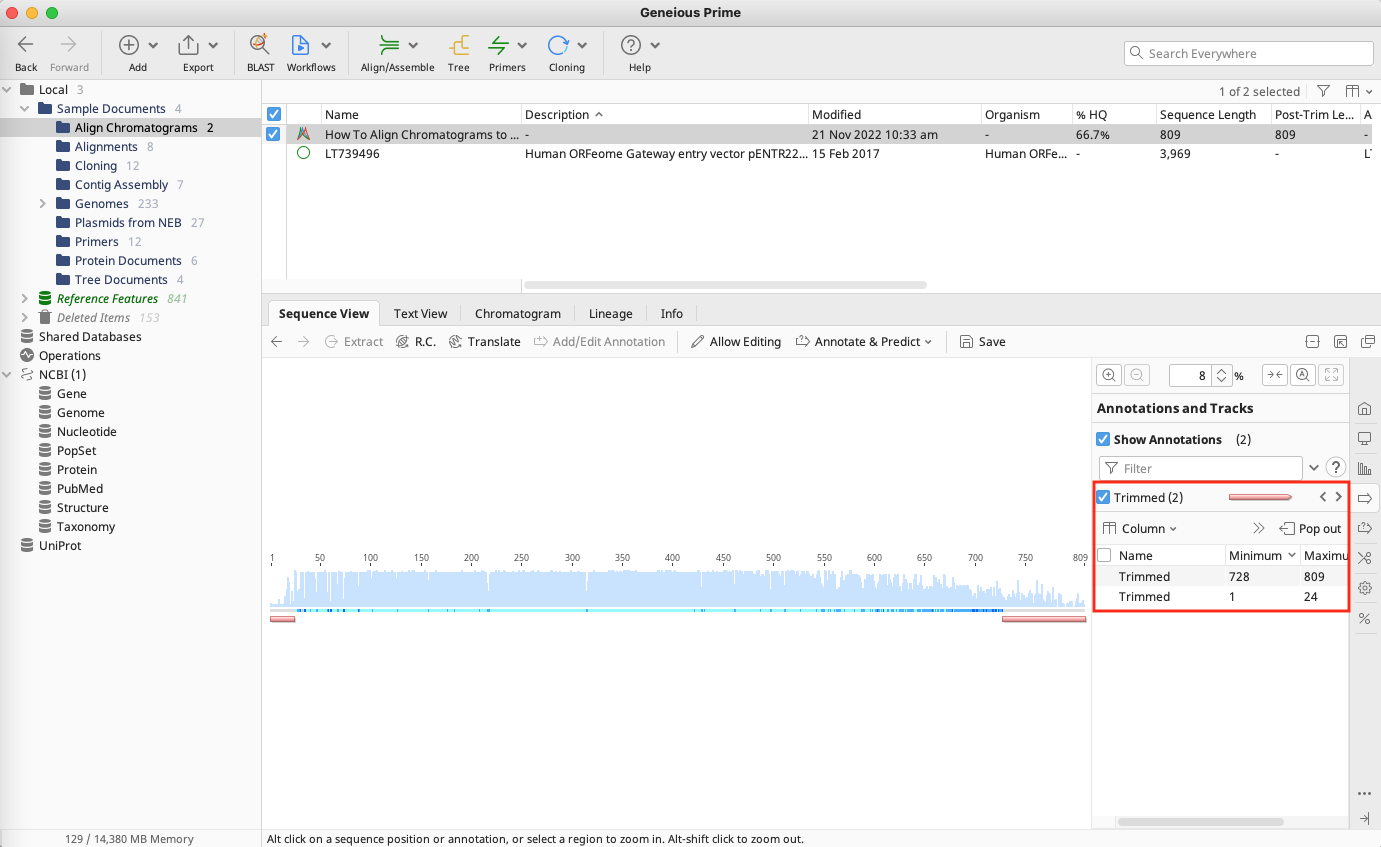
トリミング領域がグラフに追加され、またトリミングされた情報はコントロールパネルに表示されます。
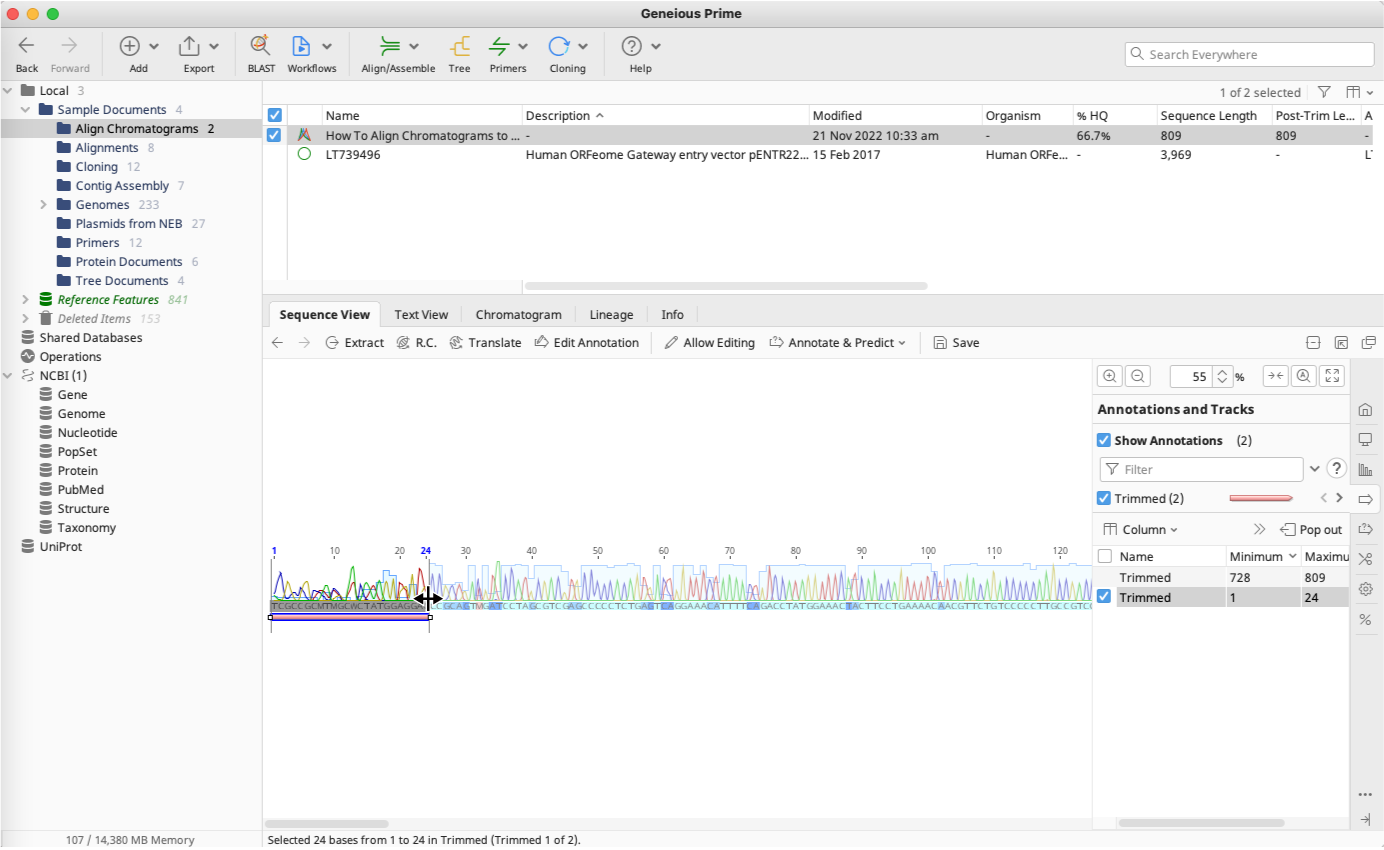
トリミング領域を変更する場合、トリミング領域を示すバーを選択し、バーの両端に表示される白い正方形をドラッグします。
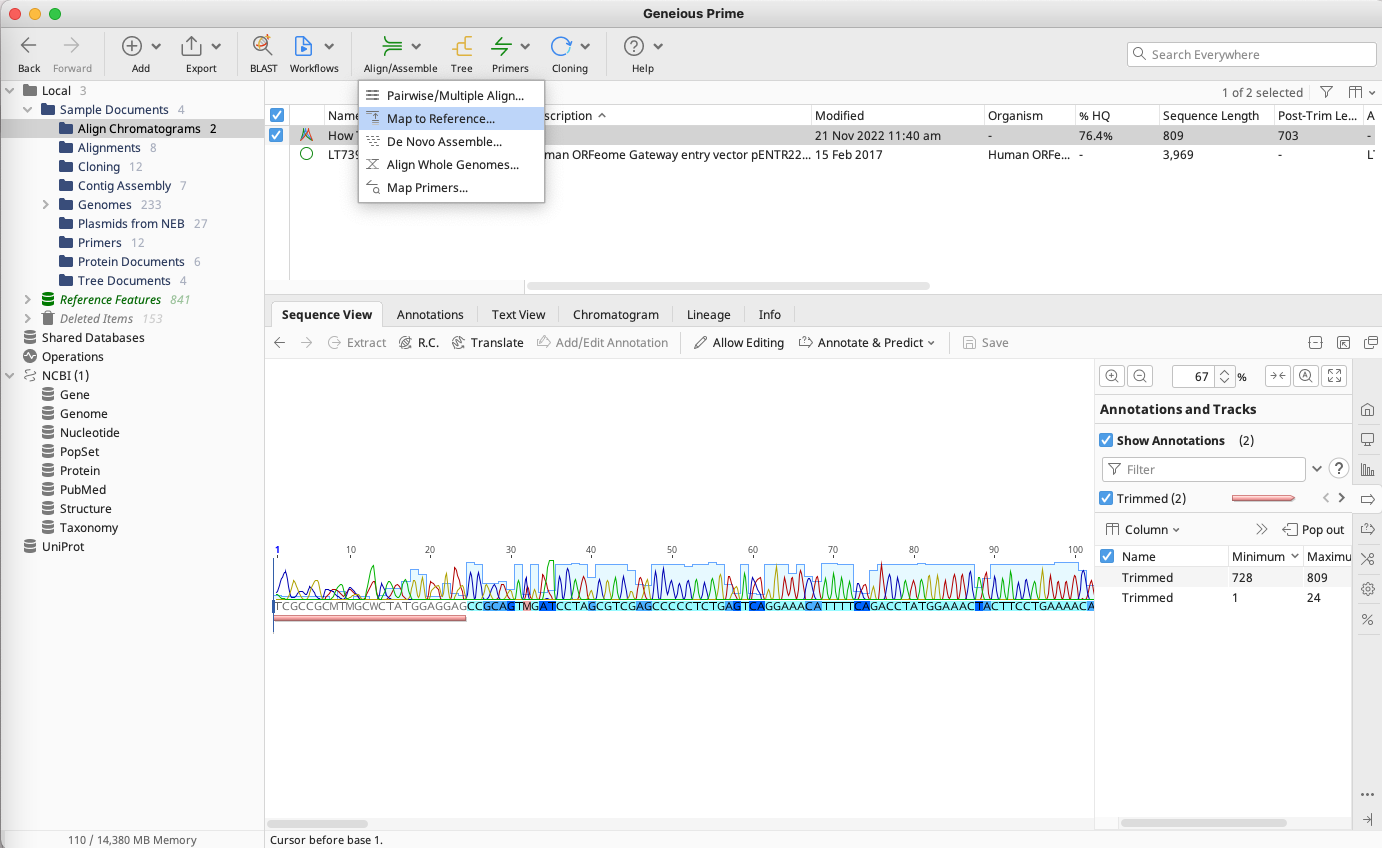
2-2. リファレンス配列へのマッピング
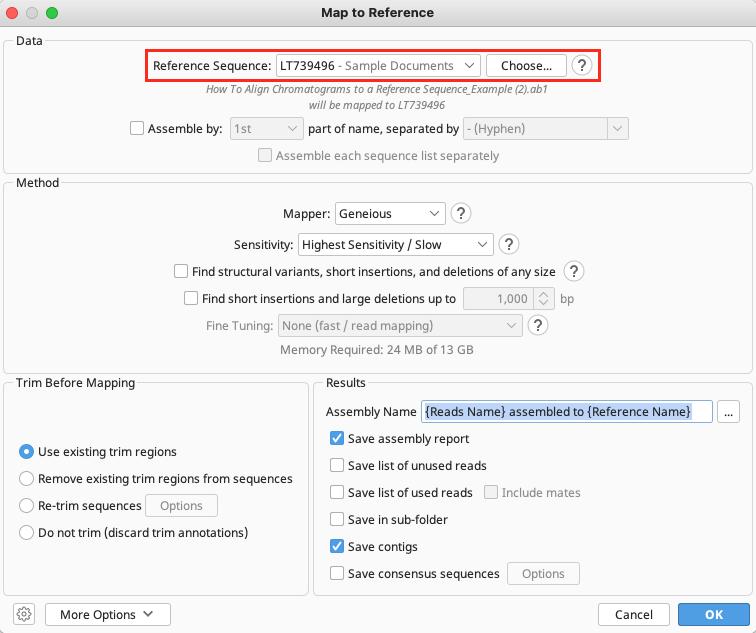
トリミングが完了したら、 ![]() をクリックしてデータの保存を行います。 そして、リファレンスシークエンスへのマッピングを行います。ツールバーの Align/Assemble から Map to Reference… へと進みます。
をクリックしてデータの保存を行います。 そして、リファレンスシークエンスへのマッピングを行います。ツールバーの Align/Assemble から Map to Reference… へと進みます。
Reference Sequence :で、使用するファイルを指定します。 Chooseボタンをクリックし、リファレンスシークエンスのファイル(今回は LT739496 )を選びます。必要に応じて各種設定を変更し、OKボタンをクリックします。
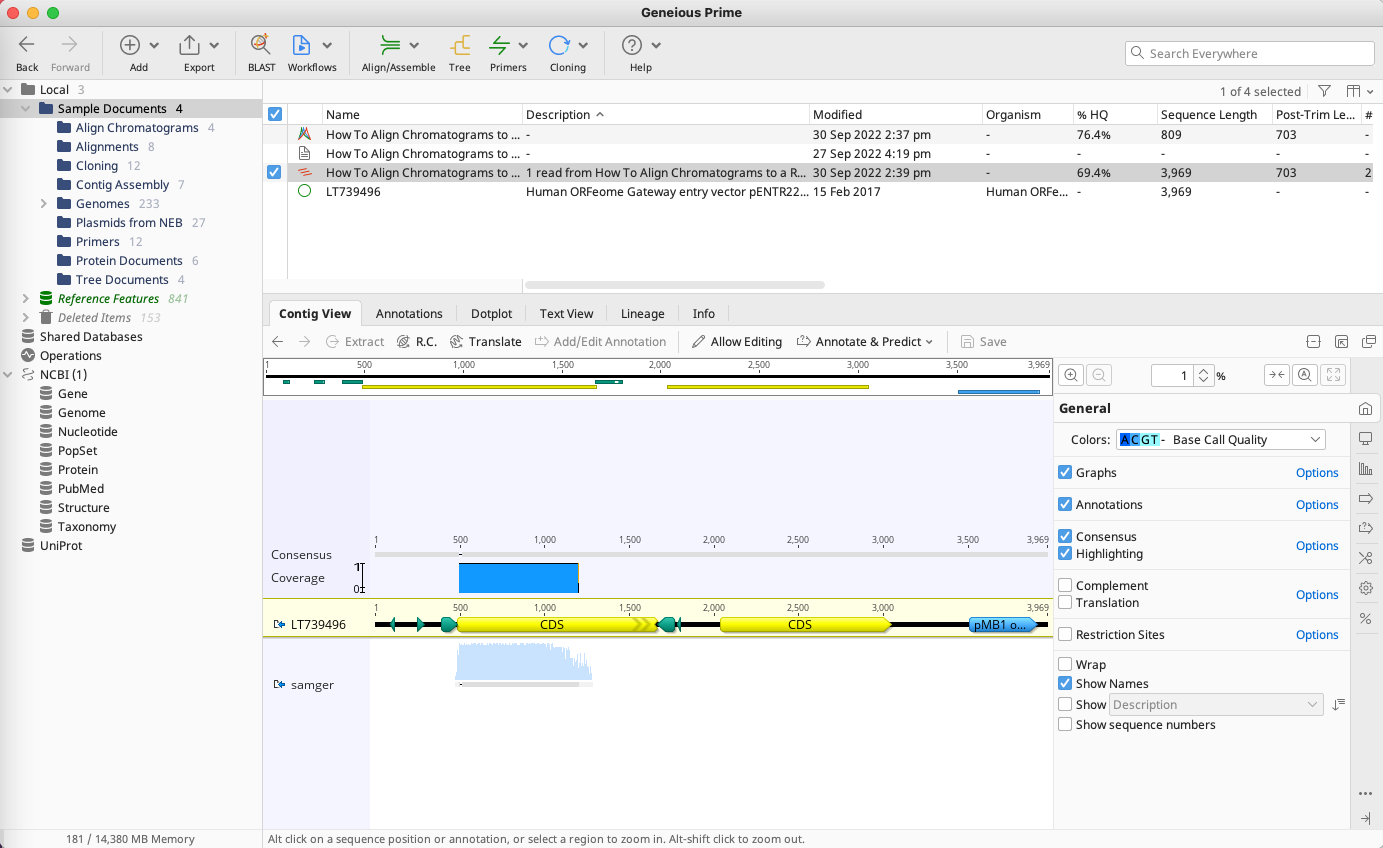
ドキュメントテーブルに ![]() How To Align Chromatograms to a Reference Sequence_Example.ab1 assembled to LT739496 が追加され、 ドキュメントビューアにマッピング結果が表示されます。
How To Align Chromatograms to a Reference Sequence_Example.ab1 assembled to LT739496 が追加され、 ドキュメントビューアにマッピング結果が表示されます。
マッピング結果では、データがリファレンスと一致している場合は灰色に表示され、不一致の場合は黒色または - になります。
![]() をクリックすると、塩基の挿入・置換・欠失の編集が可能になります。Consensus 部分の変更は自動的に完了します。
をクリックすると、塩基の挿入・置換・欠失の編集が可能になります。Consensus 部分の変更は自動的に完了します。