Geneious Prime FAQ
購入や注文方法に関するお問い合わせは、購入に関するFAQをご参照下さい。
一般
- Geneious Primeのシステム条件はどのようになっていますか?
-
Windows
Windows 7, 8, 8.1, 10 and 11
- Geneious Prime 2020 以降のバージョンは32 bit版には対応しいません。
- 次のプラグインには対応していません:"TopHat "、"Velvet Optimiser"
macOS
Geneious Prime 2023: MacOS 10.14 以降
Geneious Prime 2020 ~ 2022: 10.11 以降
Geneious Prime 2019: MacOS 10.8 ~ 10.15** Geneious Prime2019 は MacOS 10.8-10.15 のみで起動します。
- 32 bit版には対応していせん。
Linux
Geneious R10 以降:Ubuntu Desktop LTS (18.04 and 20.04).
Geneious R9 以前:Ubuntu Desktop LTS 16.04
- Geneious R10 以降のバージョンは32 bit版には対応していません。
HD空き容量 各OS共通
プロセッサ:x86 64-bit
メモリ容量:2 GB 以上
HD空き容量:2 GB 以上
ディスプレイ: 1024 x 768以上
参照:Supported Operating Systems - Geneious Prime はオフラインでの使用可能ですか? 使用する際は常時インターネットに接続されている必要がありますか?
-
Geneious はインストールまたはアップデート/アップグレードを行う際、インターネットに接続されている必要があります。インストール後はオフラインでも利用は可能ですが、NCBI データベースなどにアクセスする場合はインターネットに接続されている必要があります。
- Geneious Primeのトライアル版はありますか?
-
Geneious Primeトライアル版はBiomatters社のウェブサイトよりダウンロード頂けます。
https://www.geneious.com/free-trial/
トライアル版は機能制限なく14日間ご利用頂けます。トライアル版が終了すると、機能が制限される Restriction Mode(制限モード)となり、主にビューアーとしてご利用いただけます。

- 日本語によるユーザーサポートの提供はありますか?
-
弊社より Geneious Prime をご購入頂いたお客様であれば、日本語によるインストールやアクティベーション解除に関する無償サポートを、ライセンスが有効な限り(弊社より新規購入/更新1年以内)何度でもご利用いただけます。
ご質問は開発元である Biomatters社への照会を要する場合がございますので、ご回答まで1〜2営業日ほどお時間を頂きますことご了承ください。
サポートはGeneious サポートフォームよりお申し込みください。
Note:サポート対象外
操作や機能、結果解釈に関しましては、サポート対象外となります。
開発元である Biomatters社に直接お問い合わせ下さいますようお願い致します(対応言語:英語)。
お問い合せ先:
Geneious - Submit a request弊社よりご購入頂いたお客様であれば、ユーザーページより弊社オリジナルのステップ・バイ・ステップガイドをご利用頂けます。
- Geneious Prime ステップ・バイ・ステップガイドとはどのようなものですか?
-
Geneious Primeステップ・バイ・ステップガイドは、開発元 Biomatters 社の提供するチュートリアルをベースに、弊社で作成したオリジナルガイドです。専用ページにアクセスして閲覧頂くオンラインによるガイドとなります(PDF による提供はありません)。下記ページよりガイドの目次をご覧いただけますのでご参照下さい。
- Geneious Prime を論文で引用するには?
-
Geneious Prime を論文で引用するには、Methods セクションに以下のように記述下さい。
"Geneious Prime 20XX.x.x (https://www.geneious.com)"
XX.x.x. にはバージョンが入ります。バージョン情報を確認するには、メニューバーの Helpから、About Geneious Prime を選択します。Geneious Prime® に続く数字がバージョンとなります。
Geneious R11 以前のバージョン
論文でGeneious R11 以前のバージョンを引用する場合は、Methods セクションに以下のように記述下さい。
"Geneious x.x.x (https://www.geneious.com)" .
上記は 開発元である Biomatters Inc 社が FAQ にて掲示している情報となります。論文投稿先によって規程が異なりますので、投稿先の投稿規定に従い記載下さい。Geneious が使用された論文・ジャーナルで、実際に利用されている論文やジャーナルを紹介しておりますのでご参照ください。
参照 How do I cite Geneious or Geneious Prime in a paper? - Geneious Prime と SnapGene はどのように違いますか?
-
Geneious Prime は、NGSの処理と解析(トリミング、アセンブル、マッピング、バリアントコールなど)を行うことが可能です。ゲノム配列のアセンブリーやアノテーションなどで多く利用される機能を備えており、ゲノム疫学の研究など大量サンプルデータを比較解析したり網羅的に解析するのに適しています。
SnapGene は、クローニング実験手順やプラスミドなどの DNA 断片の視覚化および文書化をサポートします。また、DNAクローニングやPCRの計画や記録、塩基配列データの管理など遺伝子工学実験のスムーズな進行を支援します。 SnapGene はアセンブリ機能は備えていないため、de novo アセンブリまたは参照配列へのマッピングなどはできません。
一般的に、PCRプライマー設計やクローニング・シミュレーションなど分子クローニング作業には SnapGene が利用され、NGS のようなハイスループットシークエンシングを扱う必要があったり、微生物ゲノムで作業する場合は Geneious Prime が利用されます。
Geneious Prime と SnapGene は分子生物学研究やバイオテクノロジー分野で利用される幅広い機能を備えており、共通する機能がいくつかあります。SnapGene と Geneious Prime それぞれのラーニングページで、共通の機能を紹介したチュートリアルを用意しておりますのでご参考下さい。
- Geneious Prime のカタログはありますか?
-
Geneiousのデジタルパンフレットを下記よりダウンロード頂けます。
エディション
- Geneious Prime のアカデミック版対象について知りたいのですが。
-
アカデミック対象ユーザは下記の通りです。
1. 大学、高専、高校等教育機関
2. 文部科学省が認定している医療機関(大学付属病院)
3. 国立研究開発法人、独立行政法人、大学共同利用機関法人、公益財団法人、地方公営企業(地方公共団体が経営する企業)
4. 特定非営利活動法人などのNPO団体 - Geneious Prime の一般・アカデミック版は機能的に違うのですか?
-
一般・アカデミック版に機能的な違いはありません。製品の価格のみが異なります。
- 産学官連携のパートナーとして大学と共同研究を行っておりますが、Geneious Prime アカデミック版は購入できますか?
-
パーソナルライセンスの場合:民間企業の場合はアカデミック版の対象とはならず、一般向け価格が適用されます。実際に使用されるユーザーが教育機関と兼任している場合はアカデミック版の対象となります。
グループライセンスの場合:Geneiousを使用する機関・団体(請求先情報)がアカデミック版対象の判断基準となります。使用者に教育機関に所属される方を含む場合でも、使用する場所が民間企業の場合は一般向け価格となります。
- Geneious Prime アカデミック版の購入に必要なものは何ですか?
-
アカデミック版をご購入頂く場合、所属する団体名とE-mailアドレスにてアカデミック版対象か判断します。アカデミック版の対象となるメールアドレスは、アカデミック版対象機関のドメイン(.ac、.govなど)である必要があります。
- 大学を卒業したら、アカデミック版は使用できなくなりますか?
-
ライセンスが有効であれば継続してご利用頂けますが、ライセンス更新時に教育機関発行のEメールアドレスメールアドレスをご提示いただく必要があります。 客員教授/研究員として大学に在籍されているものの、教育機関からのe-mailアドレスが発行されていない場合は、在籍・在職を証明する書類のコピーを提出下さい。
- Geneious Prime 学生版はありますか?
-
学生を対象としたライセンス (Student Personal) につきましては、弊社では販売はしておりません。Student Personal の購入は開発元であるBiomatters, Ltd.に直接ご注文下さいますようお願い致します。
ライセンス
Geneious の License Agreement は、下記 Biomatters社のウェブサイトよりご覧いただけます。
Geneious Terms and Conditions | Effective 19 March 2024
- パーソナルサブスクリプションとチームサブスクリプションの違いは何ですか?
-
パーソナルサブスクリプション vs チームサブスクリプション
パーソナルサブスクリプションの場合、1ライセンスにつき1ユーザーのみ利用が可能です。チームサブスクリプションの場合、1ライセンスに付属するシート数分のユーザー利用が可能です。
シートごとにユーザーを指定する必要がある点はパーソナルサブスクリプションと同じですが、パーソナルサブスクリプションが注文時にユーザー情報の登録が必要なのに対し、チームサブスクリプションの場合はライセンス管理者情報の登録のみとなります。ユーザーの指定(シートの割り当て)は納品後に行うことができ、また、ユーザーの変更もライセンス管理者が実施することができます。
2024年3月21日、チームサブスリプションの販売開始の伴い、従来のグループサブスクリプションの新規販売は終了いたしました。グループサブスクリプションのアクティベーションはライセンスキーを利用して行う方法(マシンベースドライセンス)でしたが、チームサブスクリプションは Email による方法(ユーザーベースドライセンス)になります。グループサブスクリプションスはシート数 = インストール可能台数でしたが、チームサブスクリプションはシート数 = ユーザー数となります。グループサブスクリプションスからチームサブスクリプションへの切り替えはできません。 すでにグループサブスクリプションを導入頂いている(ライセンスキーのあるライセンスを利用中)場合は、従来通りのアクティベーション方法となります。 設定変更などの必要はなく継続してご利用頂けます。パーソナルサブスクリプション
- 最小発注単位:1ライセンス
- ご注文時にライセンスごとにユーザー(使用者)情報の提出が必要
- ご注文時に提出頂いた情報をもとに、 GraphPad Software社 がユーザーを登録します
- 1ライセンスにつき、1ユーザーのみ使用ができます
- ライセンス または コンピューターを共用し、第三者が使用することはできません
- 同ライセンスにライセンス数の追加不可
- Geneious クラウド ストレージ容量:10GB(1ライセンスあたり)
- クラウドワークスペースの共有不可
チームサブスクリプション
- 最小発注単位:1ライセンス 2シート(シート数 = ユーザー数)
- ご注文時にユーザー全員の情報を提出する必要はなく、ライセンス管理者情報のみ提出が必要です
- ライセンス管理者がユーザーを登録します
- 1ライセンスにつき、シート数分のユーザーが使用できます
- 同ライセンスにシート数の追加可能
- Geneious クラウドストレージ容量:20GB(1シートあたり)
- クラウドワークスペースの共有可
パーソナルサブスクリプションおよびチームサブスクリプション共通
- ライセンス有効期間中であれば、アップグレードおよびアップデートは無料です。
- パーソナルサブスクリプションもチームサブスクリプションもユーザーを指定する必要があります。
- オンラインによるライセンス管理が可能です - ユーザー自身/ライセンス管理者自身でアクティベーション解除、ライセンス管理を行うことができます。
- インストール及びアクティベーションに関する弊社サポートの提供は、弊社より新規/更新を購入頂いた日より1年となります。ライセンスが有効な場合でも、新規/更新の購入日より1年以上が経過している場合はサポートのご利用はできません。
チームサブスクリプションのメリット
チームサブスクリプションでは、パーソナルライセンスにはないワークスペースの共有などの機能を利用できます。
1シートあたり 20GB のクラウドストレージを利用できます
無料で Geneious クラウドを利用できます。利用可能なストレージは 1シートあたり 20 GB(パーソナルサブスクリプションの場合は 10 GB)となっています。
Geneious クラウド ワークスペースの共有が可能です
Geneious クラウドのワークスペースをユーザー間で共有することができます。Geneious クラウドワークススペース機能の詳細についてはGeneiousクラウドをご参照ください。
ライセンス管理が簡単です
チームライセンスの場合、ライセンス管理者が My Account ページからシートの利用状況を把握できるようになっており、インストール状況の一元管理が可能です。パーソナルライセンスを複数購入した場合、誰が/どのコンピューターにインストールしているか包括的に把握するためには、別途エクセルなどで管理する必要があります。
割り当てたユーザーを変更できます
チームサブスクリプションはライセンス有効期間中ユーザーを変更することがでます。パーソナルサブスクリプションは、ライセンス有効期間中にユーザーを変更(ライセンス譲渡)することはできません。
【例】
チームサブスクリプションの場合:1ライセンス 2シートを購入し、AさんとBさんにシートを割り当てます。数ヶ月後にAさんが使用しなくなった場合、Cさんにシートを割り当てることができます。 パーソナルサブスクリプションの場合:2ライセンスを購入し、AさんとBさんが使用します(ご注文時に Aさん・Bさんをユーザーとして登録されます)。数ヶ月後に Aさんが使用しなくなった場合、ライセンスを譲渡することはできません。 - マシンベースドライセンス (Machine-based license) 、ユーザーベースドライセンス(User-based liccense) とは何ですか?
-
マシンベースドライセンスは、ライセンスキーを利用して使用するコンピューターを指定します。ユーザーベースドライセンスは、メールアドレスを利用して使用者を指定するライセンスです。マシンベースドライセンスとユーザーベースドライセンスは、それぞれのライセンスによってアクティベーションを行う方法が異なります。
ご注意ください
ユーザーベースドライセンスでは Geneious Prime 2024 以前のバージョンをご利用いただけません。旧バージョンの利用をご希望の場合は、その旨を注文備考としてご連絡下さい。Geneious Prime を既に導入しており、2024以前のバージョンの継続利用をご希望の場合は、ライセンスキーを明記頂き備考としてマシンベースドライセンス更新の旨を明記下さい。マシンベースド(Machine-based)ライセンス
My Account ページにライセンスキーが表示されている場合、マシンライセンスとなります。

アクティベーションにライセンスキーを使用します。
ユーザーベースド(User-based)ライセンス
My Account ページにライセンスキーが表示されていない場合、ネームドユーザーライセンスとなります。

アクティベーションにメールアドレスを使用します。

- Geneious は何台のコンピューターにインストールできますか?
-
パーソナルライセンス
1つのライセンスにつき、登録された個人ユーザーが所有するコンピューター2台にインストールすることができます(同時起動はできません)。
ユーザーベースドライセンス:30日に2回までコンピューターを変更することが可能です。
マシンベースドライセンス:ライセンス有効期間中(1年ごと)に2回までコンピューターを変更することが可能です。
Note:License Agreement 3.1 Personal License: A Personal License entitles a single Authorized User to install and use the Software on up to 2 computers running any of the supported operating systems, provided that only one copy of the Software is running at any given time and that the same License Key or User Credential is not used by anyone other than the Authorized User. The Authorized User may also access Subscription Services through the Personal License if supported in your geographical region.参照: Geneious Terms and Conditions | Effective 19 March 2024チームライセンス
1つのシートにつき、1名のユーザーのみが利用することを条件に、2台までにインストールして使用することが許諾されています(同時起動はできません*)。
1つのシートにつき、30日に2回までコンピューターを変更することが可能です。
*異なるシートであれば同時起動可能です。
Note:License Agreement 3.3 Team License: A Team License permits a specified Authorized User to install and use the Software on up to 2 computers running any of the supported operating systems, provided that only one copy of the Software is running at any given time and that the same User Credential is not used by anyone other than the specified Authorized User. The Authorized User may also access Subscription Services through the Team License if supported in your geographical region. - ライセンスの更新はどのように行いますか?
-
弊社にて販売している Geneious のサブスクリプションライセンスは自動更新ではないため、製品を継続してご利用になられる場合は1年毎に更新頂く必要があります。
継続利用(更新)を希望される場合は、ご注文の際に Geneious Prime のライセンスキーを入力下さい。
Geneious Prime 更新版 注文方法
更新版のご注文は有効期限の 60 日前より承っております。
Geneious Prime 価格ページより該当する商品の「購入」ボタンをクリックすると、オンラインショップの該当ページに移動します。新規/更新とも同一価格、同じ商品コードと商品名になります。
 オンラインショップの「送付先の入力」の注文備考で登録コードを入力ください
オンラインショップの「送付先の入力」の注文備考で登録コードを入力くださいご注意ください
My Accountページから更新することも可能ですが、その場合は開発元から直接の購入ということになり、弊社のサポート対象外となりますのでご注意下さい。 - Geneious グループライセンスの提供は終了したのでしょうか?
-
グループライセンスの新規での提供は、2024年3月21日を持って終了致しました。すでにグループサブスクリプションを導入頂いている(ライセンスキーのあるライセンスを利用中)場合、グループライセンスの更新/継続が可能です。
グループライセンスの使用条件について
- グループライセンスは、複数のユーザーがそれぞれのコンピューターで Geneious を使用することが可能です。
- 1つのライセンスキーにつき、購入頂いたシート数分をコンピューターにインストールしてご利用いただけます。
- コンピューターを共有して複数のユーザーが利用することも可能です。
- ネットワーク接続によるリモートアクセスはできません。
- 永続ライセンス(買切タイプ)の提供はありますか?
-
Geneious に永続ライセンスの提供はありません。すべて年次更新型のサブスクリプションライセンスとなります。
- 別のコンピューターにインストールしたいのですが、新規にライセンスの購入が必要ですか?
-
アクティベーション解除(ライセンスリリース)を行うことで、別のコンピューターにインストールして使用することができます。新たにライセンスを購入する必要はありません。
一定期間に利用可能なリリースの数は限られており、度重なるライセンスのリリースは、ユーザーが個人的なライセンスを他の人と共有しようとしていると認識される可能性があります。ライセンスのリリースは必要な場合のみ行って下さい。
参照:Geneious FAQ: My license has already been activated the maximum number of times - what can I do? - ライセンスキーはどのように確認しますか?
-
ライセンスキーは納品時に弊社より納品した登録確認書または My Account page (https://manage.geneious.com/auth/login) よりご確認頂けます。

Geneious Prime を2024年2月28日以降に新規でご購入頂いた場合はライセンスキーは発行されません。アクティベーションは、ライセンスキーではなくメールアドレスで行います。
- アクティベーション解除(ライセンスリリース)はどのように行いますか?
-
Geneious を使用する予定がなくなった場合は、使用を停止するコンピューターからライセンスをリリースする必要があります。ライセンスをリリースには以下の2つの方法があります。
1. Geneious を起動し、Helpメニューから Release License(s) を選択します。
2. My Account ページにログインし、Activations からアクティベーションの解除を行います。
ご注意ください
- コンピューターから Geneious をアンインストールしても、ライセンスはリリースされませんのでご注意ください。
- 一定期間に利用可能なリリースの数は限られており、度重なるライセンスのリリースは、ユーザーが個人的なライセンスを他の人と共有しようとしていると認識される可能性があります。ライセンスのリリースは必要な場合のみ行って下さい。
- 複数のシート数を所有していますが、1つのライセンスで異なるOSにインストールして利用できますか?
-
1つのライセンスで異なるOS(Win、Mac、Linux) にインストールしてご利用いただけます。 各OSのシステム条件は、 システム条件はどのようになっていますか?をご参照下さい。
参照:Geneious FAQ: Can I use my Geneious license on different operating systems? - ライセンスの有効期限はどのように確認しますか?
-
ライセンスの有効期限は My Account ページから確認できるほか、About Geneious Primeダイアログから確認することが可能です。
1. メニューバーの 「Geneious Prime」 から、「About Geneious Prime」 を選択します。
2. 有効期限は expires - 日 月 年 で表示されます。
ご注意ください ライセンス有効期限の開始日は「納品日」となります。アクティベーション実施日が開始日とはなりません。 終了日は「納品日」から 1 年後となります。
インストール
インストールの方法は、ユーザーベースドライセンス(メールアドレスによるアクティベーション)またはマシンベースドライセンス(ライセンスキーによるアクティベーション)により異なりますのでご注意ください。
- Geneiousのインストール方法 : メールアドレスによるアクティベーション
-
Geneious Prime 2024 以降、アクティベーションの方法が選択方式になりました。登録確認書にシリアル番号が記載されている場合、Geneiousのインストール方法 : ライセンスキーによるアクティベーションをご参照ください。
1. My Account ページよりインストーラーをダウンロードし、インストーラーを起動します。
Geneious Prime インストーラーは、開発元のウェブサイトからもダウンロード可能です。
Geneious Prime Download
https://www.geneious.com/downloadMy Account ページ
2. インストーラーを起動すると、 Welcome to Geneious Prime ダイアログが開きます。Geneious Prime がデータを保存するフォルダ(データベースフォルダ)を指定し、Activate a License ボタンをクリックします。
インストールダイアログではなく、制限付きモード*で Geneious Prime が起動した場合は、Helpメニューから "Activate License..." を選択します。
*ウィンドウの左下に "Features Restricted" が表示されます.
3. Activate License ダイアログが開きます。ダイアログで Email Sign Inを選択します。

4. Sign In ダイアログに登録メールアドレスを入力し、Continue ボタンをクリックします。
メールアドレスとパスワードは、「登録確認書」に記載があります。

5. Enter Password のフィールドにパスワードを入力します。パスワードが不明な場合は、 Forget your password? をクリックしてパスワードを再設定してください。

6. License Agreement (ライセンス規約) に同意頂けましたらチェックボックスにチェック頂き、Accept ボタンをクリックします。
Geneious ライセンスアグリーメント
http://assets.geneious.com/legal/eula.html
6. アクティベーションが完了すると You're All Set ダイアログが表示されます。 Start Using Geneious ボタンをクリックすると、Geneious が起動します。

- FLEXnetをインストールする必要がありますか?
-
Geneious Prime 2022.1.1より、FLEXnet のインストールは不要となりました。
2022.1.1以前のバージョン:
Geneiousをアクティベートするには、FLEXnet(ライセンス管理プログラム)がインストールされている必要があります。 通常は、Geneiousインストール時にインストールされます。Geneiousインストール時にFLEXnetがインストールされない場合は、Helpメニュー → Install FLEXnet を選択してFLEXnetを実行してください(FLEXnetの実行が必要な場合は、Geneiousの画面に表示されます)。
参照:Geneious FAQ: Do I need to install FLEXnet?
- Geneiousのインストール方法 : ライセンスキーによるアクティベーション
-
Geneious Prime 2024 以降、アクティベーションの方法が選択方式になりました。Geneious Prime 2024 以降は ステップ 2-1、Geneious Prime 2024 以前のバージョンはステップ 2-2 をご参照ください。
1. My Account ページよりインストーラーをダウンロードし、インストーラーを起動します。
Geneious Prime インストーラーは、開発元のウェブサイトからもダウンロード可能です。
Geneious Prime Download
https://www.geneious.com/downloadMy Account ページ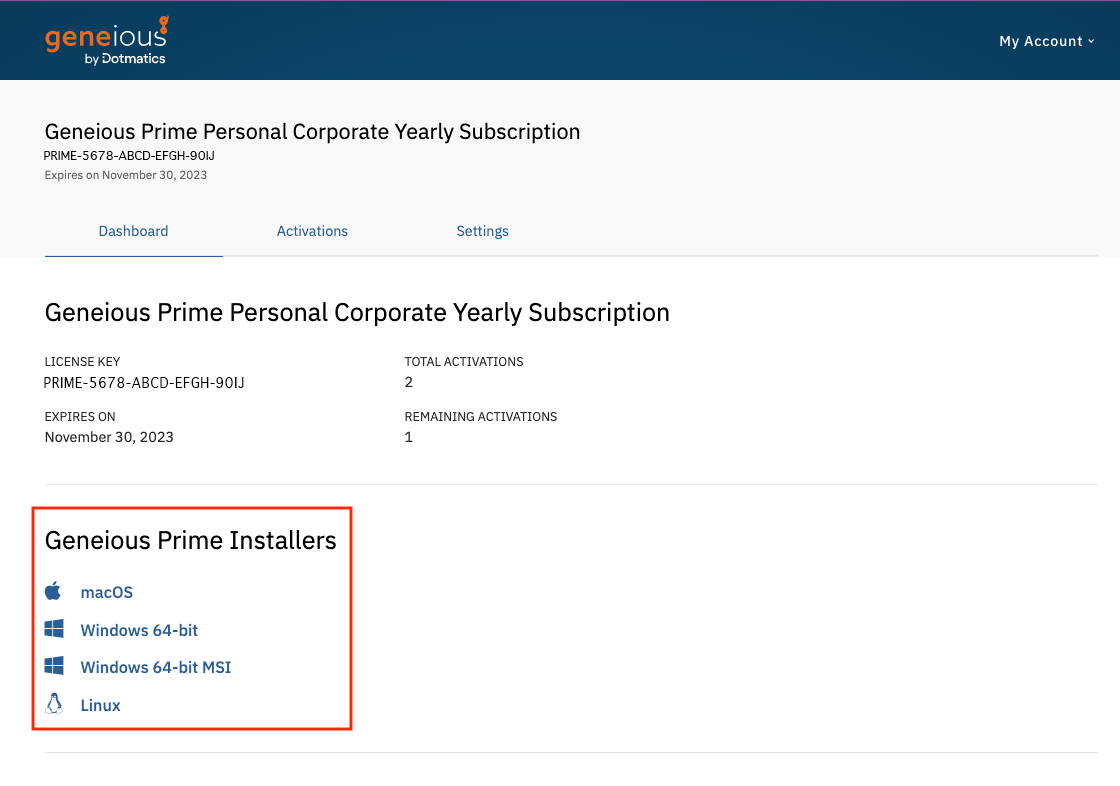
2-1. Geneious Prime 2024 以降
インストーラーを起動すると、 Welcome to Geneious Prime ダイアログが開きます。Geneious Prime がデータを保存するフォルダ(データベースフォルダ)を指定し、Activate a License ボタンをクリックします。
インストールダイアログではなく、制限付きモード*で Geneious Prime が起動する場合は、Helpメニューから "Activate License..." を選択します。
*ウィンドウの左下に "Features Restricted" が表示されます.Activate License ダイアログが開きます。ダイアログで License Key を選択します。


2-2. Geneious Prime 2024 以前
インストールが完了し、Welcome to Geneious Prime ダイアログが開きます。Geneious Prime がデータを保存するフォルダ(データベースフォルダ)を指定し*、Activate a License ボタンをクリックします。
*データベースフォルダの場所はインストール後に変更することも可能です。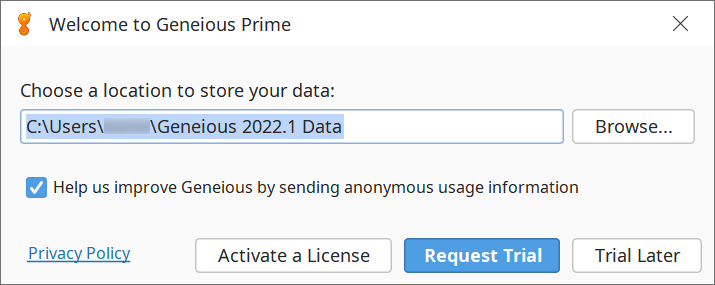
3. Enter your License Details ダイアログにEメールアドレスとライセンスキーを入力し、OKボタンをクリックすると Geneious が起動します。
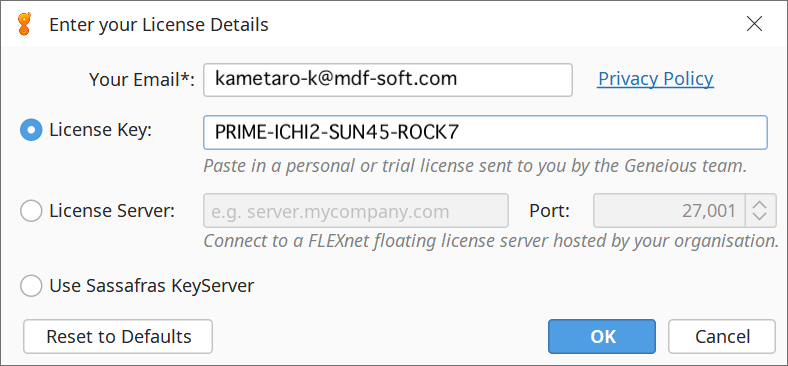
- Geneiousは何台のコンピューターにインストールできますか?
-
パーソナルライセンス
1つのライセンスにつき、登録された個人ユーザーが所有するコンピューター2台にインストールすることができます(同時起動はできません)。
ユーザーベースドライセンス:30日に2回までコンピューターを変更することが可能です。
マシンベースドライセンス:ライセンス有効期間中(1年ごと)に2回までコンピューターを変更することが可能です。
Note:License Agreement 3.1 Personal License: A Personal License entitles a single Authorized User to install and use the Software on up to 2 computers running any of the supported operating systems, provided that only one copy of the Software is running at any given time and that the same License Key or User Credential is not used by anyone other than the Authorized User. The Authorized User may also access Subscription Services through the Personal License if supported in your geographical region.参照: Geneious Terms and Conditions | Effective 19 March 2024チームライセンス
1つのシートにつき、1名のユーザーのみが利用することを条件に、2台までにインストールして使用することが許諾されています(同時起動はできません*)。
1つのシートにつき、30日に2回までコンピューターを変更することが可能です。
*異なるシートであれば同時起動可能です。
Note:License Agreement 3.3 Team License: A Team License permits a specified Authorized User to install and use the Software on up to 2 computers running any of the supported operating systems, provided that only one copy of the Software is running at any given time and that the same User Credential is not used by anyone other than the specified Authorized User. The Authorized User may also access Subscription Services through the Team License if supported in your geographical region. - ライセンスキーはどのように確認しますか?
-
ライセンスキーは納品時に弊社より納品した登録確認書またはMy Account page (https://manage.geneious.com/auth/login)よりご確認頂けます。

- アクティベーション解除(ライセンスリリース)はどのように行いますか?
-
Geneious を使用する予定がなくなった場合は、使用を停止するコンピューターのアクティベーションを解除する必要があります。アクティベーション解除には2つの方法があります。
My Account ページにログインして行う
My Account ページにログインします。ユーザーベースドライセンは My Account ページの Seats タブから、マシンベースドライセンスは Activation タブから行います。
ご注意くださいGeneious Prime を起動してコンピューターから行う
Helpメニューから Release License(s) を選択し、ダイアログに表示される Release License ボタンをクリックします。
- コンピューターからGeneiousをアンインストールしても、ライセンスはリリースされませんのでご注意ください。
- 一定期間に利用可能なアクティベーション解除の回数は限られています。ライセンスのリリースは必要な場合のみ行って下さい。
パーソナルライセンス
ユーザーベースドライセンスは30日ごとに2回、マシンベースドライセンスは1年ごとに2回のアクティベーション解除が可能です。
チームライセンス
ライセンス有効期間中(1年毎)1シートにつき、30日ごとに2回のアクティベーション解除が可能です。
グループライセンス
ライセンス有効期間中(1年毎)、シート数分のアクティベーション解除が可能です。
アップグレード/アップデート
- Geneious をアップグレード/アップデートしても、データを失うことはありませんか?
-
データは自動的に新しいバージョンに移行され、アップグレード/アップデート前と変わらずご利用頂けます。
新しいバージョンのGeneious の初回起動時に、旧バージョンのバックアップを保持するかどうか尋ねられますが、アップグレードする前に、Geneious のすべてのデータのバックアップを別途取ることをお勧めします。
新バージョンを初めて開いたときに、何らかの理由で旧バージョンのデータフォルダが見つからない場合は、「Tool」→「Preferences」→「General」で、 Data Storage Location でデータの保存場所を旧バージョンのデータフォルダに設定してください。
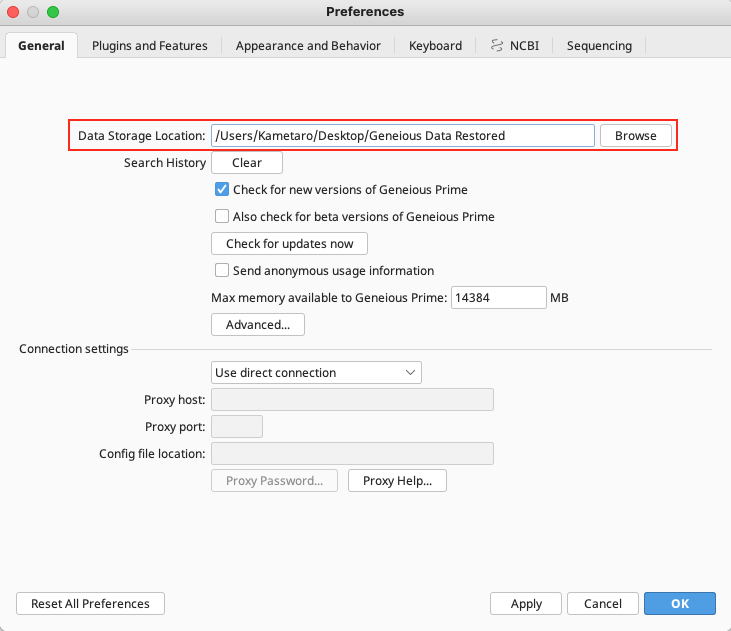
- アップデートをインストールしようとすると、エラーが表示されてアップデートできないのですが。
-
アップデーター(Helpメニュー → Check for updates) から、アップデートできない場合は、製品インストーラー をダウンロード頂き、新しいバージョンをインストール下さい。
製品インストーラーは My Account ページ、または、 Biomatters 社の Geneious Prime Download ページよりダウンロード頂けます。
エラーメッセージ例
- 旧バージョンにダウングレードすることはできますか?
-
Geneious のライセンスを新しいバージョンにアップデートした場合、以前のバージョンにダウングレードすることができます。ダウングレードする場合、Geneiousの新しいバージョンをアンインストールし、以前使用していた Geneious のバージョンを再インストールする必要があります。旧バージョンのインストーラーは、www.geneious.com/previous-versionsよりダウンロード頂けます。
ご注意ください- ユーザーベースドライセンス(メールアドレスを利用してアクティベーションを行うライセンス)をご利用の場合、Geneious 2024 以前のバージョンを利用することはできません。
- Geneiousのデータは通常、Geneiousがインストールされている場所とは別の場所に保存されています。Geneiousをアンインストールしてもデータは削除されませんが、アンインストールする際は、Geneiousのバックアップ機能を使ってデータベースのバックアップを作成しておくことをお勧めします。
- ダウングレード後に、旧バージョンのGeneiousを再起動すると、自動的に以前のデータベースが認識されるようになっています。Geneiousが自動的にこのデータベースを認識しない場合は、「Tool」→「Preferences」→「General」で、 Data Storage Location でデータフォルダを指定して下さい。
Note:
Geneiousが自動的に旧バージョンのデータベースを選択しない場合は、手動で該当するフォルダを指定します。手動でフォルダを指定する方法は、「データベースフォルダの場所を変更した場合、どのようにフォルダの場所を指定しますか」をご参照下さい。
参照:Geneious FAQ: Downgrading to an earlier version of Geneious
ファイル
- Geneiousに対応しているファイルのフォーマットはどのようなものがありますか?
-
Geneiousでインポート可能なファイル形式は以下の通りです。
Format Extensions Data Types Common Sources BED *.bed Annotations UCSC Common Assembly Format *.caf Contigs Sequencher Clone Manager *.cm5 Nucleotide sequences Clone Manager Clustal *.aln Alignments ClustalX CS FASTA *.csfasta Color space FASTA ABI SOLiD DNAStar *.seq, *.pro Nucleotide & protein sequences DNAStar DNA Strider *.str Sequences DNA Strider Embl/UniProt *.embl, *.swp Sequences Embl, UniProt Endnote (8.0) XML *.xml Journal article references Endnote, Journal article websites FASTA *.fasta, *.fas, etc. Sequences, alignments PAUP*, ClustalX, BLAST, FASTA FASTQ *.fastq, *.fasq Sequences with quality Solexa/Illumina GCG *.seq Sequences GCG, GCG codon usage table *.cod Codon usage table GCG CodonFrequency tool GenBank *.gb, *.xml Nucleotide & protein sequences GenBank Geneious *.xml, *.geneious Preferences, databases Geneious Geneious Education *.tutorial.zip Tutorial, assignment etc. Geneious GFF, GFF3, GTF *.gff, *.gff3, *.gtf Annotations NCBI, Ensembl and other genome browser MEGA *.meg Alignments MEGA Molecular structure *.pdb, *.mol, *.xyz, *.cml, *.gpr, *.hin, *.nwo 3D molecular structures 3D structure databases and programs Newick *.tre, *.tree, etc. Phylogenetic trees PHYLIP, Tree-Puzzle, PAUP*, ClustalX Nexus *.nxs, *.nex Trees, Alignments PAUP*, Mesquite, MrBayes & MacClade PDB *.pdb 3D Protein structures SP3, SP2, SPARKS, Protein Data Bank PDF *.pdf Documents, presentations Adobe Writer, LATEX, Miktex Phrap ACE *.ace Contig assemblies Phrap/Consed PileUp *.msf Alignments pileup (gcg) PIR/NBRF *.pir Sequences, alignments NBRF PIR Qual *.qual Quality file Associated with a FASTA file Raw sequence text *.seq Sequences Any file that contains only a sequence Rich Sequence Format *.rsf Sequences, alignments GCGs NetFetch Comma/Tab Separated Values *.csv, *.tsv Spreadsheet files Microsoft Excel SAM/BAM *.sam, *.bam Contigs SAMtools Sequence Chromatograms *.ab1, *.scf Raw sequencing trace & sequence Sequencing machines SnapGene *.dna, *.prot Sequences, annotations, metadata Geneious Test/html *.txt, *.rtf, *.html Any text Simple text editors Variant Call Format *.vcf Variant calls/SNPs Variant callers and databases Vector NTI sequence *.gb, *.gp Nucleotide & protein sequences Vector NTI Vector NTI/AlignX alignment *.apr Alignments Vector NTI, AlignX Vector NTI Archive *.ma4, *.pa4, *.oa4, *.ea4, *.ca6 Nucleotide & protein sequences, enzyme sets and publications Vector NTI Vector NTI/ContigExpress *.cep Nucleotide sequence assemblies Vector NTI Vector NTI database VNTI Database Nucleotide & protein sequences, enzyme sets and publications Vector NTI Geneiousでエクスポート可能なファイル形式は以下の通りです。
参照: Import and Export Sequence DataData Type Format DNA sequence FASTA, Genbank XML, Genbank flat, Geneious Amino acid sequence FASTA, Genbank XML, Genbank flat, Geneious Chromatogram sequence ABI, Geneious Sequence with quality FastQ, Qual, Geneious Annotation GFF, BED, VCF, Geneious Multiple sequence alignment Phylip, FASTA, NEXUS, MEGA3, Geneious Assembly Phrap ACE, Geneious, SAM/BAM, Geneious Phylogenetic tree Phylip, FASTA, NEXUS, Newick, MEGA3, Geneious PDF document PDF, Geneious Publication EndNote 8.0, Geneious Graphs CSV or WIG, or images - サブスクリプションライセンスの更新をしない場合、ファイルは開けなくなりますか?
-
サブスクリプションライセンスの有効期限が切れた場合、Geneiousは制限付きモードとなります。 制限付きモードで利用できる機能は、自身が作成したデータの閲覧、インポート及びエクスポートのみとなります。
ご注意ください
ライセンス有効期限の開始日は「納品日」となります。アクティベーション実施日が開始日とはなりません。 終了日は「納品日」から 1 年後となります。
インポート & エクスポート
- シークエンスやアライメントを、どのようにテキストフォーマットでエクスポートしますか?
-
Geneious Prime 2019.1以降 Text Viewタブで、配列やアラインメントをカスタマイズ可能なプレーンテキストで表示することができるようになりました。エクスポートやコピーが可能なほか、テキストエディタやMicrosoft Wordドキュメントにコピー&ペーストできるリッチ(フォーマットされた)テキスト形式でも表示することができます。
シングルシークエンス
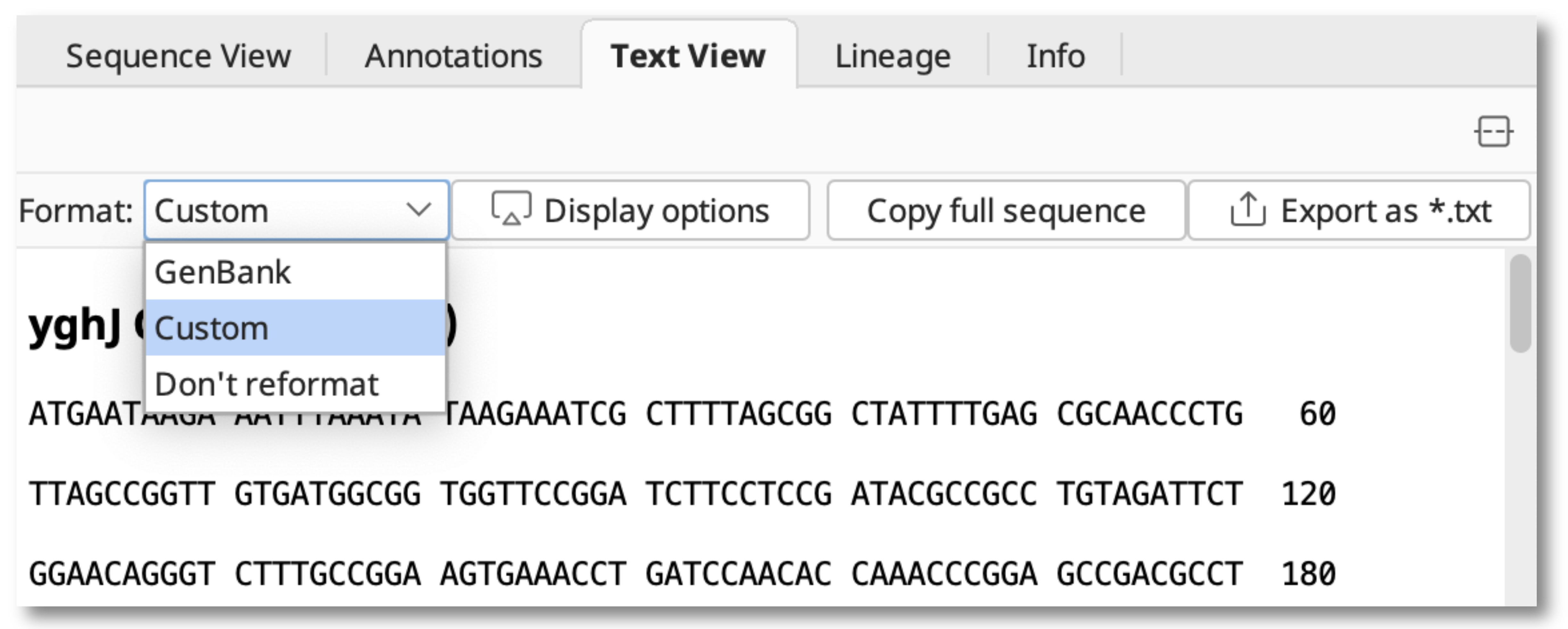
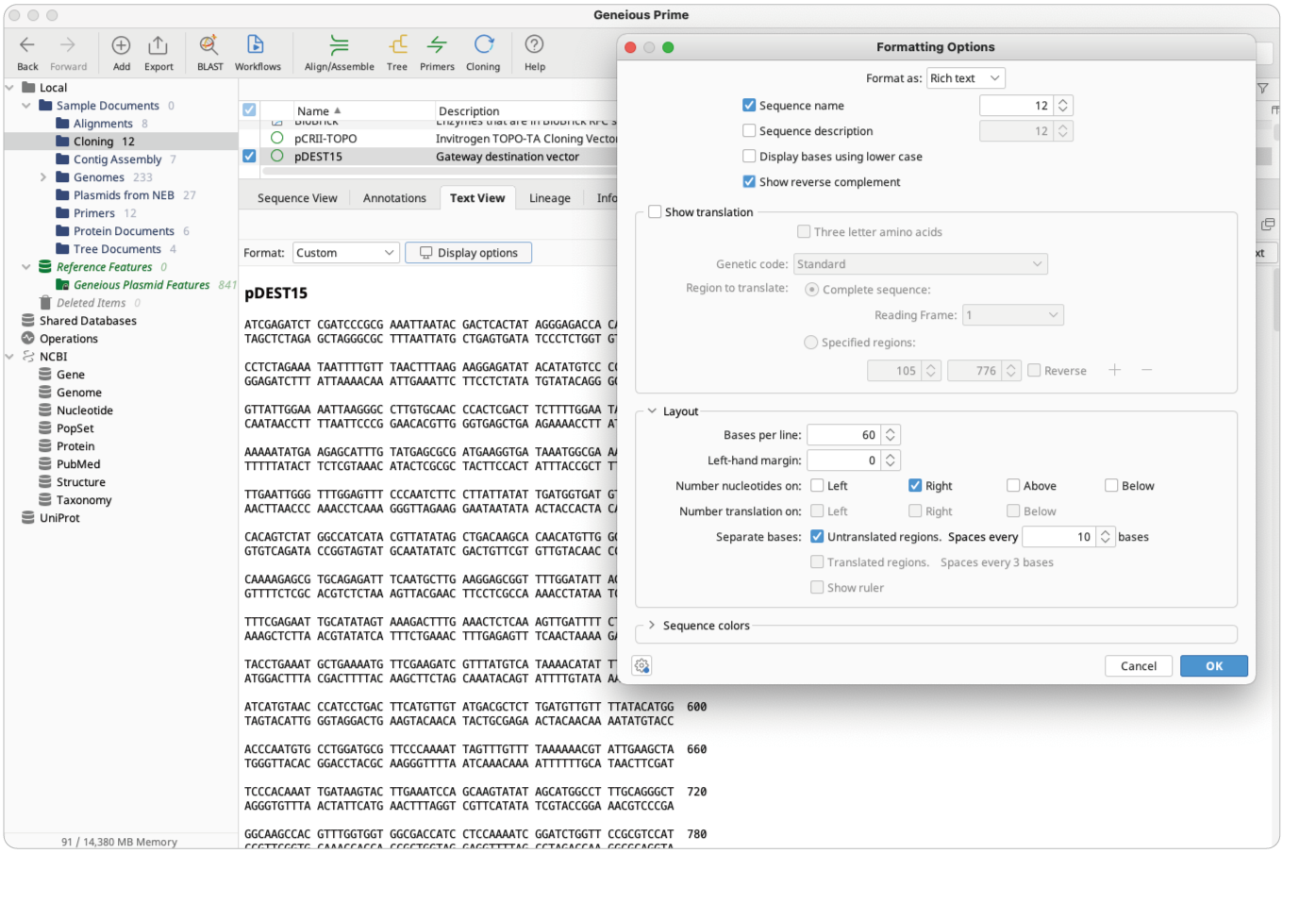
同じ種類(DNAまたはタンパク質)の配列または配列リストを選択した状態で、Text Viewタブに切り替えます。
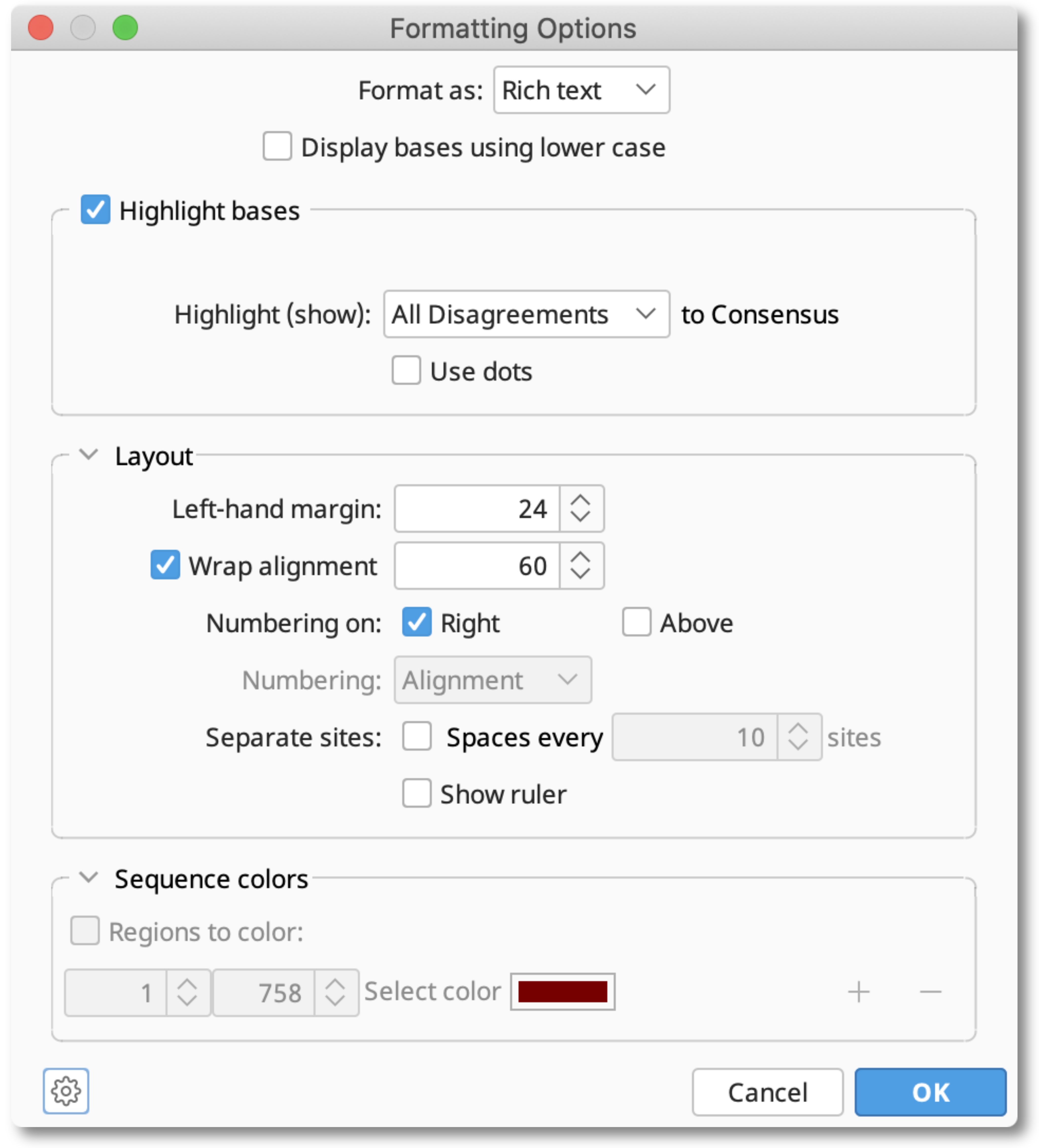
Formatドロップダウンメニューから Custom を選択します。
Display options ボタンを押すと Formatting Options ダイアログが表示されます。
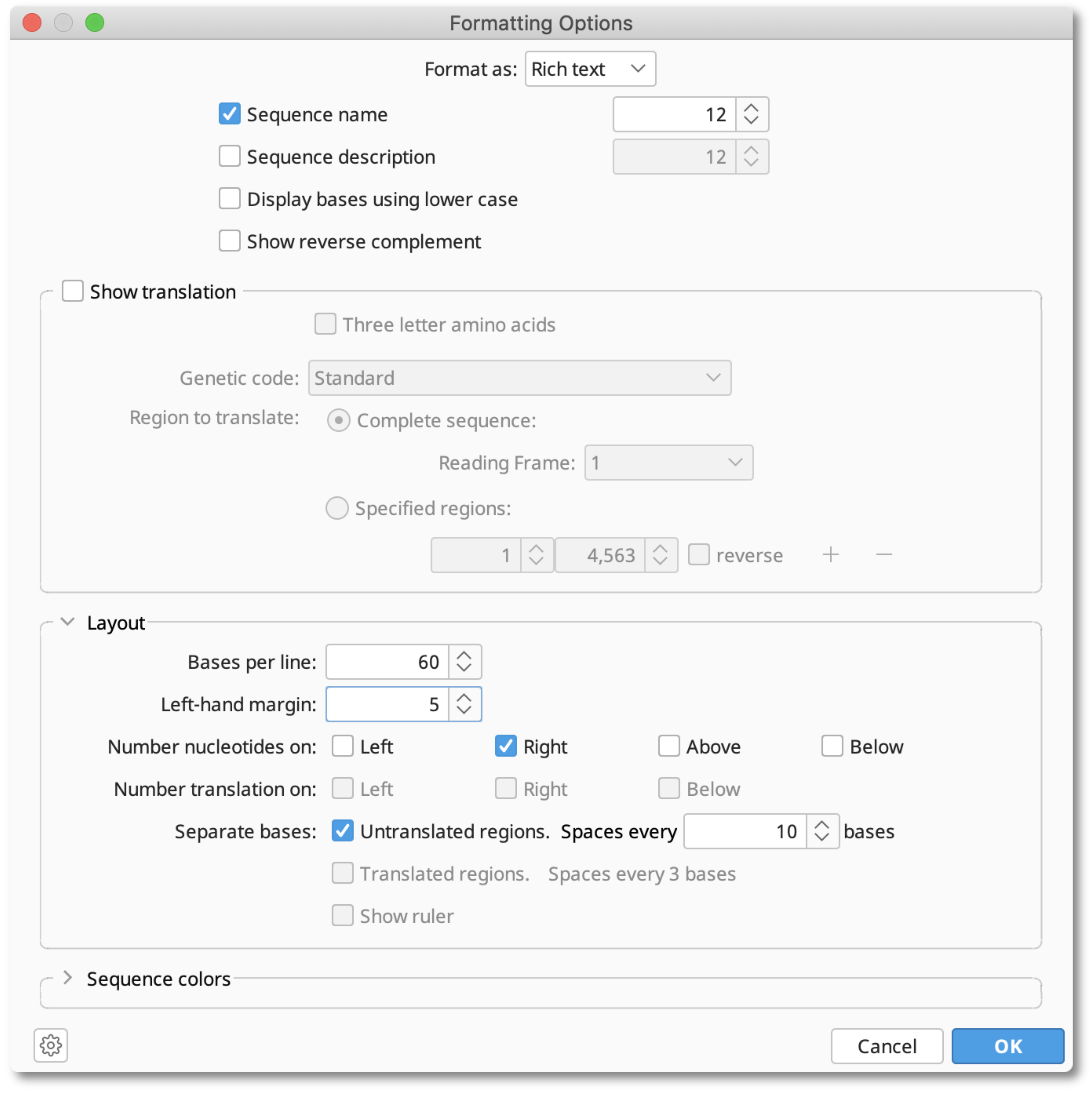
Format as のドロップダウンメニューから、プレーンテキストで表示するか、リッチテキスト形式で表示するかを選択します。
また、配列名と説明文の表示、塩基の大文字(デフォルト)と小文字の表示を選択できます。 相補的な逆鎖を表示するには、Show reverse complement にチェックを入れます。
Show Translationセクションから、特定のフレームを完全な配列で表示、または複数の範囲の翻訳を表示を選択ことが可能です。
Layoutセクションでは、Bases per lineの設定、ナンバリング/ナンバリングポジションの設定、マージン幅、ルーラーの設定ができます。オプションの Separate bases にチェックして定義すると、シークエンスをブロックに分けることが可能です。
リッチテキスト形式を選択した場合は、Sequence colors オプションを使用して、シークエンスまたはアラインメントの1つまたは複数の領域を色で強調することもできます。
Note:
Formatting Optionsダイアログの選択内容が、プレビューのようにText Viewに反映されます。
アライメント
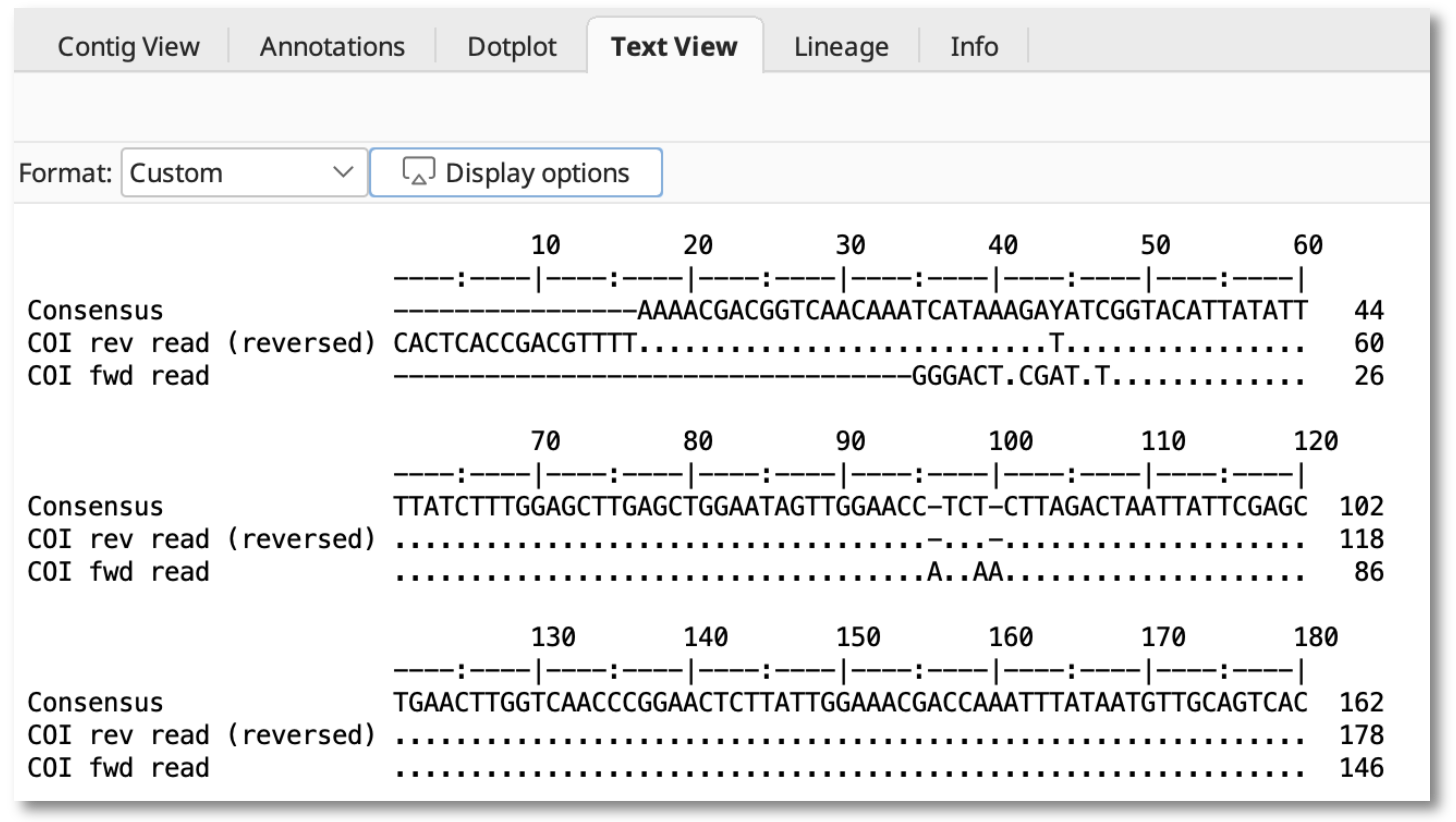
アラインメントファイルを1つ選択し、Text Viewタブをクリックします。
Formatドロップダウンメニューから Custom を選択します。
Highlight basesセクションでは、Consensusに対する不一致(disagreements)、合意(agreements)、類似性(similarities)、非類似性(dissimilarities)の表示を選択できます。
また、シングルシークエンスの場合と同様にLayoutセクションやSequence Colorsセクションの設定を行うことができます。
Layoutセクションでは、Bases per lineの設定、ナンバリング/ナンバリングポジションの設定、マージン幅、ルーラーの設定ができます。オプションの Separate bases にチェックして定義すると、シークエンスをブロックに分けることが可能です。
リッチテキスト形式を選択した場合は、Sequence colors オプションを使用して、シークエンスまたはアラインメントの1つまたは複数の領域を色で強調することもできます。
エクスポート (Text View タブ)
リッチテキスト(カラーの書式付き)の場合は、
 ボタンをクリックし、表示されているテキストをクリップボードにコピーします。
ボタンをクリックし、表示されているテキストをクリップボードにコピーします。プレーンテキスト(書式なし)の場合は、
参照:Geneious FAQ: How do I import metadata from a spreadsheet onto existing Geneious documents? ボタンを使用します。表示されているテキストが、.tx形式でエクスポートされます。カラー書式による表示は反映されませんのでご注意下さい。
ボタンを使用します。表示されているテキストが、.tx形式でエクスポートされます。カラー書式による表示は反映されませんのでご注意下さい。 - メタデータをスプレッドシートから Geneious にインポートできますか?
-
スプレッドシートを利用したメタデータを、.csvまたは .tsv形式のデータをGeneiousにインポートしてドキュメントに追加することができます(Geneious Prime 2020.1以前のバージョンには対応していません)。
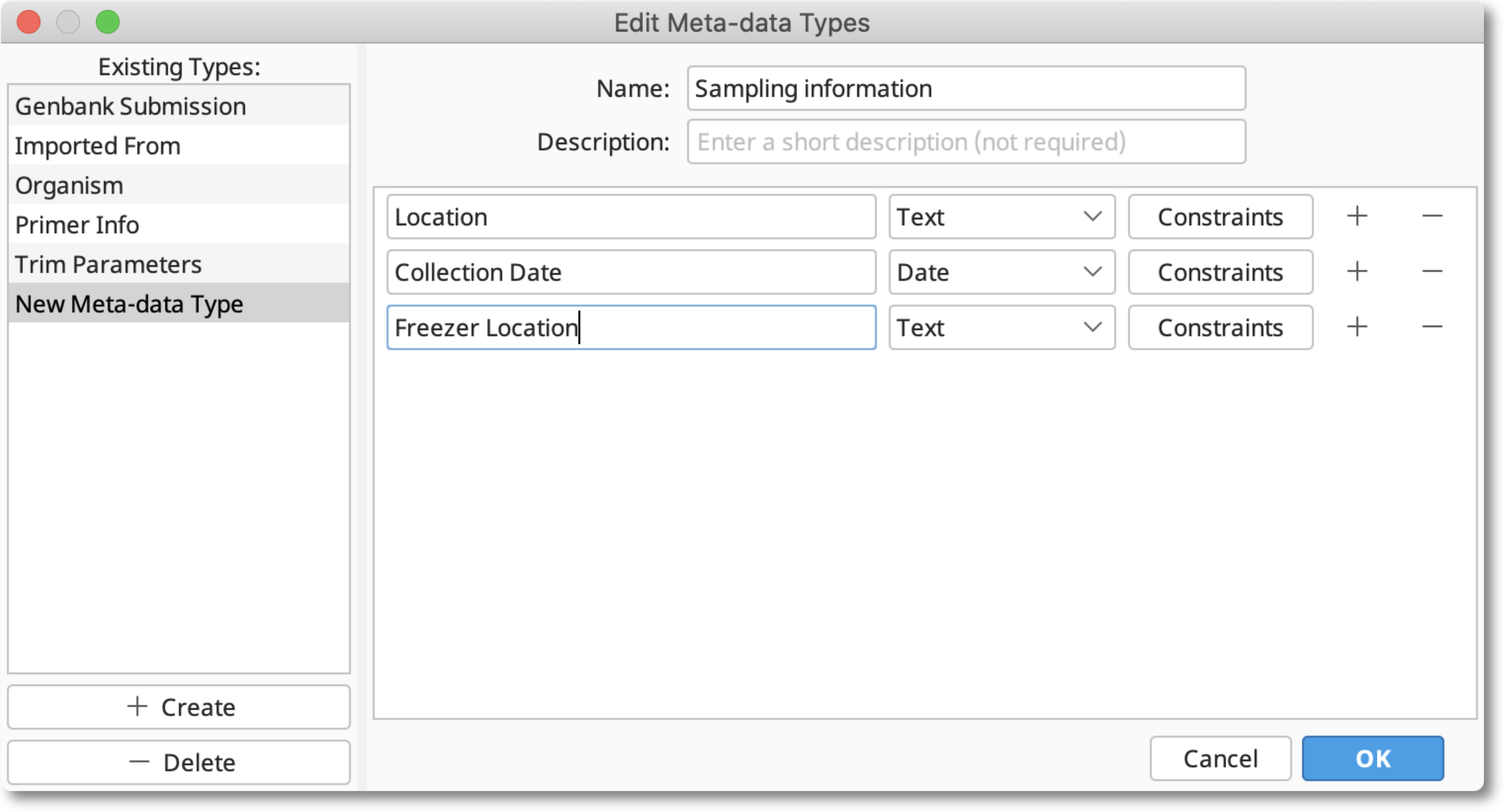
メタデータのファイルには、Geneiousが正しいドキュメントにデータを照合できるように、名前やシークエンスIDのフィールドなど、データをインポートしたいドキュメントと共通のフィールドが求められます。そのため、ファイルをインポートする前に、スプレッドシートから追加する予定のメタデータフィールドをGeneiousに設定する必要があります。
データをインポートする前に、メタデータフィールドを設定します。
どのフィールドが存在するかを確認したり、新しいフィールドを追加するには、任意のドキュメントを選択して、[Info]タブに移動します。
 をクリックすると、現在Geneiousで利用可能なフィールドの一覧が表示されます。
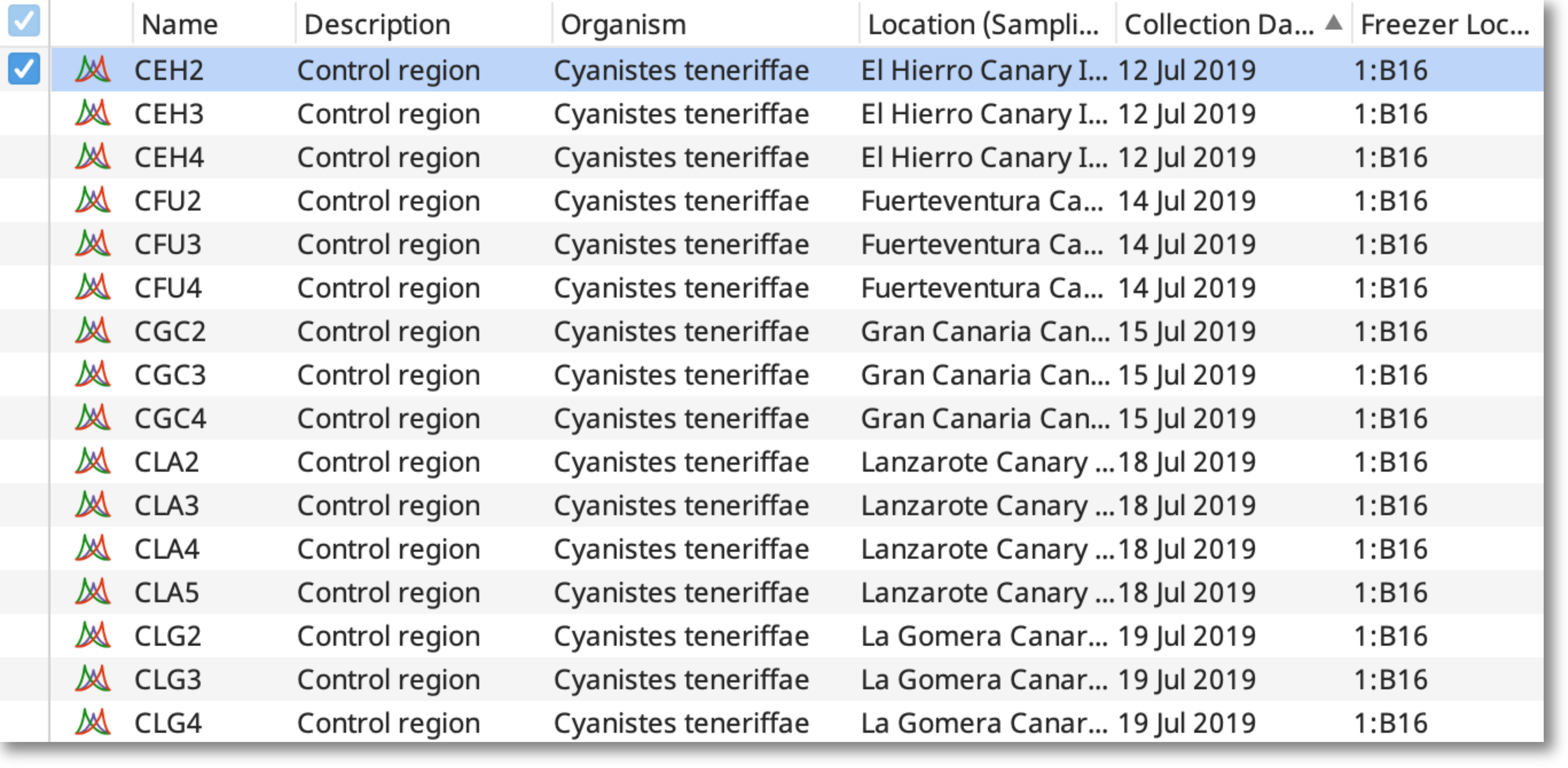
をクリックすると、現在Geneiousで利用可能なフィールドの一覧が表示されます。 ここに示す例では、スプレッドシートには、シークエンスされたサンプルのサンプリング場所(Location)、サンプリング日(Collection Date)、冷凍庫の保管場所(Freezer Location) の情報が含まれています。 下のスクリーンショットでは、"Sampling information "という新しいメタデータタイプの下に、これらのスプレッドシートのカラムのメタデータフィールドを追加しています。
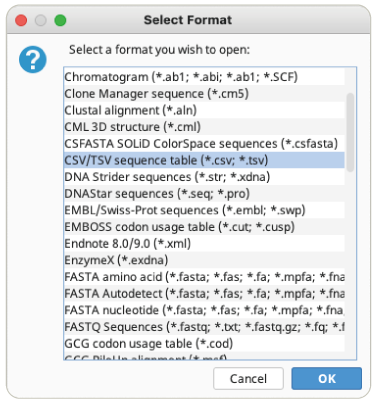
メタデータを追加したいドキュメントを含むフォルダを選択します。次に、メニューバーのFile の Import から、Filesをクリックして Select Formatダイアログを表示します。
Select Formatダイアログから、フォーマットとしてCSV/TSVを選択し、メタデータのファイルを選択します。
Note:
データのインポートは、CSVファイルをフォルダにドラッグ&ドロップすることもできます。
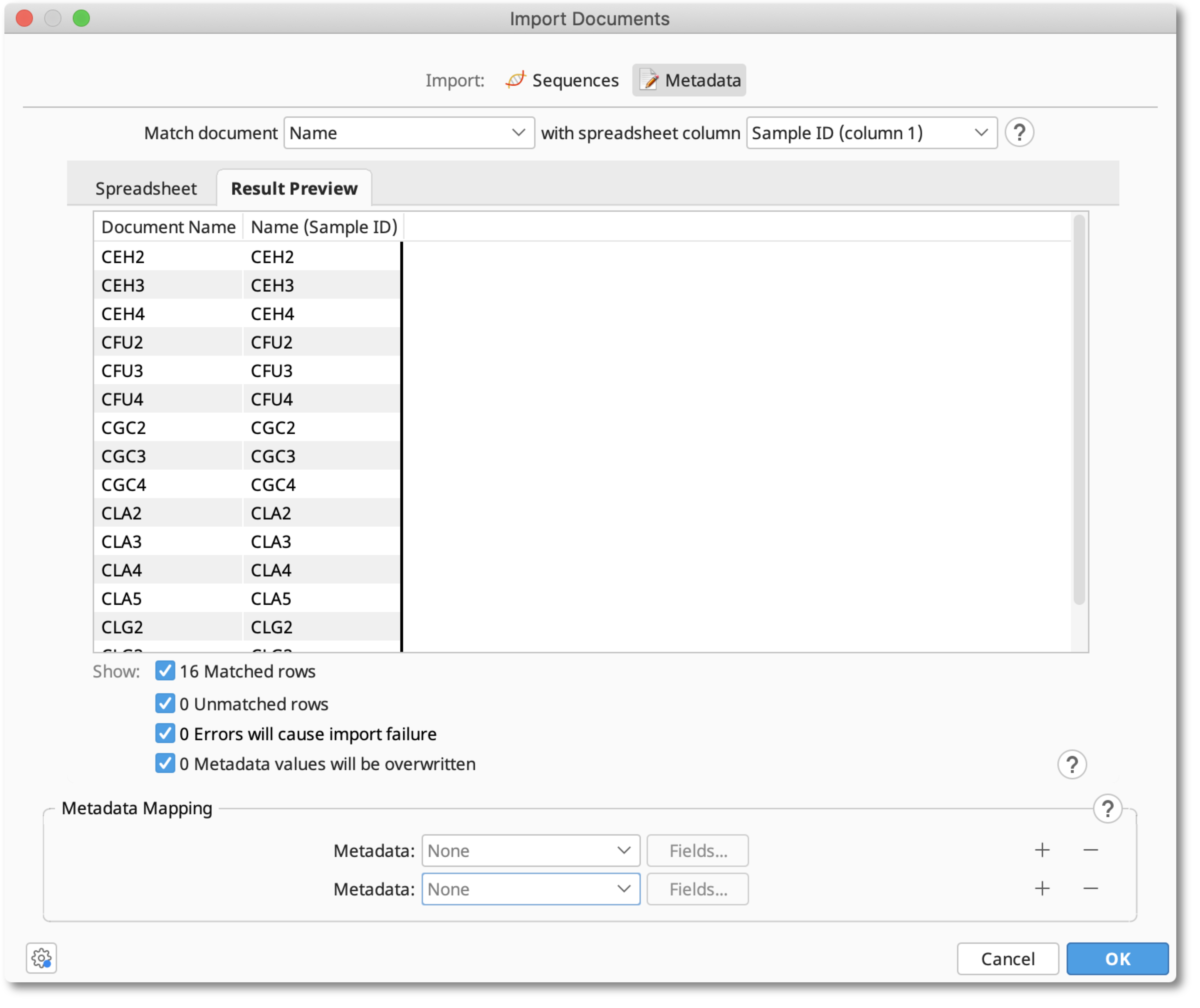
Import Documentsダイアログで、インポートするドキュメントの種類として Metadata をクリックします。
Match document では、Geneiousドキュメントやスプレッドシートファイルの中で、一致するデータを含むフィールドを選択します。
この例では、Geneiousのドキュメント名とスプレッドシートのSample IDカラムが一致しています。 一致したエントリは、下図のように Result Preview タブから確認できます。
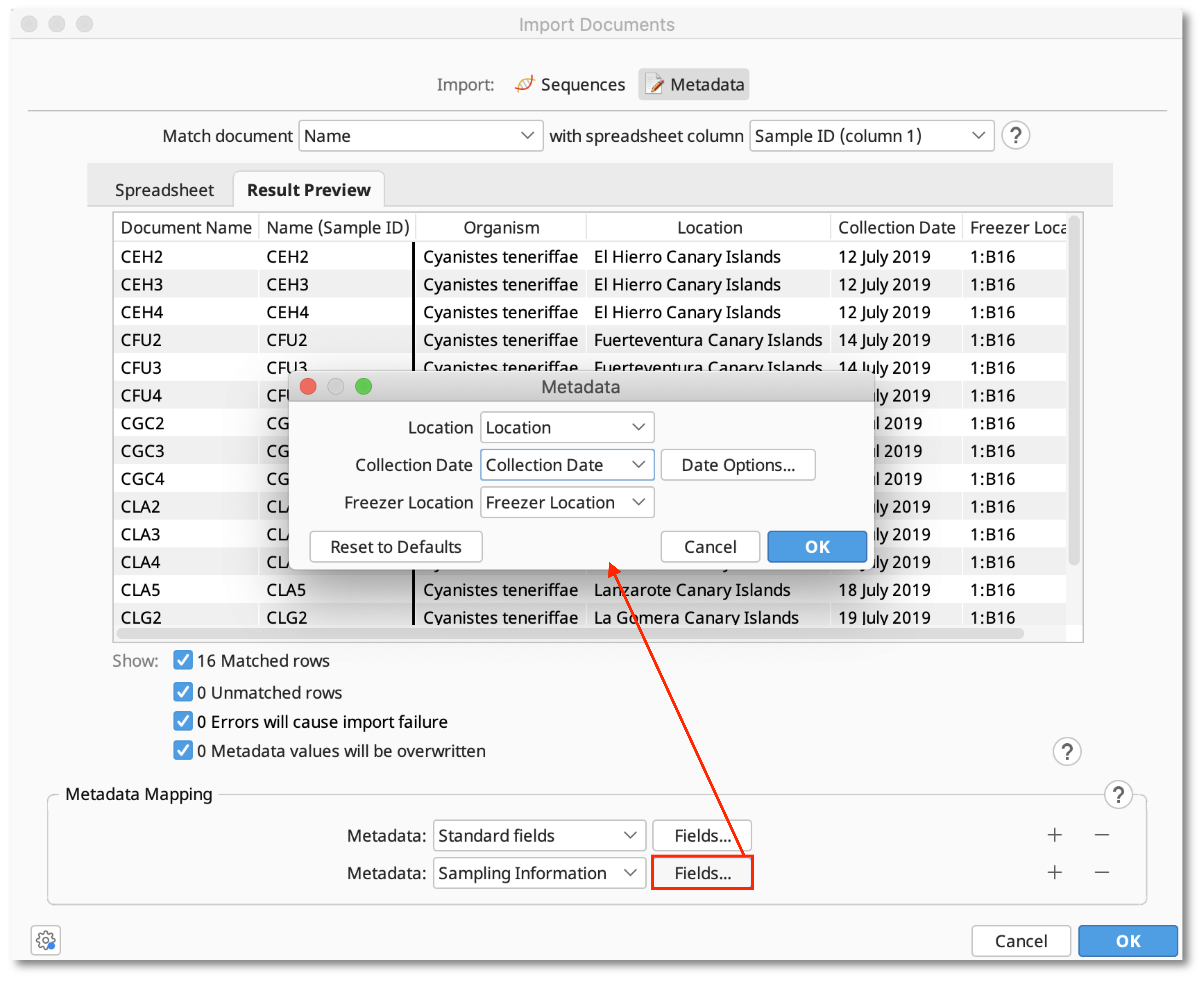
次に、Metadata Mappingボックスで、Geneiousドキュメント上でメタデータ情報を追加したいフィールドを選択します。
Metadata のドロップダウンメニューには、自分で追加したものも含めて、現在データベースで利用可能なすべてのメタデータタイプが表示されます。
Fields ボタンをクリックして、各メタデータタイプのフィールドをスプレッドシートの列にマッピングします。
図例を見ると、Sampling Informationメタデータタイプの下にSampling Location、Sampling Date、Freezer Locationのスプレッドシートの列が追加され、Standard Fieldsタイプの下にOrganismの列が追加されているのが分かります。
参照:Geneious FAQ: How do I import metadata from a spreadsheet onto existing Geneious documents?Result Previewタブには、Geneiousドキュメントにインポートされるフィールドのプレビューが表示され、エラーやGeneiousフォルダに一致するドキュメントがないスプレッドシートの行も表示されます。
インポート内容に問題がなければ、[OK]をクリックしてメタデータを読み込みます。 ドキュメントテーブルにメタデータが表示されます(新しい列を表示するには、テーブルの右側にスクロールする必要があります)。
- 印刷に適したクオリティの画像をエクスポートするには?
-
印刷する場合、拡大しても画質が損なわれないベクターフォーマット(PDF、EPS、SVG)での出力をお勧めします。 これらのファイル形式を指定してエクスポートを行います。
1. 画像をエクスポートするには、ツールバーの Exportボタンから、Export to Image を選択します。
1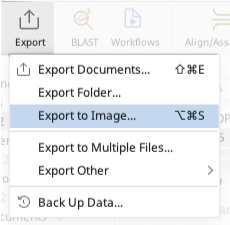
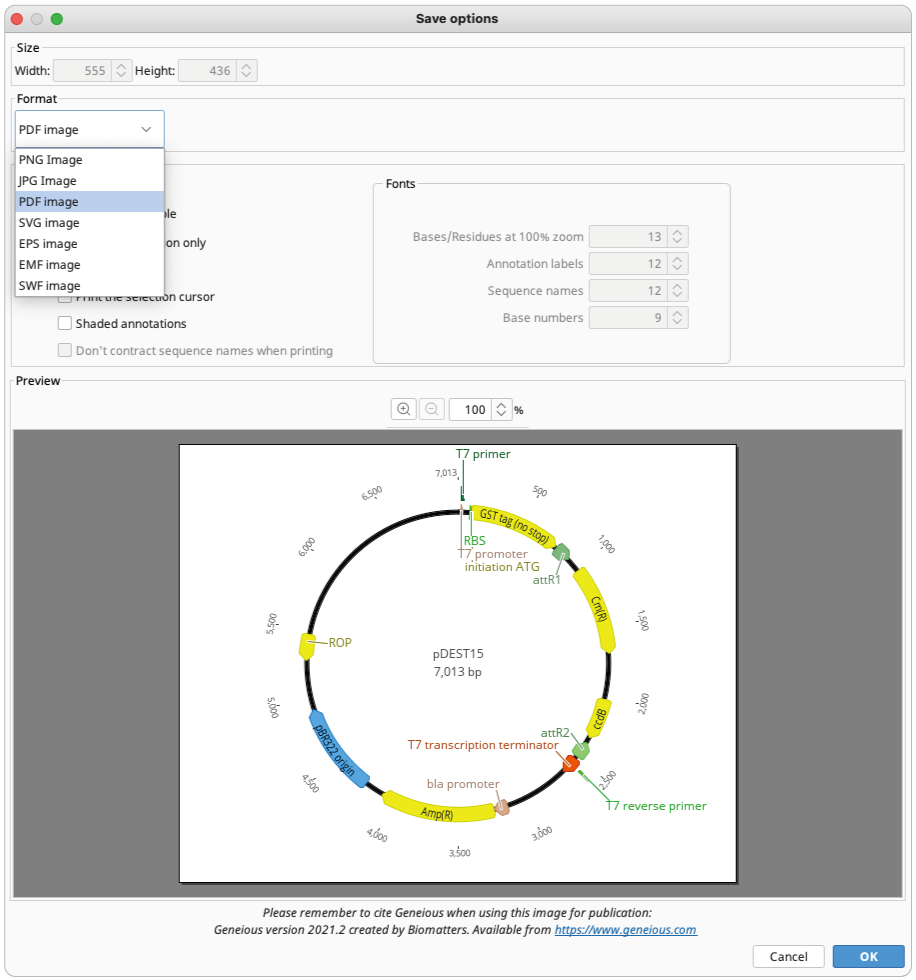
2. Save Options ダイアログで、エクスポートするファイルのフォーマットを選択します。
Note:対応しているファイル形式は以下の通りです。
.png, .jpg, .pdf, .svg, .eps, .emf, .swf2参照:Geneious FAQ: How do I export publication-quality images?
- Dotmatics Bioregister にシークエンスを登録するには?
-
Dotmatics Bioregister にシークエンスを登録するに当たって、事前にプラグインをインストール頂き設定を頂く必要がございます。プラグインは下記よりダウンロードください。
https://www.geneious.com/plugins/dotmatics-platform-integration/
プラグインのダウンロードが完了したら、メニューバーの 「Tools」 から、「Plugins...」 を選択します。

1 Install plugin from a gplugin file... ボタンをクリックし、ダウンロードしたプラグイン(DotmaticsPlatformIntegration.gplugin ) をインストールします。
2 Installed Plugins にプラグインがインストールされているのを確認後、OK ボタンをクリックします。


ツールバーの Geneious から Preference を選択し、Dotmatics Integration タブを開きます。Dotmatics Bioregister Server にパスを入力し、 OK ボタンをクリックすると Geneious と Dotmatics Bioregister の接続が完了します。

Dotmatics Bioregister に登録するファイルを選択し、右クリックメニューから Register を選択します。または、ツールバーの Register ツールバーの Register ボタンをクリックします。

初回のアップロード時に Dotmatics Bioregister の username と password を入力する必要があります。username と password を入力し、login ボタンをクリックします。

シークエンスの エンティティタイプ、サブタイプをドロップダウンメニューから選び、Create ボタンをクリックします。Keep me logged in にチェックを入れると、セッションが切れるまでログイン状態を保ちます。

登録ダイアログで登録情報を設定し、 Upload をクリックします。
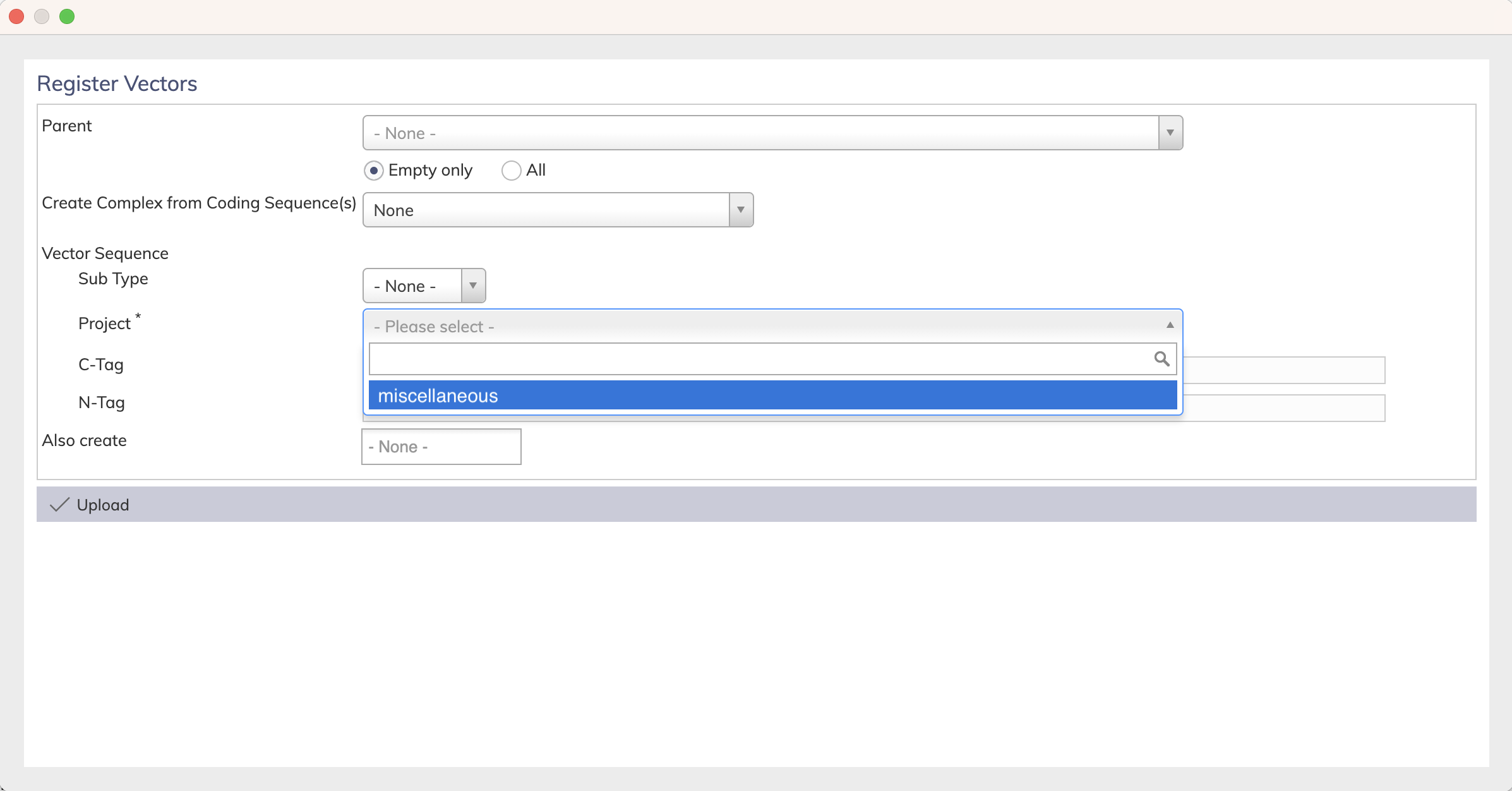
登録が完了すると、 Entity Registered ダイアログ に Bioreg ID が表示されます。
参照:Geneious Knowledge Base: Register sequences in Dotmatics Bioregister
操作方法
- データベースフォルダとは何ですか?
-
データベースフォルダには、Geneious のデータや設定内容がすべて保存されています。
データのバックアップもデータベースフォルダが利用されます。
Note:データベースフォルダの場所は Preferences の General タブにある Data Strage Location: で確認できます。

- データベースフォルダの場所を変更した場合、どのようにフォルダの場所を指定しますか?
-
データベースフォルダの場所の指定は、Preferences ダイアログの General タブから行います。
メニューバーの Tools から Preferences を選択し、Preferences ダイアログを開きます。
General タブの Data Strage Location: のフィールドに該当するデータベースフォルダのパスを入力するか、Browse ボタンを使用してフォルダを指定します。
フォルダの場所を指定したら、OKボタンをクリックします。

- RNA-Seqデータに対応していますか?
-
Geneious RNA assemblerを使用して、RNA-seqリードをゲノム参照配列にマッピングすることができます。 このアセンブラは、新規のイントロンを発見し、リードの末端をこれらの新規のイントロンの周辺に正しくマッピングし、参照配列上のCDS、mRNA、junctionアノテーションを介してリードをイントロンにマッピングすることも可能です。また、RNA-seqマッピングツールのTophatプラグインが用意されています。
トランスクリプトームのde-novoアセンブリには、de-novoアセンブリのオプションで利用できるSPAdesの使用をお勧めします。 これをRNAモードで実行するには、データソースとしてRNAを選択します。 SpadesRNAの詳細については、こちらの論文:rnaSPAdes: a de novo transcriptome assembler and its application to RNA-Seq data(Oxford University Press サイトに移動します)を参照してください。 Geneiousアセンブラーをde novoトランスクリプトームアセンブリーに使用することはお勧めしません。
Geneious R8以降から、デジタル遺伝子発現解析のためのツールが含まれています。個々のサンプルの参照マッピングに対して、正規化された発現指標であるTPM値、RPKM値、FPKM値を計算したり、2つのサンプル間で値を比較して、異なる発現遺伝子を見つけることができます。
個々のサンプルのTPM値、RPKM値、FPKM値を計算するには、リファレンスアセンブリを選択し、「Annotate and Predict」メニューの「Calculate Expression Levels」に進みます。結果は、ヒートマップアノテーショントラックとしてリファレンスシークエンス上に表示されます。
2つのサンプルコンディション間の発現量を比較するには、Geneiousの発現解析ツールを使用するか、各コンディションに複製がある場合は、GeneiousのDESeq2の実装を使用します。 これらのオプションは、「Annotate and Predict」メニューの「Compare Expression Levels」で利用できます。 2つのサンプルを比較するには、まず各サンプルレプリケート(同一の参照配列である必要があります)のリファレンスアセンブリでCalculate Expression Levelsを実行し、Saveをクリックして結果トラックを参照配列ドキュメントに適用します。次に、リファレンスシークエンスを選択して、「Compare Expression Levels」を実行します。
参照:Geneious FAQ: Can Geneious Prime handle RNA-Seq data? - プラグインはどこにインストールするのですか?
-
Geneious Primeには、さまざまなプラグインが用意されています。これらのプラグインは追加機能を提供するもので、Pluginメニュー(Tools→Plugins)からインストールすることができます。
インストールされたプラグインの場所は、そのプラグインの機能によって異なります。 例えば、ツリービルダー用のプラグイン(Phyml、Paup*、MrBayesなど)は「Tree」メニューのオプションとして表示され、GenBank送信用のプラグインは「Tools」メニュー(「Tools」→「Submit to GenBank」)から利用できます。 その他のプラグインは、特定のドキュメントタイプに関連付けられており、そのタイプのファイルを開いたときに機能が表示されます。 例えば、マイクロサテライトの機能は、Geneiousでマイクロサテライトのドキュメントを開いたときに表示されます。
参照:How do I install new plugins in Geneious Prime?
Geneious クラウド
参照:https://help.geneious.com/hc/en-us/articles/10895046844180
- Geneiousクラウドとは何ですか?
-
Geneious クラウドは、Geneious Prime で利用できる無料のオンラインストレージです。パーソナルサブスクリプションでは 10 GB、チームサブスクリプションでは 1シート(ユーザー)あたり 20 GB* の容量を利用することができます。
*シート数 x 20 GB の合計ストレージをチーム単位でご利用いただけます。
セキュリティに関して
Geneious の開発元である Dotmatics 社は、情報セキュリティマネジメントシステム(ISMS)の外部審査を受け、シェルマン社から国際標準化機構(ISO)27001:2013 認証を取得しています。
参照:Geneious : Ensuring Data Security in the Cloud
- Geneiousクラウド上でデータを共有できますか?
-
パーソナルサブスクリプションは、Geneiousクラウド上にあるデータの共有をサポートしていません。チームサブスクリプションは、Geneious クラウドワークスペースのデータをユーザー間で共有することができます。
- Geneiousクラウド上のデータを共有するには?
-
1. Geneious Cloud にログインすると、ソースパネルの Cloud の直下に Workspace が表示されます。
Workspace で共有するフォルダの上で右クリックし、メニューから Manage Sharing... を選択します。
ご注意くださいパーソナルサブスクリプションは、Geneiousクラウド上にあるデータの共有をサポートしていません。Geneious クラウドワークスペースのデータをユーザー間で共有することができるのは、チームサブスクリプションのみとなります。

2. Share With のドロップダウンメニューから、フォルダーを共有するユーザーを選択し、Add ボタンをクリックします。

3. アクセス可能なユーザーが表示されます。ユーザーのアクセス権限をドロップダウンメニューから選択し、Share ボタンをクリックします。
- Full Access: 閲覧、編集、共有管理
- Read and Write: 読み書き
- Read only: 読み取り専用
- Remove Access: アクセス権削除
アクセス権限を与えられたユーザーの Geneiousクラウド ワークスペースにフォルダーが表示されます。

- Geneiousクラウドはブラウザで動作しますか?
-
Geneiousクラウドはブラウザでは動作しません。Geneious Prime による解析や計算はローカルコンピューター上で実行され、解析結果を含むデータは Geneious Prime 内からクラウド上に保存する流れとなります。
- NGS データの保存に対応していますか?
-
技術的には可能ですが、Geneiousクラウドはローカルコンピューター上で解析を実行します。そのため、ローカルコンピューター上で解析を実行できるメモリとデータを保存できる要領のストレージが必要となります。
- ライセンスの有効期限が迫っています。Geneiousクラウド上のデータにアクセスできなくなりますか?
-
ライセンスが無効となっても、ライセンス有効期限終了後より1ヶ月 Geneious クラウドにアクセスすることができます。ライセンスの有効期限が切れると Restriction モードとなり利用できる機能が制限されますが、 Geneiousクラウドへのログイン、ローカルフォルダへのファイル転送、Geneious からのファイルエクスポートは可能です。
- ローカルのデータベースを Geneiousクラウドにバックアップできますか?
-
ローカルデータベースにあるデータは自動的に Geneious クラウドにバックアップされません。Geneious Backup Tool を使用して、Geneious Prime のローカルデータベースをバックアップし、このバックアップデータを Geneiousクラウド にコピーする必要があります。