系統樹の作成
このチュートリアルでは、系統樹を構築する方法や、系統樹の表示・操作方法をご紹介します。
系統樹を作成する前に、マルチプルアライメントを作成する必要があります。系統樹作成に必要な配列アライメントの作成方法ついては、シークエンスアラインメントの作成をご覧ください。
1.データの準備
このチュートリアルでは、後天性免疫不全症候群(AIDS)の原因であるヒト免疫不全ウイルス(HIV)と類人猿の免疫不全ウイルス(SIV)の配列アライメントを利用します。
データは以下よりダウンロード頂けます。
HIV sequences alignment.geneious
ダウンロードしたファイルを Geneious 上にドラッグ&ドロップするか、メニューバーの「File」→「Import」→「Files...」を選択し、ファイルをインポートします。
2. 配列アライメントの作成
今回は、Tamura-Nei モデルと Neighbour-Joining 法(近隣結合法)で系統樹の構築を実施していきます。
分子進化の解析では、各ヌクレオチドの予想される頻度とヌクレオチド間の変化の割合を利用します。Tamura-Nei モデルでは、各塩基が異なる平衡頻度を持つと仮定し、トランジションとトランスバージョンが異なる速度で発生することを前提としています。加えて、HIV の配列には宿主由来の酵素によって引き起こされる変異も蓄積しています。これらの変異には「分子時計の仮説」を適用できません。以上より、HIVの配列を分析する場合は Tamura-Nei モデルと Neighbor-Joining 法がフィットすると考えることができます。
![]() HIV Sequence Alignment を選択して、ツールバーの Tree ボタンをクリックします。
HIV Sequence Alignment を選択して、ツールバーの Tree ボタンをクリックします。

Geneious Tree Builder を選択し、Genetic Distance Model で Tamura-Nei を、 Tree Build Method で Neighbor-Joining を選択します。
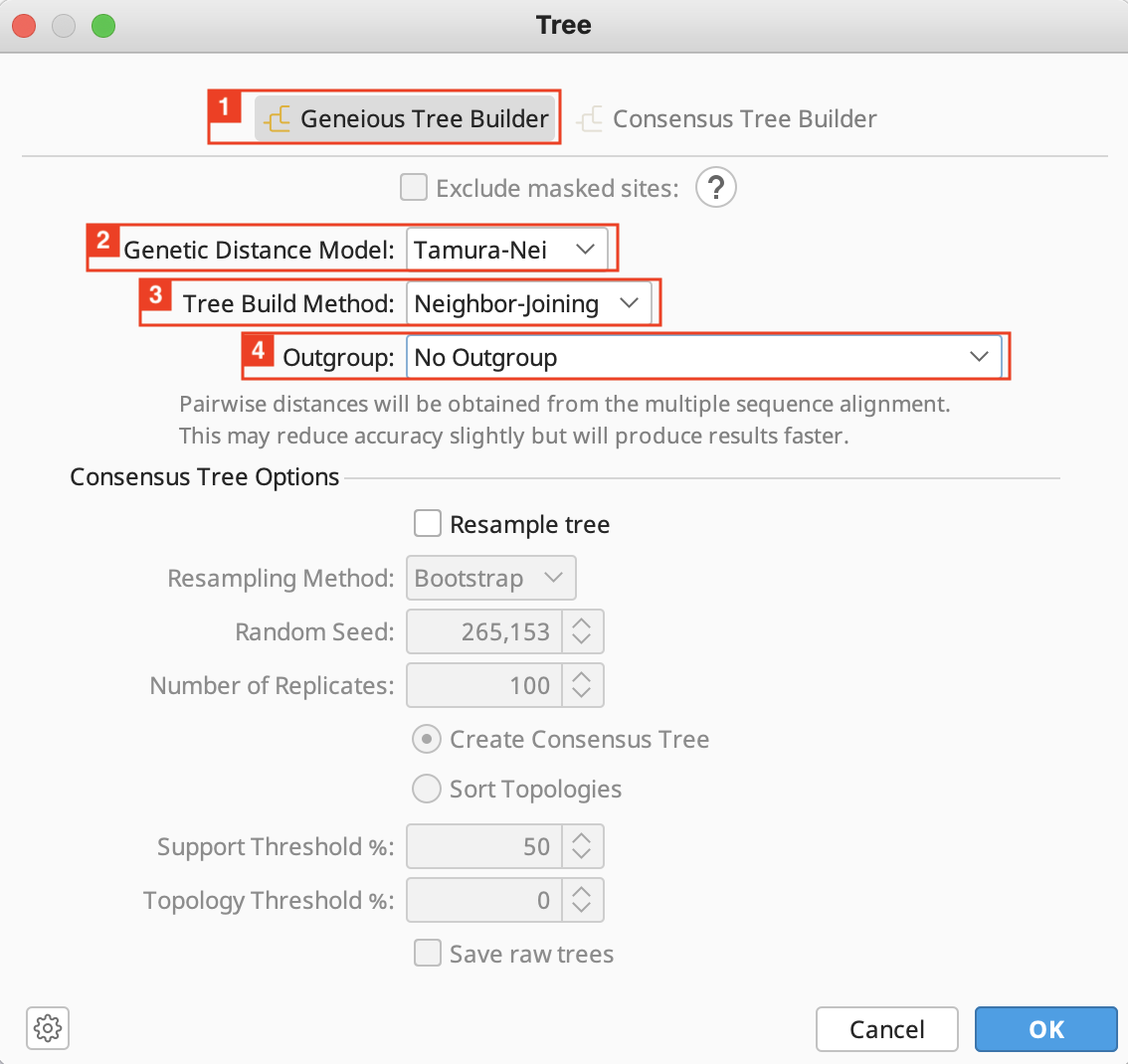
Outgroup は SIV-MON; Mona monkey; AY340701 を選択します。
ドロップダウンメニューを開いた状態で、目的のアウトグループの文字列を入力すると対象を絞り込むことができます。

ブートストラップ値を計算する場合は、 Resample tree にチェックを入れ、 Resampling method で Bootstrap を選択します。また、 Number of Replicates を 100 に、 support threshold % を 0 に設定して OKボタンをクリックします。
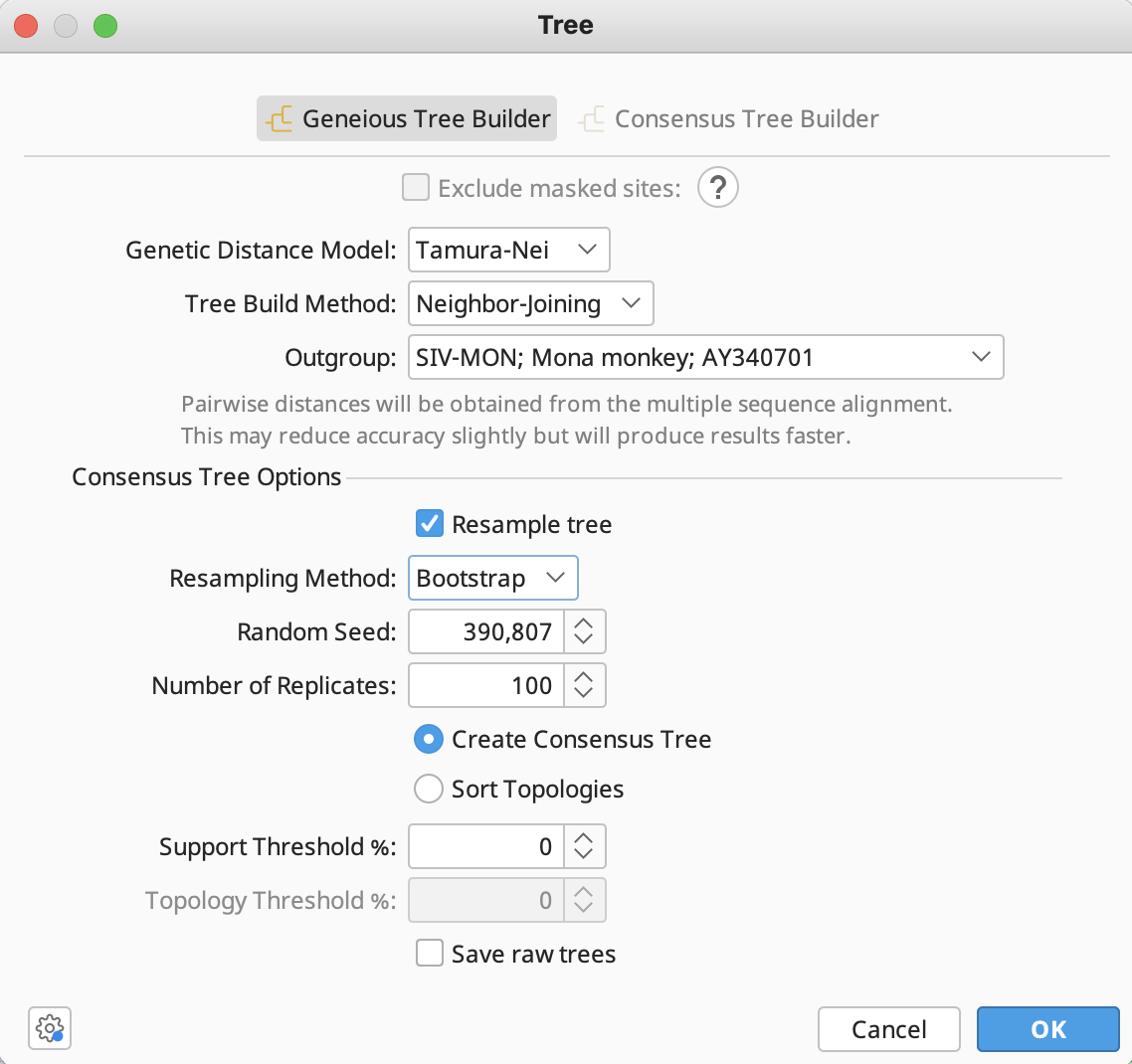
OKボタンをクリックすると系統樹作成が開始し、完了するとドキュメントビューアに結果が表示されます。
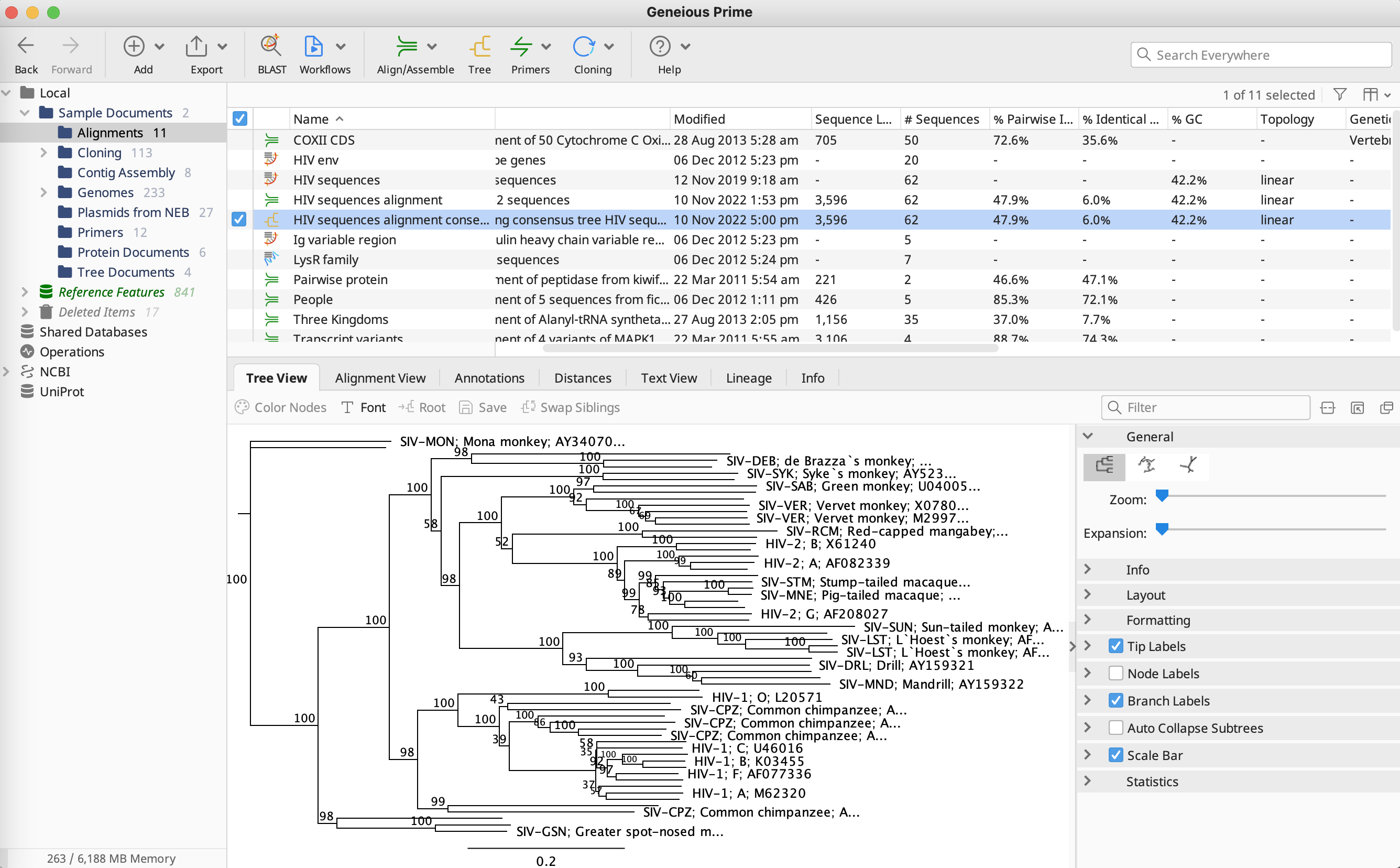
2-1. 系統樹の表示編集
系統樹の大きさによっては、系統樹上に全ての配列名を表示することができなくなります。その場合は、一部の配列名のみを表示しています。コントロールパネルにある Zoom や Expansion を使用することで、表示される配列名の数を調整することができます。

ドキュメントビューアの Alignment View タブでは、系統樹作成に使用したアライメント結果を合わせて表示することができます。

アラインメント内の配列は、系統樹のトポロジー順に並び替えされており、個々のトポロジーは系統樹とリンクしています。例えば、アラインメントで HIV-1; O; L20587" の配列を選択して Tree View タブに切り替えると、この配列が系統樹上でも選択された状態になります。


解析に使用している個々の配列に追加データが登録されている場合は、このアラインメントと系統樹にもそれが関連付けられており、追加データを系統樹上で表示することができます。
例えば、コントロールパネルの Tip Labels の Display から Organism を選択すると、トポロジーの表示名を Organism に変更できます。

複数のデータを選択して、系統樹上に表示することも可能です。Ctrl( ) または Command( ) を押しながら、 Organism と Host Organism を選択すると併記されて表示されます。

ブランチグループを回転させて系統樹の外観を変える場合は、ノードを選択しタブの上部にある ![]() をクリックします。
をクリックします。

選択したサブツリーのブランチは回転しますが、系統樹が含む情報は変わらないということに注意してください。

系統樹の表示方法には有根タイプと無根タイプがあります。今回の系統樹を無根タイプとして表示したい場合は、コントロールパネルから行います。コントロールパネルの General の下にある無根タイプ( ![]() : Circular Tree Layout |
: Circular Tree Layout | ![]() : Unrooted Tree Layout)をクリックします。なお、Neighbour-Joining 法で作成する系統樹は無根タイプになります。
: Unrooted Tree Layout)をクリックします。なお、Neighbour-Joining 法で作成する系統樹は無根タイプになります。

系統樹の枝の長さを表示するには、 Tree View タブ の右側のパネルの Branch Labels から行います。 Display から Substitutions per site を選択してください。

Display から Consensus Support (%) を選択すると、ブートストラップ値がツリーに表示されます。

系統樹の枝の長さを任意タイプのクラドグラムに変換するには、コントロールパネルの Formatting で行います。 Transform branches にチェック入れ、Transform のドロップダウンメニューから cladogram を選択します。

ブートストラップ値が少ないノードを潰して、ツリーのトポロジーに貢献しないようにしたい場合は、ツリーを構築する際のブートストラップオプションで、 support threshold % を変更する必要があります。
このチュートリアルでは support threshold % の値を 0 にしましたが、support threshold %: を 70 に設定してみると、いくつかのノードが折りたたまれていることが分かります。



