SnapGeneステップ・バイ・ステップガイド目次
SnapGeneステップ・バイ・ステップガイドはユーザーページ(エムデーエフよりSnapGeneをご購入頂いたユーザー様のみアクセス可)よりご覧頂けます。
SnapGeneライセンス管理ガイド
ネームドユーザーライセンス
メールアドレスでアクティベーションを実施
使用者を指定するライセンスです。シートは個人ユーザーに割り当てられます。シートを割り当てられたユーザー(シート数が1つの場合は登録ユーザー)のみが利用でき、第三者による使用はできません。
メールアドレスを使用してコンピューターの登録(アクティベーション)を行います。
- My Accountページ
- Dashboard タブ | インストーラーのダウンロード
- Seats タブ | ユーザー追加とアクティベーション解除
- Settings タブ | ライセンス管理者の追加
- インストールと登録
- SnapGene Viewer から製品版への切替
- アクティベーション解除(登録解除)
- ライセンス管理者パスワード/管理者情報の変更
マシンベースドライセンス
登録コードでアクティベーションを実施
使用するコンピューターを指定するライセンスです。シートはコンピューターに割り当てられます。使用するコンピューターを1台指定し共用することで、不特定多数のユーザーが使用することができます。
登録コードを使用してコンピューターの登録(アクティベーション)を行います。
- My Accountページ
- インストーラをダウンロードする
- アクティベーションを解除する
- 再アクティベートする
- 管理者権限を追加する
- インストールと登録
- SnapGene 7 以前のバージョンのインストールと登録
- SnapGene Viewer から製品版への切替
- トライアル版から製品版への切替
- コンピューターの登録解除
- ライセンス管理者パスワード/メールアドレス変更
SnapGeneスタートアップガイド目次
SnapGene 基本操作
このチュートリアルでは、SnapGeneの基本的な機能と操作方法について学びます。また、合わせてシミュレーションツールについても紹介しています。
SnapGene インポート機能
このチュートリアルでは、SnapGene から NCBI、UniProt、Ensembl のデータベースにアクセスし、アクセッション番号を使って SnapGene にシークエンスをインポートする方法を紹介しています。また、Addgene から複数のプラスミドをインポートする方法も合わせて紹介しています。
- NCBI からのインポート
- 配列のコピー&ペーストによる読み込み
- UniProt からのインポート
- Ensembl からのインポート
- フィーチャー機能:イントロンを非表示にする
- Addgene プラスミドをインポート
- プラスミド番号を使ってインポートする
- FASTAファイルのインポート
- GFF3、GTF、BED ファイルのインポート
- カスタムフィーチャータイプのインポート
- カスタムフィーチャータイプのエクスポート
配列のアライメント
このチュートリアルでは、クローニングや変異導入の検証で行うアライメント(DNAまたはアミノ酸配列を参照配列に合わせる)方法をご紹介します。
- 【準備】配列を入手する
- ファイルの読み込みと保存
- DNA配列のアライメント
- シークエンスタブで確認
- シークエンス結果の確認
- 複数の配列を比較する
- 確認済みの記録
- 参照配列と入れ替える
フィーチャー機能
このチュートリアルでは、SnapGeneでDNAフィーチャーを作成・編集する方法およびフィーチャーの自動アノテーションをカスタマイズする方法をご紹介します。
- 【準備】DNA配列を入手する
- ファイルの読み込み
- フィーチャーを追加する
- CDS領域の追加
- CDS領域の分割
- 切断部位の追加
- フィーチャーを自動アノテーション機能に追加
- フィーチャーの一括編集
- GTF, GFF3 , BED ファイルのインポート
- カスタムフィーチャータイプを作成する
- 修飾子の追加と削除
- カスタムフィーチャーの管理
- カスタムフィーチャーのエクスポート
- カスタムフィーチャータイプのインポート
プライマーの作成とPCRのシミュレーション
このチュートリアルでは、SnapGeneを使ってプライマーを作成し、PCR反応をシミュレートする方法をご紹介します。
- 【準備】DNA配列を入手する
- ファイルの読み込み
- SnapGeneでプライマー作成
- PCRをシミュレーション
翻訳機能
このチュートリアルでは、オープンリーディングフレーム(ORF)や全塩基配列の翻訳をどのように表示・編集できるかをご紹介します。
- 【準備】DNA配列を入手する
- ファイルの読み込み
- SnapGeneの翻訳機能
- ORF以外の翻訳
- CDSの翻訳
- アミノ酸配列の番号変更
ペアワイズアライメント
チュートリアルでは、DNA/RNA配列およびアミノ酸配列のペアワイズアライメントを行う方法を紹介します。
- DNA/RNA配列のペアワイズアライメント
- アミノ酸配列のペアワイズアライメント
マルチプルアライメント
チュートリアルでは、DNA/RNA配列のマルチプルライメント(多重配列整列)を行う方法を紹介します。
- 【準備】配列を入手する
- マルチプルアライメント
変異導入のシミュレーション
このチュートリアルでは、SnapGeneを使用してミュータジェネシス用のプライマーを作成し、変異導入をシミュレーションする方法をご紹介します。
- 【準備】DNA配列を入手する
- ファイルの読み込み
- 変異導入用のプライマー作成
- 変異導入のシミュレーション
サイレント変異導入
SnapGene6よりコード配列にサイレント変異を導入できるようになりました。このチュートリアルではコード配列にサイレント変異を導入し、翻訳を変えず制限部位を加える方法を紹介します。
- 酵素部位の追加
- 新しいサイトの基準を設定
- 新しいサイトの選択
- 選択部位の追加方法を選択
- シークエンスに変異を導入する
- 変異導入プライマーで変異を導入する
- 変異にフィーチャーで注釈をつける
- 変異にフィーチャーと変異導入プライマーで注釈をつける
- エクスポート
コドン頻度を表示
SnapGene は CDS の同義コドン使用頻度を算出し、結果を表として出力することが可能です。チュートリアルでは、 CDS におけるコドンの使用頻度を表示する方法を紹介しています。
コドンの最適化
このチュートリアルでは、バクテリアの CSD コドン使用頻度を、カスタマイズしたコドン使用頻度表(CUT)を用いて最適化を行う例を紹介します。使用する CUT が優先コドンとするのは、タンパク質発現宿主 Trichoderma reesei(真菌)で高い発現量が見られるものです。
- 【準備】配列を入手する
- コドン最適化
- すべての希少コドンを自動で置換する
サンガーシークエンスデータの De Novo アセンブリ
SnapGene の "コンティグをアセンブル (Assemble Contigs)" ツールは、CAP3 assembler を使ってアセンブリを実行します。CAP3 assembler は 小規模なアセンブリを行うためのプログラムです。5'端と3'端のクオリティの低い領域をトリムし、リードのアライメント実行し、コンセンサス配列をアセンブリ結果として生成します。
RNA二次構造予測図
SnapGene6.1 より、ViennaRNA で計算された最適な構造予測図が表示され、一本鎖RNA配列がどのように折り畳まれるかを視覚化できるようになりました。SnapGeneでは、3000塩基までのssRNA配列の二次構造予測図を表示することができます。
アガロースゲル電気泳動のシミュレーション
このチュートリアルでは、アガロースゲル電気泳動のシミュレーションを行い、制限酵素処理やPCRした後のゲル上のバンドを予測する方法をご紹介します。
- 【準備】DNA配列を入手する
- ファイルの読み込み
- 制限酵素処理後のゲル上のバンドを予測
- PCR後のゲル上のバンドを予測
- 泳動時間をシミュレーション
Vecotor NTIフォーマットのファイルを開く
このチュートリアルでは、Vecotor NTIフォーマットのファイルをSnapGeneで開く方法、またデータベースについてご紹介します。
クローニングチュートリアル目次
制限酵素処理による挿入クローニングをシミュレーション
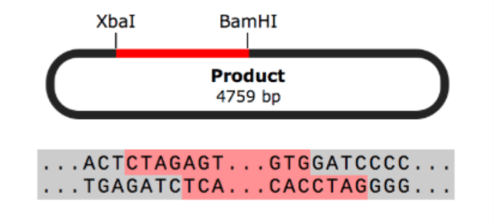
制限酵素処理による挿入クローニングは最も一般的なクローニング法です。制限酵素(1種類または2種類)を用いてインサートとベクターを調整し、DNAリガーゼを使ってライゲーション反応を行います。
Gateway®クローニングをシミュレーション
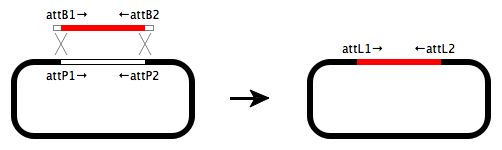
制限酵素処理を行わずに目的のプラスミドを構築するには、組換えによってDNA断片を挿入するGateway cloning®法が便利です。
- 【準備】DNA配列を入手する
- ファイルの読み込み
- Gateway Cloning® をシミュレーション
- BP クローニング反応のシミュレーション
- LR クローニング反応のシミュレーション
- BP反応とLR反応を同時にシミュレーション
Gibson Assembly®をシミュレーション
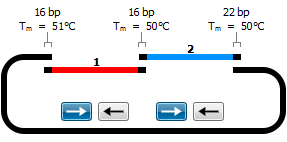
Gibson Assembly®では、DNA(フラグメント、ベクター)をPCRで増幅し、その末端にオーバーラップする領域を作ります。少々複雑ですが、SnapGeneを使えば、この方法を可視化することができます。
In-Fusion®クローニングをシミュレーション
In-Fusion®Cloningは、制限酵素を使わずに目的断片をプラスミドにクローニングする方法です。Gibson Assemblyと同様に、DNA(フラグメント、ベクター)をPCRで増幅し、その末端にオーバーラップする領域を作ります。
NEBuilder HiFi DNA® アッセンブリーをシミュレーション
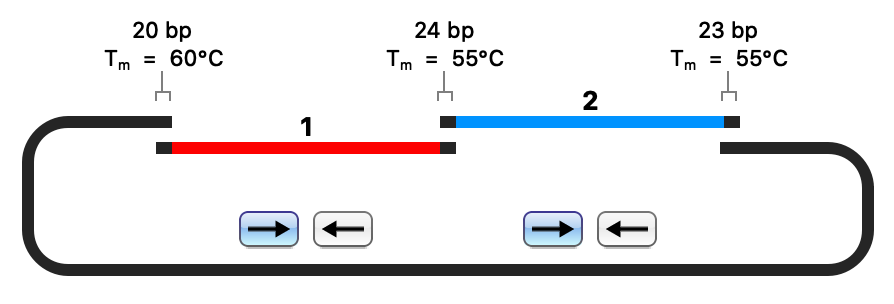
NEBuilder HiFi DNA® アセンブリは、DNA断片を1ステップでライゲーションすることができるシステムです。また、精製することなく形質転換に使用できるので、これまでのクローニングよりも作業時間を短くすることが可能です。
- 【準備】DNA配列を入手する
- NEBuilder HiFi DNA® アセンブリをシミュレーション
TA/GCクローニングをシミュレーション
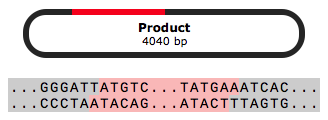
TAまたはGCクローニングは、PCR産物のクローニング方法です。この方法は、校正活性をもたないポリメラーゼによって増幅中に付加される3′端のAまたはGを利用します。PCR産物の3′端にAまたはGがあるので、3′端にTまたはCを持つ直線化ベクターにクローニングすることができます。
- 【準備】DNA配列を入手する
- TA クローニングをシミュレーション
- GCクローニングをシミュレーション
Directional TOPO®クローニングをシミュレーション
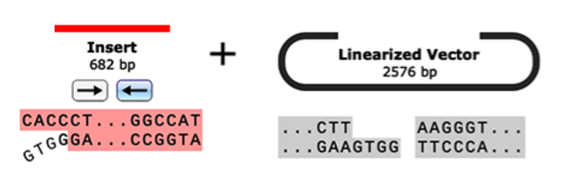
Directional TOPO®クローニングは、平滑末端のPCR産物を5´→ 3´の向きに発現ベクターに直接クローニングする方法です。
サブクローニングのステップがないため、作業時間を節約することが可能になります。
- 【準備】DNA配列を入手する
- ディレクショナルTOPO®クローニングのシミュレーション
Golden Gate アッセンブリーをシミュレーション
Golden Gate Assemblyは Type IIS 制限酵素を用いたクローニング方法で、余分な配列を含まずシームレスにクローニングを行うことが可能です。
- 【準備】DNA配列を入手する
- Golden Gate®アッセンブリーをシミュレーション
SnapGeneクローニンチュートリアルビデオを GSL Biotech社のウェブサイトよりご覧頂けます。
Resources : CRISPR
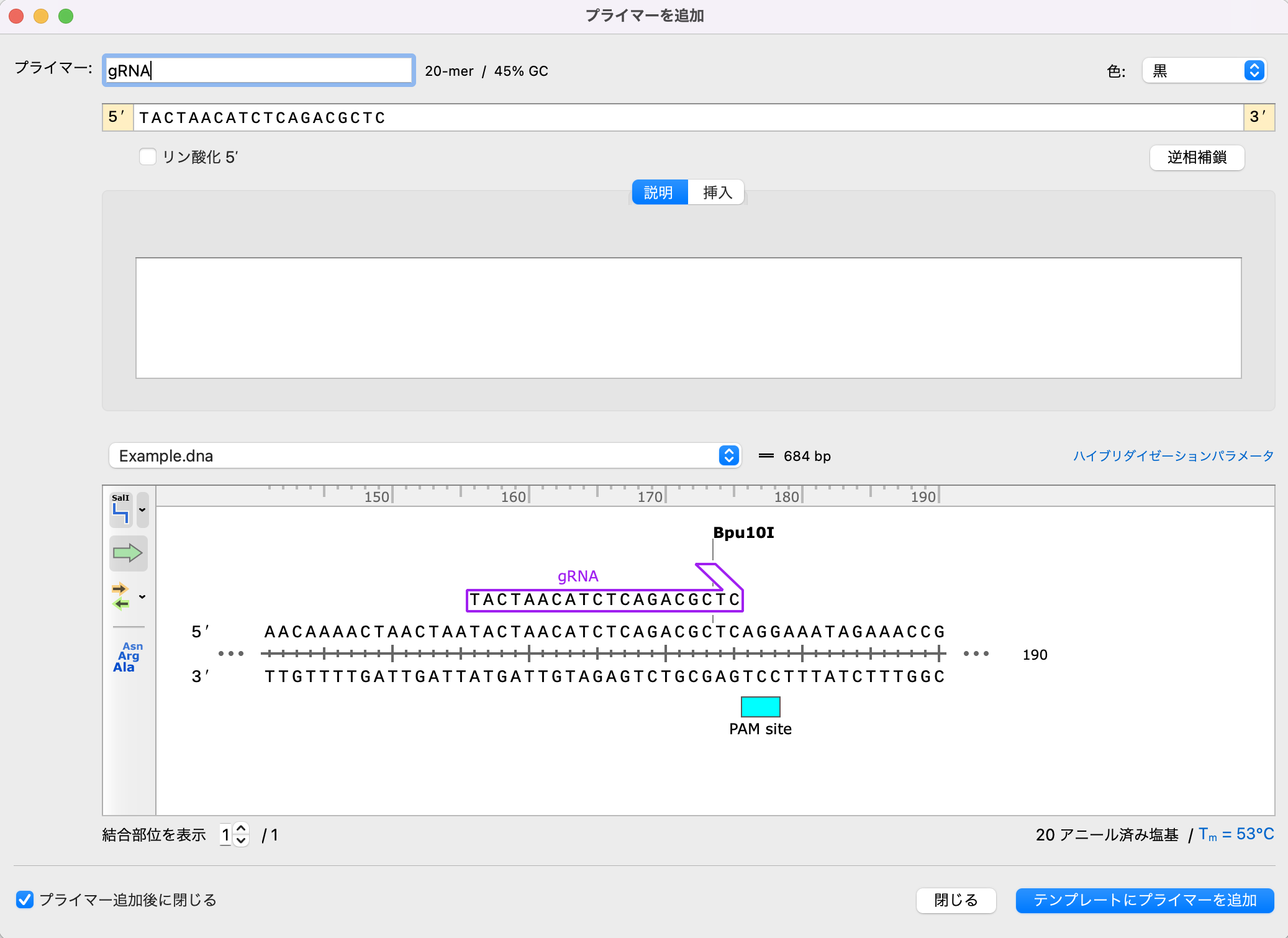
CRISPR-Cas9の実験では、「gRNA」と「修復テンプレート」の2つの異なる分子試薬を設計する必要があります。SnapGeneを使用して試薬を設計する利点は、ユーザーがシステム用に開発した注釈付きフィーチャーと対応する試薬を簡単に考慮できることです。
- gRNA
- gRNAの役割は、Cas9の切断をターゲットとすることです。実験の詳細によっては、比較的大きな配列からgRNAの候補を検索し、それらをスコア化するオンラインgRNAデザインツールを使用した方がいいかもしれません。しかし、実験が小さなゲノムターゲットに限定されている場合は、SnapGeneを使用してgRNAを設計することができます。
- 修復テンプレート
- SnapGeneでは、DNA配列を直接編集し、その編集がタンパク質翻訳に与える影響を予測することができます。さらに、ゲノム編集と既存のPCRやシーケンシング試薬との関連性を確認したり、ゲノム編集の成功を確認するための新しい試薬を特定することが可能です。
Resources : SnapGene と Addgene
addgeneのプロトコルビデオで紹介されているSnapGeneでシミュレーションする方法、シークエンス解析の結果の見方を解説しています。
オリゴオーバーラップクローニングをシミュレーション
このチュートリアルでは、addgeneのプロトコルビデオPlasmid Modification by Annealed Oligo Cloningで紹介されている「オリゴオーバーラップクローニングのシミュレーション」を解説しています。
- 【準備】DNA配列を入手する
- ファイルを読み込む
- オリゴオーバーラップクローニングをシミュレーション
SnapGeneでシークエンス解析結果を確認する
このチュートリアルでは、addgeneのプロトコルビデオで紹介されているSequence Analysis of a Plasmidに準じた内容で、シークエンス解析の結果の見方をご紹介しています。
- 【準備】DNA配列を入手する
- シークエンス結果のアライメント
- 波形データの見方
- 解析結果の自動トリミング
- 解析完了の記録